Практикум 7
Резюме: В ходе работы над этим практикумом было проведено сравнение методов предсказания трансмембранных участков белков
Сравнение предсказаний трансмембранных участков в β-листовом белке
Для выполнения этого пункта я выбрал Outer membrane phospholipase A (фосфолипаза А внешней мембранны) — это белок, расположенный на внешней мембране грамотрицательных бактерий. Идентификатор PDB - 7XZJ. Идентификатор Uniprot - PA1_ECOLI. Организм - E. Coli
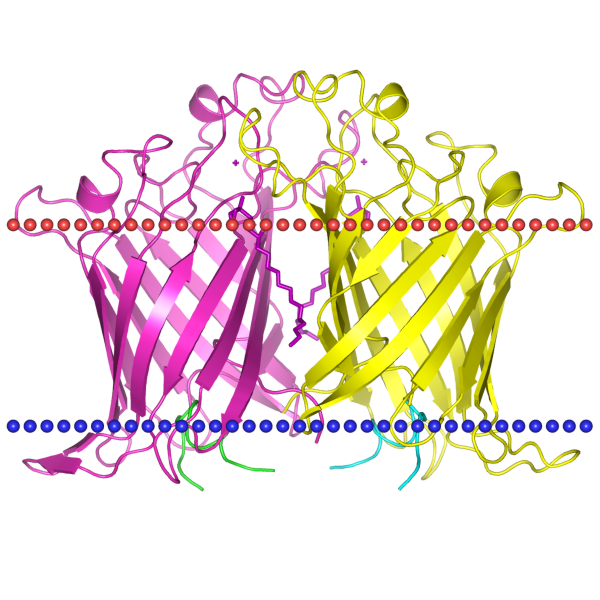
Активная форма представляет собой димер (как и на данной структуре, Рис. 1). С функциями этого белка не все понятно, пишут, что он активируется (димеризуется) при различных стрессорных воздействиях на бактериальную мембрану (тепловой шок, действии токсинов), и, соответственно, может изменять состав мембраны (за счет расщепления некоторых фосфолипидов), тем самым изменяя ее характеристики (главным образом, текучесть). Также пишут, что OMPLA (сокращенное название фермента) может участвовать в некоторых путях секреции бактериальных токсинов, модифицируя мембрану, тем самым облегчая секрецию [источник 1, источник 2].
Далее я запустил DeepTMHMM для последовательности белка (взята из UniProt) - Рис. 2

Таблица 1. Координаты трасмембранных участков OMPLA, предсказанные двумя разными методами
| Метод | Предсказанные координаты |
|---|---|
| На основе структуры | 1(38-43), 2(67-78), 3(87-96), 4(113-122), 5(135-143), 6(155-164), 7(169-179), 8(195-201), 9(206-215), 10(221-232), 11(235-243), 12(259-264) |
| На основе последовательности | 1(33-43), 2(67-78), 3(88-96), 4(113-122), 5(135-143), 6(156-164), 7(168-179), 8(195-203), 9(205-213), 10(222-229), 11(237-243), 12(259-267) |
Сравнение предсказаний трансмембранных участков в α-спиральном белке
Мне досталась структура 3LUT - Potassium channel Kv1.2. Это потенциал-зависимый калиевый канал, один из главных участников потенциала действия (отвечает за фазу реполяризации). Организм: Rattus norvegicus
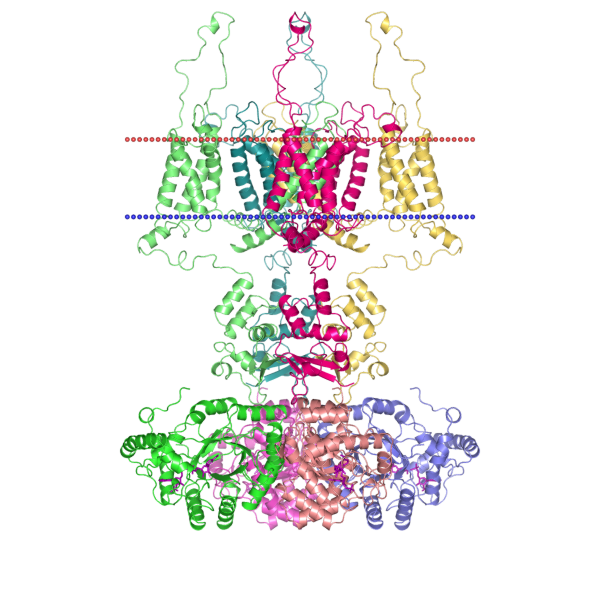
Белок представляет из себя октамер из 4х α-субъединиц - собственно формируют канал, имеют трансмембранные участки (ID UniProt - KCNA2_RAT) и 4х регуляторных β-субъединиц.
Далее я запустил DeepTMHMM для последовательности α-субъединицы (взята из UniProt) - Рис. 4

Таблица 2. Координаты трасмембранных участков α-субъединицы Potassium channel Kv1.2, предсказанные двумя разными методами
| Метод | Предсказанные координаты |
|---|---|
| На основе структуры | 1(164-182), 2(222-241), 3(254-270), 4(294-310), 5(328-346), 6(386-406) |
| На основе последовательности | 1(164-182), 2(222-242), 3(254-275), 4(290-305), 5(328-349), 6(389-410) |