Внутримолекулярные взаимодействия в структуре 2NV1
Общая характеристика структуры 2NV1
Идентификатор белка PDB: 2NV1.
Название белка: глутаминамидотранфераза пиридоксальфосфатосинтетаза (glutamine amidotransferase for pyridoxal phosphate synthesis).
Количество цепей в 2NV1: 6. Цепи соединяются между собой водородными и ионными связями. Общий вид структуры можно увидеть на рисунке 1-1 b 1-2.
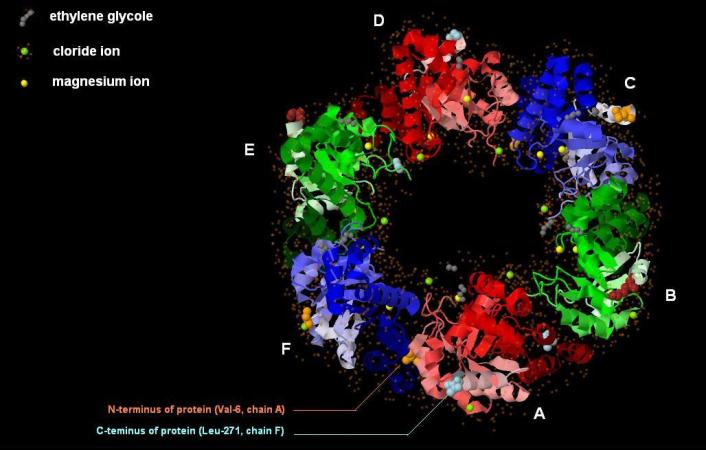
Рис. 1-1. Показаны N-конец и C-конец белка (N- и C-terminus of protein), это остаток валина (Val-6) - первый определенный в цепи А белка - и остаток лейцина (Leu-271) в цепи F. Цепи белка подписаны латинскими буквами. «Направление» цепи белка от N- до C-конца прослеживается на рисунке: происходит переход цветов от более светлых к более ярким и затем темным (для цепей A и D - оттенки красного, для цепей B и E - оттенки зеленого, для цепей C и F - оттенки синего). «Направление» бета-листов - элементов вторичной структуры - также показано стрелками (режим cartoon), их можно заметить на рисунке. Также видны отедельные атомы магния (magnesium ions) зеленого цвета, атомы хлора (cloride ions) желтого цвета, молекулы этандиола (EDO) серого цвета (2 атома углерода находятся в центре, они связаны между собой и с атомами кислорода).
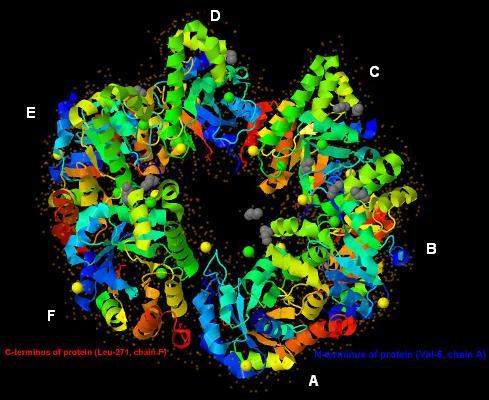
Рис. 1-2. Показана структура белка аналогично с рис. 1-1. Здесь переход окраски, показывающий направление цепей белка, выполнен с помощью радужных цветов (от N- до C-конца - переход от синего к красному). Также видны отедельные атомы магния (magnesium ions) зеленого цвета, атомы хлора (cloride ions) желтого цвета, молекулы этандиола (EDO) серого цвета (2 атома углерода находятся в центре, они связаны между собой и с атомами кислорода), аналогично рис. 1-1.
В PDB-файле помимо координат атомов белка и молекул воды приведены координаты молекул этиленгликоля (этандиола-1,2) (EDO, ethylene glycol, 1,2-ethandiol), количество этих молекул равно 11. Также присутствуют координаты атомов магния (Mg, magnesium ion), которые окружены молекулами воды, количество таких комплексов равно 12 (из них 3 в цепи А, 1 в цепи B, 3 в цепи C, 2 в цепи D, 2 в цепи E, 1 в цепи F). Интересно, что молекулы воды, окружающие атомы магния, условно принадлежат разным цепям. Приведены координаты атомов хлора (Cl, chloride ion), количество которых также равно 12 (по два иона на каждую цепь). На рисунке 2 показано расположение молекул этандиола-1,2 и атомов магния с окружающими их молекулами воды.
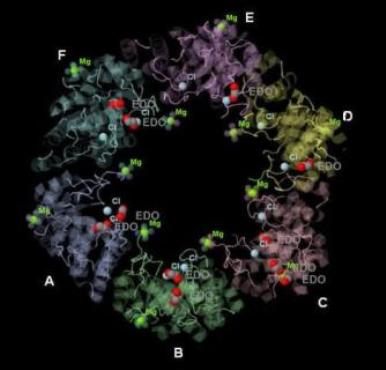
Рис. 2. Расположение молекул этандиола-1,2 (EDO), атомов хлора (Cl) и атомов магния (Mg), окруженных молекулами воды.
Обозначения и подписи на рисунке. Цепи белка окрашены в разные цвета и подписаны латинскими буквами.
Атомы магниz: Mg, зеленый цвет; атомы хлора: Cl, голубой цвет, молекулы этандиола: ED),
серым цветом выделены атомы углерода, красным – атомы кислорода.
Принадлежность молекул воды, окружающих атомы магния, к той или иной цепи можно определить по цвету.
Цепи белка близки по структуре. Каждая цепь содержит около 270 аминокислотных остатков, расположенных
в сходной последовательности.
Полипептидные последовательности цепей (и, соответственно, сами цепи), сложенные в относительно независимые друг от друга области,
представляют собой домены. [1]
Описываемый в работе белок составлен из шести копий доменов,
характерных для определенного семейтва белков - SOR/SNZ.
[2]
Домены данного белка называются PF01680. Их можно увидеть на рисунке 3-1.
[3]
Благодаря доменной структуре белков легче формируется их трехмерная структура. «Готовая» структура белка состоит из двух описываемых субъединиц и представляет собой канал (рис. 3-2).

Рис. 3-1. Домены PF01680 в белке исследуемом белке 2NV1. Домены показаны областями, выделенными красным цветом. Источник изображения: web-сайт европейского института биоинформатики.
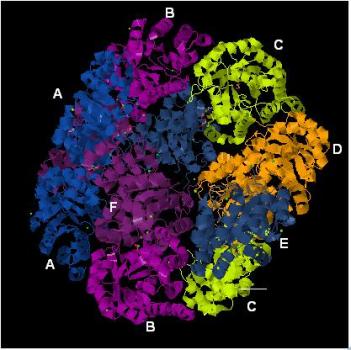
Рис. 3-2. Трехмерная структура белка. Показано соединение цепей двух субъединиц белка в реальной структуре.
Анализ структуры альфа-спиралей и бета-листов в цепи А белка 2NV1
В каждой цепи белка присутствуют и альфа-спирали и бета-листы. Далее в работе рассмотрены структуры этих элементов вторичной структуры в цепи А.
На рисунках 4 и 5 видны все альфа-спирали и бета-листы цепи А.
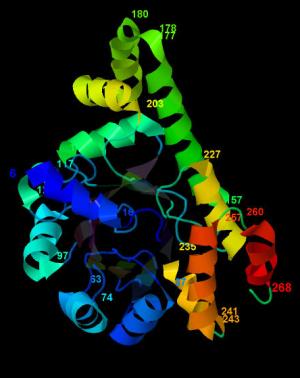
Рис. 4. Положение альфа-спиралей в цепи А. Количество спиралей равно 14. Данное изображение может быть полученo в программе JMol при использовании последовательности команд, которую можно найти при переходе по данной ссылке.
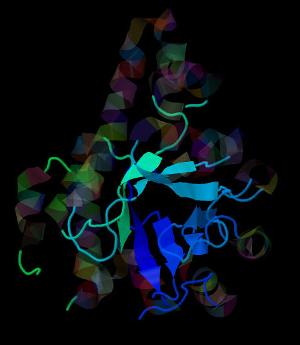
Рис. 5. Положение бета-листов в цепи А. Количество листов равно 8. Данное изображение может быть полученo в программе JMol при использовании последовательности команд, которую можно найти при переходе по данной ссылке.
Альфа-спираль цепи А
Далее приведены некоторые данные о структуре альфа-спирали, состоящей из 10 аминокислотных остатков (номера от 6 до 16) цепи А (рисунки 6-7). Эта спираль начинается с N-конца цепи А и, соответственно, всего белка.
На рисунке 6 показана структура спирали. Аминокислотные остатки выделены разными цветами в "радужной" последовательности от N-конца к C-концу.
Альфа-спирали характеризуются такими параметрами, как шаг спирали, число остатков на один виток и т. п. Под шагом спирали понимается расстояние вдоль оси спирали, соответствующее одному полному витку, то есть повороту на 360 градусов. Шаг спирали в данном случае может вариироваться от 5,84 до 6,09 Ангстрем (расстояния в Ангстремах между некоторыми атомами спирали показаны на рисунке 6). На один виток спирали приходится по 3,6 аминокислотных остатка (всего в спирали 11 аминокислот на 3 витка, 11:3=3,(6)7). Данные о прдеставленной альфа-спирали соответствуют показателям большинства альфа-спиралей, которые встречаются в различных белках (типичный виток составляет 3,6 аминокислотных остатка). [4]
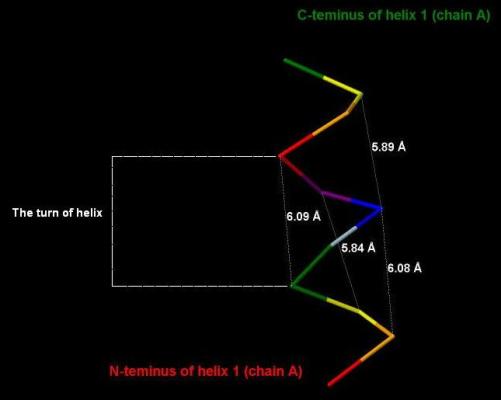
Рис. 6. Шаг спирали (альфа-спираль цепи А) - от 5,84 до 6,09 Ангстрем. Показан виток спирали (turn of helix), то есть один поворот на 360 градусов. На один виток спирали приходится примерно 3,6 аминокислотных остатка. Аминокислотные остатки выделены разными цветами в "радужной" последовательности от N-конца к C-концу.
Водородные связи для остатка n в цепи будут образовываться для атомов азота аминогруппы с атомами карбонильной группы остатков n+4 и n-4, для атомов карбонильной группой - наоборот (рис. 7).
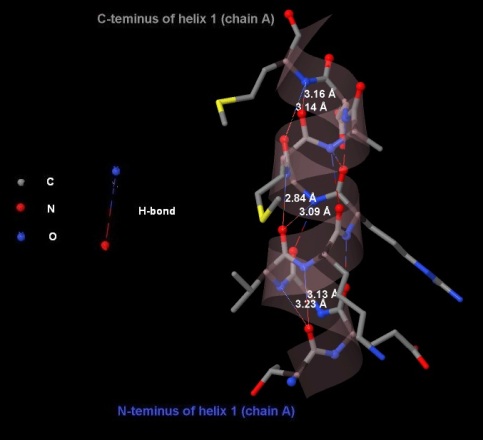
Рис. 7. Водородные связи в одной альфа-спирали цепи А (пуктирные линии). Водородные связи между карбонильной группой одного аминокислотного остатка (атомы кислорода красного цвета) и аминогруппой другого, лежащего ближе к N-концу полипептида (атомы азота синего цвета). Видно, что у большинства атомов кислорода образуется по две водородные связи, т. е. сразу с двумя атомами азота. Длина водородных связей примерно равна 3 Ангстремам.
Бета-лист цепи А
Далее приведены некоторые данные о структуре бета-листа цепи А, состоящего из 9 бета-тяжей (рисунки 8-10). Аналогичное этому листу строение имеют листы других цепей белка.
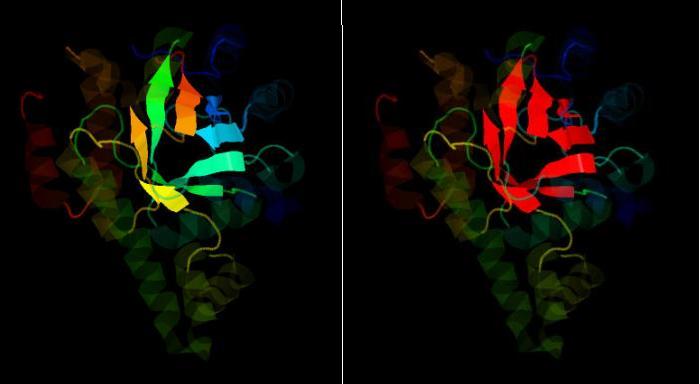
Рис. 8. Внешний вид бета-листа. Ракурс 1. На изображении слева изображен переход цветов в виде радуги от конца цепи к началу (задана прозрачность для всех элементов цепи, кроме бета-листов, для большей наглядности).
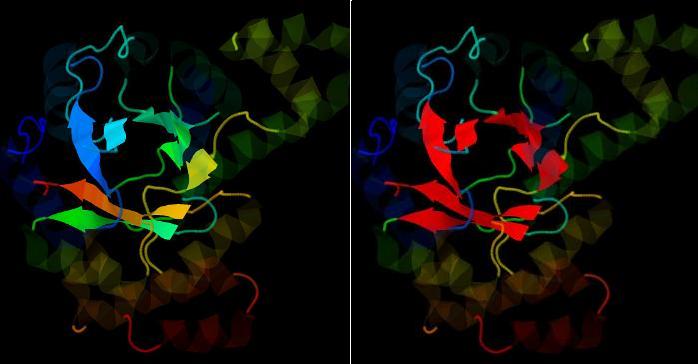
Рис. 9. Внешний вид бета-листа. Ракурс 2. На изображении слева изображен переход цветов в виде радуги от конца цепи к началу (задана прозрачность для всех элементов цепи, кроме бета-листов, для большей наглядности).
Бета-лист представляет собой винтовую выпукло-вогнутую структуру из 8 параллельных бета-тяжей (рис. 8-10). В пространстве все тяжи направлены в одну сторону (параллельность тяжей).
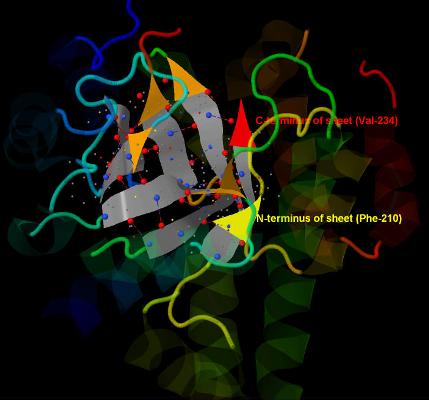
Рис. 10. Внешний вид бета-листа. Ракурс 3. Показаны водородные связи (между каждыми двумя бета-тяжами по три Н-связи). Показаны начало и конец листа (N-конец - аминокислотный остаток Phe-210, С-конец - аминокислотный остаток Val-234). Стрелками показаны направления бета-тяжей (они параллельны друг другу и образуют винтовую структуру листа).
Внутримолекулярные взаимодействия боковых групп белка 2NV1
I. Цистеиновые остатки и дисульфидные мостики.
В представленном белке в каждой цепи встречается по два аминокислотных остатка цистеина (рис. 11). Между этими остатками не образуются дисульфидные связи, т. к. расстояние между атомами серы слишком велико, оно равно 8,78 Ангстрем (рис. 12).
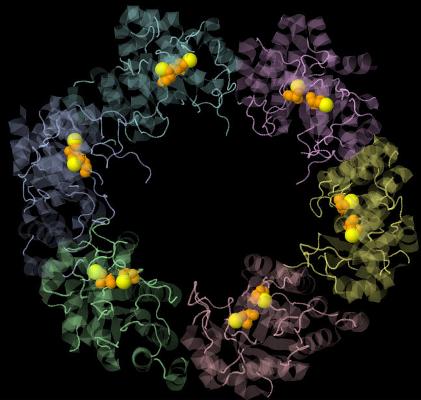
Рис. 11. Общий вид структуры белка. Цепи выделены разными цветами. Аминокислотные остатки цистеина выделены оранжевым цветом, атомы серы, входящие в состав цистеина, - крупнее - выделены желтым цветом.
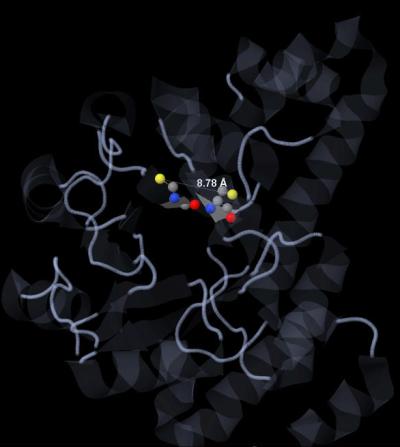
Рис. 12. Два аминокислотных остатка цистеина в цепи А белка. Атомы серы окрашены в желтый. Показано расстояние между атомами серы - 8,78 Ангстрем.
II. Солевые мостики.
Солевые мостики в белке – это ионная связь, которая образуется между положительно заряженными остатками («кислые остатки») аминокислот аргинина, лизина и гистидина и отрицательно заряженными остатками («основные» остатки) аспарагиновой и глутаминовой кислоты.
Атомы, участвующие в образовании ионной связи, находятся на расстоянии не более, чем 4 Ангстрема. На осноании этого в программе Jmol можно выделить все солевые мостики цепи А (рис. 13). В каждой цепи такое расположение солевых мостиков будет повторяться. Некоторые выделенные атомы визуально не имеют пары в мостике, это означает, что они образуют ионную связь с атомами соседней цепи белка. Отдельно рассмотрена структура двух солевых мостиков, образованных одной парой аминокислот - аргинином и глутаминовой кислотой (рис. 14).
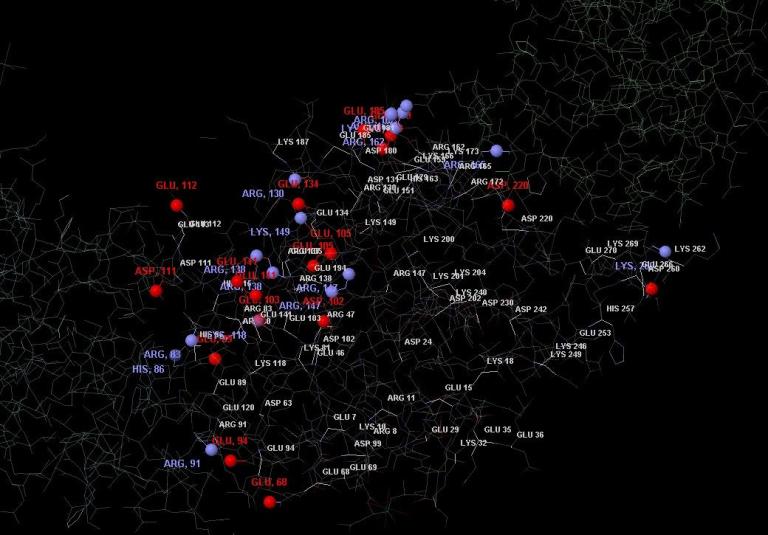
Рис. 13. Солевые мостики в цепи А молекулы белка. Видно расположение аминокислот, которые могут участвовать в образовании солевого мостика (аргинин - Arg, лизин - Lis, гистидин - His, аспарагиновая кислота - Asp, глутаминовая кислота - Glu). Выделены атомы аимнокислот, которые на самом деле образуют ионную связь (атомы азота синего цвета, атомы кислорода - красного цвета), подписаны остатки аминокислот, которым принадлежат данные атомы. Данное изображение может быть полученo в программе JMol при использовании последовательности команд, которую можно найти при переходе по данной ссылке.
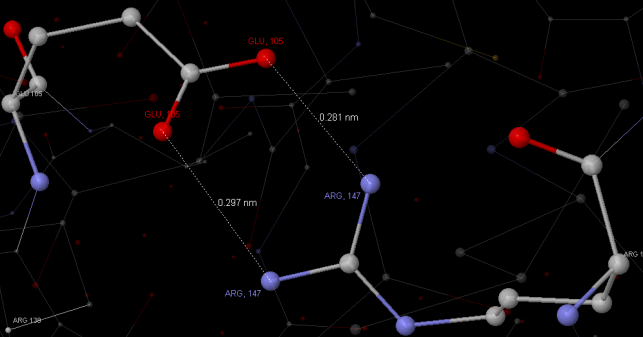
Рис. 14. Структура солевых мостиков, образованных остатками аминокислот Arg-147 и Glu-105 в цепи А. Данное изображение может быть полученo в программе JMol при использовании последовательности команд, которую можно найти при переходе по данной ссылке.
Источники информации:
1. Ресурс ЯBotanik, строение и функции белка;
2. Рерурс Pfam, семейство белков SOR SNZ;