ELVIS. Выбор Короля рок-н-ролла был запрограммирован в генах - да или нет?
Слово ELVIS встречается внутри белковых последовательностей Swiss-Prot!
Частоты встречаемости аминокислот, которые могут создать ELVIS, представлены в таблице 1.
Таблица 1. Частоты встречаемости E,L,V,I,S.
| Однобуквенный код | Полное название аминокислоты | Частота встречаемости |
| E | Глутамат | 6.75 |
| L | Лейцин | 9.66 |
| V | Валин | 6.87 |
| I | Изолейцин | 5.96 |
| S | Серин | 6.56 |
Количество аминокислотных остатков (суть число букв) в Swiss-Prot – 191670831
Вероятность нахождения “E-L-V-I-S” в одной последовательности: 0,0675*0,0966*0,0687*0,0596*0,0656 = 1.76 * 10-6
Теоретическое число “E-L-V-I-S” в последовательностях Swiss-Prot - 1.76 * 10-6 * 191670831 = 335.69 ~ примерно 335.
Сервис Prosite выдает настоящее число паттернов в базе данных. Для паттерна E-L-V-I-S это число – 173 – почти в 2 раза меньше ожидаемого. Следует отметить, что используемый расчет теоретического числа пятибуквенного паттерна является несколько грубым. Он учитывает количество совокупность символов всех последовательностей Swiss-Prot, то есть рассматривается как бы одна последовательность большой длины. На самом же деле она разбита на множество последовательностей. Так погрешность создает возможность того, что в расчет включаются и те варианты, в которых одна часть слова (паттерна) ELVIS включается в конец одной подпоследовательности, а вторая часть – в начала второй.
Видно, что и в теории, и в базе данных встречаемость "ELVIS" весьма мала. Скорее всего теория о том, что имя рок-н-рольной звезды не свяно с генетической информацией.
Поиск вероятных гомологов белка PDXS_BACSU в банке SwissProt с помощью паттернов
Поиск гомологичных последовательностей можно проводить при помощи паттернов – особого вида записи, получаемые при анализе множественных выравниваний найденных альтернативными способами гомологов. Для данной работы изначально были найдены гомологи алгоритмом BLAST, и было построено множественное выравнивание в программе JalView.
Результаты днанное работы представлены в таблице 1. Комментарии и пояснения ниже.
Таблица 1. Результаты поиска гомологов PDXS_BACSU в ProSite с помощью сильного и слабого паттерна.
| Характеристика паттерна | Паттерн | Количество последовательностей Swiss-Prot с мотивом, удовлетворяющим паттерну | Количество найденных последовательностей из исходного выравнивания (из 32-х) |
| Сильный | M-D-V-x(1)-[NT]-x(1)-E-[QE]-A-[KRQE]-[IV]-A-[EQ]-[DEAQN]-[AS]-G-A-[VCI]-[ASG]-V-M-[ASVH]-L-[DE]-x(1)-[VIL]-P-[ASY]-[DNE]-[IVL]-R-x(2)-G-G-[VI]-[AS]-R-[MT] | 108 | 16 |
| Слабый | M-D-V-[VT]-x(2)-[ED]-x(1)-A-x(2)-A-[EQ]-x(1)-[AS]-G-A-x(2)-V-M-x(1)-L-[ED]-x(2)-P-x(3)-R-x(2)-G-G-[VI]-x(1)-R-[MT] | 159 | 16 |
Примечания к таблице.
Паттерны созданы вручную, необходимо пояснить их выбор. Для из создания паттернов была выбрана область выравнивания, последовательность в которой ответственна за формирования части вторичной структуры белка, которая играет роль в сборки субъединиц в единое целое, т. е. важный сравнительно консервативный кусок, который должен быть у гомологов.
Сильный паттерн. Было найдено 108 последовательностей. Получено выравнивание частей последовательностей, соответствующих паттерну (перейти по ссылке, для находок по сильному паттерну ), созданному в ProSite с помощью программы Clustal alignment. Видно, что выравнивание «успешное», в столбцах по толщине явно большой процент идентичности, это показывает, что паттерн содержит много условий.
Также сильный паттерн должен находить гомологичные последовательности. О гомологии правильно было бы судить по полноразмерному множественному выравниванию. Такое выравнивание было проведено в программе JalView после обработки файла с последовательностями в программе Muscle.
Картинка с выравниванием представлена на рисунке 1 (минус картинки в том, что не выбран порог окрашивания столбцов при определенном проценте совпадения, окраска соответствует отношению аминокислоты к группе по свойствам радикала). Можно просто глядя на выравнивание заключить, что все найденные последовательности являются гомологами. Выровненные последовательности представлены в файле alig108.fasta.
Итак, исходя из выше сказанного, можно заключить, что сильный паттерн создан удачно.
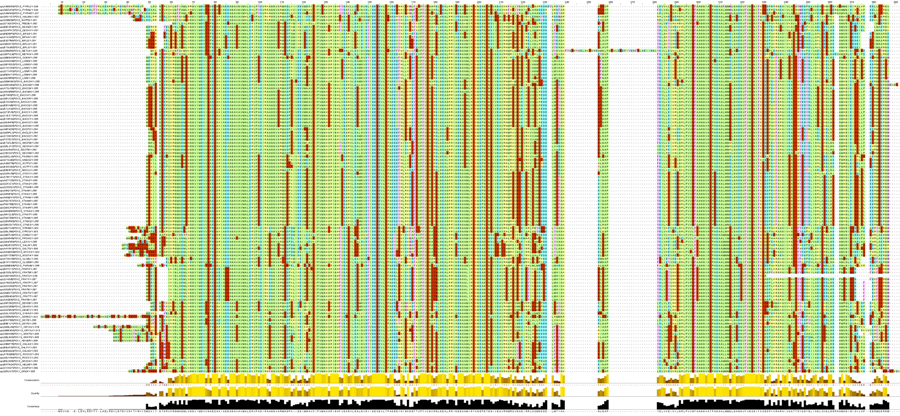
Рис. 1. Множественно выравнивание последовательностей, найденных с использованием сильного паттерна. Окраска соответствует отношению аминокислоты к группе по свойствам радикала. Порог окрашивания при определенном проценте совпадания (cut-off) - 0. Для рассмотрения полноразмерной картинки следует перейти по ссылке кликнув по рисунку.
Слабый паттерн. На слабый и более короткий паттерн было наложено значительно меньше условий. Тем не менее, было найдено не значительно больше последовательностей – всего 159. Было проведено множественное выравнивание (как для сильного паттерна). Оно представлено на рисунке 2. Наблюдается значительно меньшая гомология в совокупности последовательностей, поэтому можно говорить, что паттерн слабый. Хотя следует отметить, что можно еще сильнее облегчать условия для паттерна при необходимости. Например, если нужно строго все гомологи. При этом возможны находки явно не гомологичных белков. Выровненные последовательности представлены в файлеalig159.fasta
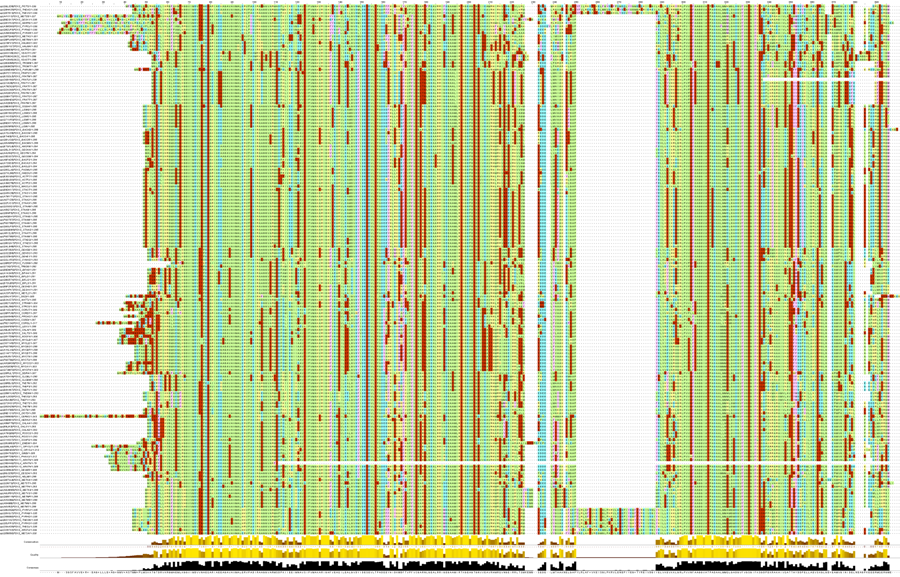
Рис. 2. Множественно выравнивание последовательностей, найденных с использованием слабого паттерна. Окраска соответствует отношению аминокислоты к группе по свойствам радикала. Порог окрашивания при определенном проценте совпадания (cut-off) - 0. Для рассмотрения полноразмерной картинки следует перейти по ссылке кликнув по рисунку.
При поиске ProSite по базе данных Swiss-Prot находится всего 16 последовательностей из исходного выравнивания. Это связано с тем, что остальные находятся в банке TrEMBL (поиск хитов с помощью BLAST проводился по базе RefSeq), при поиске по последнему находятся все исходные гомологи. Окраска соответствует отношению аминокислоты к группе по свойствам радикала. Порог окрашивания при определенном проценте совпадания (cut-off) - 0. Для рассмотрения полноразмерной картинки следует перейти по ссылке кликнув по рисунку.
На рисунках 3 и 4 показано распределение найденных последовательностей (хитов) по таксонам.
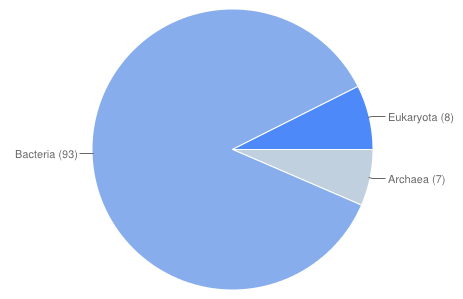
Рис. 3. Распределение хитов по таксонам для поиска по сильному паттерну.
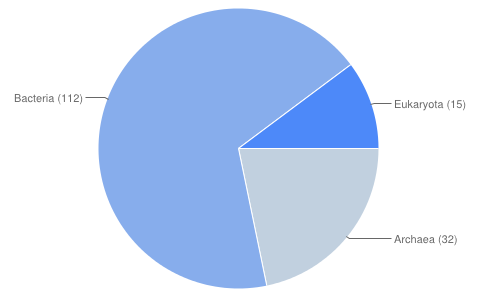
Рис. 4. Распределение хитов по таксонам для поиска по сильному паттерну.
Поиск всех мотивов ProSite в последовательности белка PDXS_BACSU
Для белков известны часто встречающиеся специфические мотивы - консервативные участки последовательностей, как правило, отвечающие за выполнение белком определенной функции или появляения типичной вторичной структуры. Система ProSite позволяет осуществлять поиск всех известных мотивов, принадлежащих данному белку. Такой поиск был проведен для последовательности PDXS_BACSU. Резульаты представлены в таблице 2.
Таблица 2. Мотивы в PDXS_BACSU.
| Идентификатор документа Prosite (AC) | Название мотива | Краткое описание мотива | Тип подписи | Паттерн | Специфичность подписи | Количество мотивов, найденных в белке |
| PS51129 | PdxS/SNZ family profile | Типичный домен для SNZ-семейства белков | MATRIX | - | Подпись неспецифична | 1 |
| PS01235 | PdxS/SNZ family signature | Типичный домен для SNZ-семейства белков | PATTERN | [LV]-P-[VI]-[VTPI]-[NQLHT]-[FL]-[ATVS]-[AS]-G-G-[LIV]-[AT]-T-P-[AQS]-D-[AGVS]-[AS]-[LM] | Подпись неспецифична | 1 |