PSI-BLAST
Данная работа посвящена программе PSI-BLAST (Position-Specific Iterated BLAST), которая позволяет производить поиск гомологов из специфичных групп белков. Это происходит при использовании профилей PSSM (Position-specific scoring matrix), построенных на основе выравниваний уже найденных гомологов. Профиль – это матрица (таблица), показывающая вес каждой аминокислоты в данной позиции (вес наибольший при наибольшей частоте встречаемости данного остатка в определенной позиции, напротив – наименьший).
Итерация – организация обработки данных, при которой действия повторяются многократно. После каждого поиска гомологов профили корректируются относительно нового выравнивания (по новым профилям идет следующий профиль), т.е. алгоритм PSI-BLAST итеративен.
После каждой итерации пользователь PSI-BLAST должен по своему усмотрению выбирать гомологов из найденных хороших (с низким e-value) и плохих (с высоким e-value). Итерации должны продолжаться до того момента, когда разница между e-value наиболее и наименее удачных находок будет сильно заметна, т. е. в список «хороших» гомологов не будут попадать новые записи.
Итоговой целью поиска является создание семейства специфичных белков и сравнение результата с данными Pfam. Поэтому в каждую следующую интерацию не включаем находки с сильно высоким e-value и последовательности белков таксономически сильно далеких организмов.
В качестве исходной последовательности был взят гомолог белка человека (Homo Sapiens) BMP and activin membrane-bound inhibitor. BMP (bone morphogenetic protein) – один из факторов роста.
Информация о белке: ID -Q9NXZ6; AC - CAB89152.1; описание NCBI.
Запуск PSI-BLAST проводили с использованием банка данных nr. Некоторая информация о проделанных итерациях приведена в таблице 1.
Таблица 1. Информация об итерациях.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 89 | XP_002408568.1 | 0.004 | XP_003427942.1 | 0.021 |
| 2 | 95 | XP_003427942.1 | 0.001 | CBY24539.1 | 0.006 |
| 3 | 100 | CBY24539.1 | 0.003 | AAC34382.1 | 0.017 |
| 4 | 99 | XP_003402273.1 | 0.005 | CBY24539.1 | 0.007 |
| 5 | 97 | XP_003251656.1 | 0.004 | XP_003701755.1 | 0.007 |
| 6 | 95 | XP_002408568.1 | 0.001 | XP_003251656.1 | 0.022 |
| 7 | 94 | XP_002408568.1 | 2.00E-04 | XP_003427942.1 | 0.016 |
| 8 | 94 | XP_002408568.1 | 2.00E-04 | XP_003427942.1 | 0.016 |
Было получено выравнивание последовательностей, найденных с помощью работы PSI-BLAST. Оно представлено на рисунке 1.

Рис. 1. Выравнивание.
Сравнение выравнивания семейства домена из Pfam с полученным PSI-BLAST
Все последовательности из выравнивания содержат один домен BAMBI (BMP and activin membrane-bound inhibitor (BAMBI) N-terminal domain). В Pfam содержится 68 последовательностей с данным доменом. Очевидно, многих последовательностей банка nr просто нет в Pfam. Судя по таксономическому распределению этих последовательностей можно сказать, что результаты работы PSI-BLAST вполне удачные. Большая часть последовательностей из полученного выравнивания содержится в семействе Pfam. Рисунок с распределением представлен ниже.
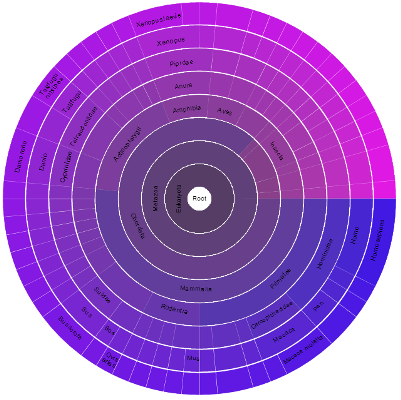
Рис. 2. Таксномическое распределение последовательностей Pfam с доменом BAMBI.