EMBOSS
Программа getorf пакета EMBOSS
Данный раздел посвящен использованию программы getorf для работы с записями последовательностей различных банков данных.
Была получена последовательность D89965 из банка EMBL:
С помощью getorf был получен набор трансляций всех открытых рамок нуклеотидной последовательности из данной записи, определенных при использовании стандартного кода и одновременно удовлетворяющих следующим условиям:
- длина более 30 аминокислотных остатков (т.е. не менее 90 нуклеотидов);
- начинаются со старт-кодона (или начала последовательности) и заканчиваются стоп-кодоном (или концом послеледовательности):
Было обнаружено 5 последовательностей. Одна из них (D89965_3) соответствует исходной CDS (прямой рамке) из записи. Интересно, что координаты начала совпадают - 163, коррдинаты конца отличаются на 3 (из-за стоп-кодона, который присутствует в CDS).
Была также получена запись данной последовательности из банка Swiss-Prot: hslv_ecoli.fasta, на которую ссылается EMBL. Эта последовательность соотвествует другой рамке - D89965_5 (обратной рамке). Это было замечено при использовании команды:
Результат выполнения представлен в данном файле.
Выводы
Заметим, что в EMBL запись относится к крысе Rattus norvegicus, а в Swiss-Prot к Ecoli. Дело в том, что EMBL - архивный банк, в котором изменения вносит только автор. Вероятно, авторы данной записи охотились за рецептором крыс, имея данные, например, о его массе и заряде. Выделив мРНК и отсеквенировав кДНК гомогенизата желкудка крыс, исследователи могли обнаружить открытую рамку, подходящую под интересующие свойства (можно судить опять же по весу предполгаемого транслята, по заряду аминокилсот). После такого обнаружения белка возможно создание записи в EMBL.
Позднее ученые могли заметить, что есть другой белок по той же рамке, и в аннотированный банк Swiss-Prot такую запись загружать не стали.
Оказалось, что реально транслируемый белок принадлежит Ecoli, которая распространена повсеместно.
Вероятность ошибкиу первых авторов была довольно мала, потому что длины между стоп-кодонами - примерно 21 кодон (т.к. всего 64 кодона), редко бывает больше. Тем не менее, такая запись - показательный пример того, что имеются открытые рамки считывания, на самом деле не транслирующиеся, а также того, что плохо секвенировать мРНК вместе с грязью.
В Swiss-Prot для данной записи есть ссылка на запись EMBL с примечанием о неправильной аннотации.
Файлы-списки
Этот раздел посвящен получению последовательностей алкогольдегидрогенез интересующих организмов (organisms.txt). Ниже перечислены использованные команды:
Последовательности алкогольдегидрогеназ для заданнах организмов можно увидеть в файле adh_15.fasta. Попутно полученные файлы: adh.fasta, adh_15.infoseq.
EnsEMBL
В данном разделе представлена некоторая информация о гене человека, кодирующего белок P2RX1_HUMAN (AC в Swiss-Prot: P51575), полученная с помощью сервиса EnsEMBL, используемого для визуализации известной информации о геномах человека и животных.
Название гена P2RX1, АС: A47363.1.
Было проведено исследование последовательности через BLAST/BLAT на сервисе EnsEMBL. Некоторая информация раздела выдачи "Region in detail" (для первой находки среди выравниваний) перечислена ниже.
Ген закодирован на 20 хромосоме, координаты гена: 51,473,295-51,477,299. На рисунке 1 красной рамочкой обведен участок хромосомы, в котором расположен ген.
Есть разные варианты проверки генов: автоматическая - компьютерная, ручная - человеком. Наиболее надежными транскриптами считаются проверенные обоими путями. Такие гены выделены желтым цветом, на рис.1. такой транскрипт пристутсвует.

Рис. 1. Расположение гена P2RX1 в хромосоме 20.
На рисунке 2 представлено более детально окружение гена. В строке "Contigs" указаны рядом лежащие гены с обозначенными АС. В строке Genes также показаны гены, кодирующие только РНК (выделены фиолетовым).
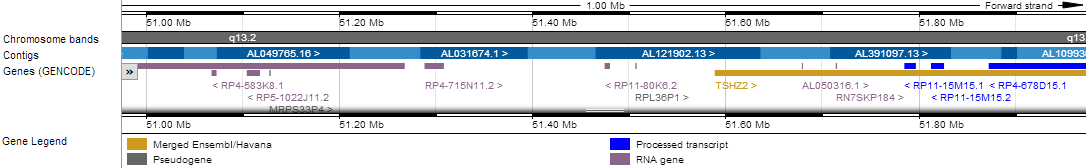
Рис. 2. Гена P2RX1 в хромосоме, его "окружение". Пояснения в тексте.
Рисунок 2 представляет собой картинку из выдачи по умолчанию. Можно изменять масштаб рисунка и различные параметры, например, искать только экзоны гена, транскрибирующиеся части его окружения, открытые рамки считывания, делать выравнивания с соответствующими участками хромосом различных животных.
Можно также добавлять свои данные. Вероятно, это помогает исследователям визуализировать сравнения новых данных с уже полученными.
Помимо выдачи "Region in detail" существует еще множество разделов в EnsEMBL.
Например, в разделе "Chromosome summary" можно увидеть некоторую информацию о генах в разных участках хромосомы в целом. Рисунок для хромосомы 20 приведен ниже. Можно заметить, что в участке q13.2, в котором закодирован интересующий нас ген, например, довольно мало кодирующих белки генов.
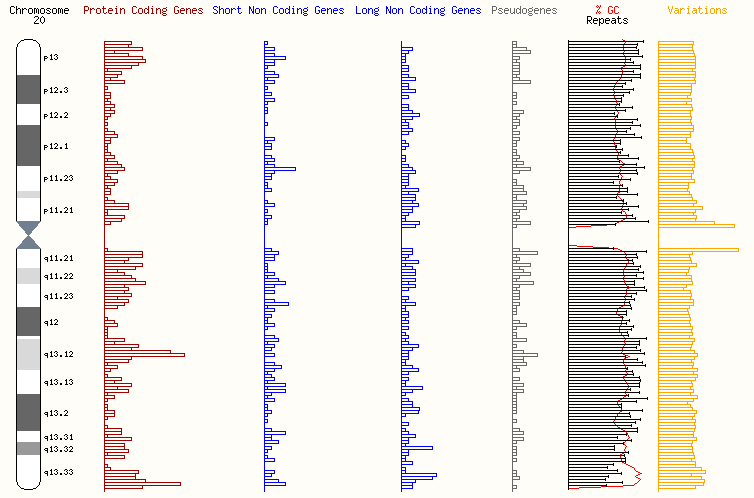
Рис. 3. Гены на всей хромосоме (Cromosome summary).
Перечисленное здесь не исчерпывает функционала EnsEMBL. Отметим, что часто исследователям необходимо знать информацию об экспрессии генов в различных тканях при разных условиях. Автор данной странички не раз обращался к сервису "Gene expression in Ensembl".