Эволюционные домены
Каспазный домен
Данная работа посвящена изучению каспазного домена (caspase domain) семейства peptidase_C14 (PF00656).
Для реконструкции эволюции доменной архитектуры белков был выбран домен каспазных белков. Каспазы - это семейство цистеиновых протеаз, расщепляющих белки после аспартата. Белки этого семейства играют важную роль в процессах клеточной смерти и воспалительных процессах. Функциональную особенность каспаз обеспечивают пептидазные домены.
| Pfam AC семейства | Pfam ID семейства | Описание | Доменные архитектуры |
| PF00656 | Peptidase_C14 | Каспазный домен | 280 архитектур, для которых доступны 4033 последовательности, принадлежащие 992 видам |
Загруженное с помощью JalView выравнивание из Pfam (File > Fetch Sequences по идентификатору PF00656) было раскрашено по консервативности (BLOSUM62 и By conservation с порогом консервативности 15%). К последовательности CASP1_HUMAN (домен идет от 163 до 400 аминокислотного остатка) была добавлена 3D структура (PDB ID 1BMQ). Выравнивание было сохранено как проект JalView в файле casp_domains.jar.
Архитектуры с каспазным доменом
Основным результатом данной работы является информация, предствленная в файле architectures.xlsx.
С использованием скрипта swisspfam_to_xls.py и файла, содержащего информацию об архитектуре всех последовательностей UniProt, была получена таблица с информацией об архитектуре последовательностей, содержащих каспазный домен. Она была импортирована в Excel ("Основная таблица" в файле).
На основе полученных данных в Excel была составлена сводная таблица ("Сводная таблица" в файле). Она содержит список разных доменных архитектур с этим доменом и указанием числа последовательностей, а также таксономию ("Таксономия" в файле). Последняя получена на основе данных Uniprot (работа с АС в сервисе Retrieve, flat text-файл), которые были преобразованы в таблицу Excel с помощью скрипта.
Полученная таксономия была перенесена в основную таблицу ("Основная таблица" в файле) с помощью ВПР, одной из любимых экселевских функций автора этой работы.
список последовательностей с указанием доменной архитектурыДля дальнейшей работы были выбраны две двух-доменные архитектуры:
- с доменами Peptidase_C14 и CARD (PF00619)
- с доменами Peptidase_C14 и DED (PF01335)
CARD - caspase activation and recruitment domains - домен-посредник, необхоимый для формирования комплексов множества белковых субъединиц. Он найден в множестве белков, ассоциированных с апоптозом. Каспазы в самом деле после активации образуют тетрамерные комлпексы для выполнения множества функций. По крайней мере, так действуют основные исполнительные каспазы клеток человека (каспазы 3,7). Но как мы видим, во многих белках, содержащих каспазный домен, не представлен CARD.
DED - death-effector domain - домен, который часто выполняет инактивирующую функцию, направленные на другие белки, у белков, необходимых для по-разному осуществяемой регуляцией клеточных сигнальных каскадов, как правило связанных с апоптозом.
ЧАСТЬ 1
ЧАСТЬ 1 посвящена исследованию одновременно двух архитектур и филогении последовательностей, имеющих ту или иную архитектуру, но в совокупности. Вторая часть работы будет ближе к получению необходимого результа, т.к. будет содержать информацию о филогении одного каспазного домена в различных архитектурах.
Архитектуры в последовательностях организмов разных таксонов - 1
Каспазный домен peptidase_C14 - очень широко представленная структура среди различных клеточных организмов всех царств - Archaea , Bacteria и Eukaryota. Более того, были обнаружены и последовательности с каспазным доменом, содержащиеся в геноме вирусов (Viruses).
Тем не менее, целью для изучения в данной работе был выбран таксон Metazoa, принадлежащий царству Eukaryota. В нем широко представленны последовательности белков различных орагнизмов, содержащие выбранные архитектуры.
В качестве подтаксонов были выбраны Chordata (хордовые) и Ecdysozoa (линяющие, по сути большая часть беспозвоночных, условный раздел, клада на основне молекулярно-генетических данных). Первая архитектура (с CARD) одинаково характерна для обоих подтаксонов. Вторая архитектура (с DED) не встречается нигде, кроме как у позвоночных животных, зато она очень растпространена. Поэтому подтаксоны этой группы тоже будут различены и указаны в работе в дальнейшем.
Была создана репрезентативная выборка для последовательностей с разными архитектурами. Следили за тем, чтобы длина доменов в последовательностях была примерно одинаковой, чтобы уменьшить вероятность получить плохое и неправильное выравнивание. Эта длина составила 200-250 а.о. В обновленном excel-файле со сводной таблицей (ссылка на скачивание) можно увидеть список последовательностей, взятых для репрезентативной выборки, с указанной таксономией, лист "Выборка". C ней удобно смотреть на выравнивание. На остальных листах файла можно увидеть некоторые пояснения к выбору этих последовательностей, узнать длины домена в них и др. Одна последовательность для каждой архитектуры (белки человека) имеет 3D структуру.
На основе созданной выборки (последовательности fasta) получено выравнивание с помощью muscle (ClustalW). Проект JalView представлен в формате jar: represent_ali.jar.
В выравнивании были удалены пустые столбцы. Были добавлены аннотации вторичной структуры на основе 3D-структур CASP1_HUMAN (1BMQ, для первой архитектуры) и CFLAR_HUMAN (3H11, для второй архитектуры), эти последовательности находятся в конце каждого блока (блоки разделены на группы по архтиктурам). Для каждой группы была выбрана раскраска Clustal, порог By Conservation в первом случае 10%, во втором - 15%. На месте спиралей и тяжей, составляющих ядро домена гэпы крайне маловероятны. В одном случае в выравнивании встретился гэп, он был удален. В N-концевой части выравнивание плохое (его нет), но он не удален.
Увидеть изображение выравнивания можно перейдя по ссылке.
Все последовательности выровнялись неплохо. Судя по вторичной структуре, выравнивание правильно. В целом, много консервативных позиций. Виден каспазный домен, он длинный, около 200 а.о. DED только в N-концевой части. Интересно посмотреть на филогению, потому что выравнивание не обладает значимой информативностью. Разные подтаксоны имеют похожие последовательности. На этом этапе инверсий, повторов и других явлений не выявлено.
Филогения последовательностей с каспазным доменом - 1
Данный раздел посвящен описанию филогенетическое дерева, полученного на основе выравнивания последовательностей представителей домена.
Последовательности выравнивания были переименованы, далее работа проводилась с новым файлом. Легенда:
1 - первая архитектура (CARD, peptidase_c14)
2 - вторая архитектура (DED, peptidase_c14)
C - Chordata
- CP - Cephalochordata
- CR - Craniata
- TU - Tunicata
E - Ecdysosoa
- A - Arthropoda
- N - Nematoda
Для построения деревьев использовался метод максимального правдоподобия (Maximum Likelihood) с бутстреп-анализом (количество реплик - 100). Метод не предполагает молекулярные часы и выдает длины ветвей. Построенные деревья приведены на рисунках 1 и 2. Скобочные формулы этих деревьях можно найти, соответственно в файлах 1 и 2.
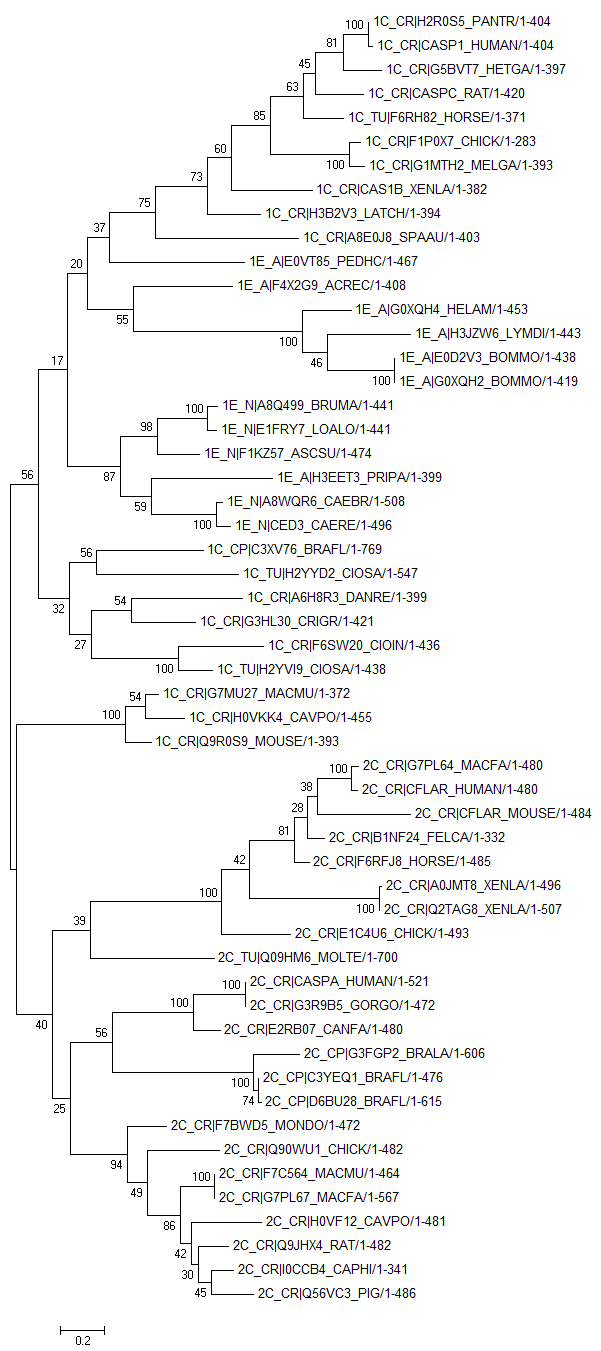
Рис. 1. Original tree.
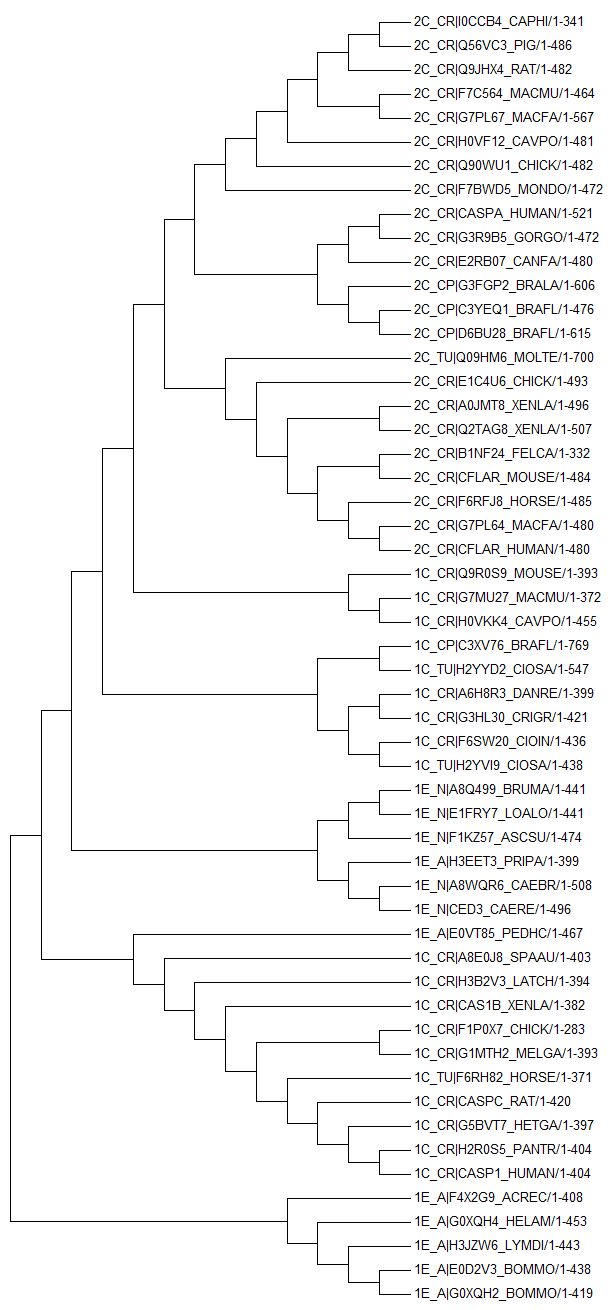
Рис. 2. Bootstrap consensus tree.
Можно видеть, что двухдоменные архитектуры 1 и 2 образуют две большие клады. Вероятнее всего, что все три домена изначально имелись у общего предка. Также можно предположить, что произошла делеция каждого домена по-отдельности при разделении на две вышеупомянутые клады.
Обратим большее внимание на дерево, полученное с бутстреп-анализом. Схема наглядна из-за названий последовательностей. Судя по всему, дерево на основе полученных последовательностей не получилось правильным. Не получается придумать вариант укоренения, чтобы результат был похож на правду. Дерево противоречиво несколько раз показывает возможность происхождения последовательностей хордовых и беспозвоночных от разных общих предков. На правду это мало похоже. Следует предположить, что отобранные последовательности довольно сходны, и по ним трудно оценить ход эволюции доменов.
Судя по дереву, домен DED появился в эволюции позднее. Есть общий предок у организмов, содержащих 1 и 2 архитектуры. С другой стороны, необходимо посмотреть на первое дерево, где указана достоверность каждой ветви (процент деревьев, в которых встретилась данная ветвь при построении множетсва деревьев бутстрепом). Выводу оно не противоречит, однако, следует отметить, что многие ветви в основнии имеют малую достоверность. Тем не менее, большую достоверность имеют ветви, у которых происходит разделение на хордовых и беспозвоночных и, конечно, на организмы, содержащие 1 и 2 архитектуры.
Видимо, интереснее было бы взять одну одно- и одну двудоменную архитектуры (или просто добавить к полученным результатам работу по однодоменной архитектуре). Возможно, тогда удалось бы проследить эволюцию появления какого-либо домена. В то же время, невозможно было иначе выбрать архитектуры вообще (кроме однодоменной), потому что для каспазного домена есть только две распространенные архитектуры. К сожалению, они очень ограничены по распространению (только в эукариотах, в основном в хордовых). Остальные архитектуры также только для узких кругов представителей.
ЧАСТЬ 2
ЧАСТЬ 2 содержит информацию о каспазном домене в последовательностях с различными архитектурами, их выравнивания и филогению.
Архитектуры в последовательностях организмов разных таксонов - 2
В этой части работы также использовалось выравнивание всех отобранных последовательностей. Проект JalView был усовершенствован. На риснуке 3 приведено это выравнивание с отмеченными доменами.

Рис. 3. Выравнивание последовательностей с различными архитектурами. Красным выделен домен CARD, синим - домен DED, зеленым - каспазный домен peptidase_C14.
Каспазный домен начинается примерно с 705 позиции выравнивания и тянется до конца. Это соответствует позициям каспазного домена в последовательности CASP1_HUMAN, для которой известна вторичная структура (163-400 а.о.).
Был выделен каспазный домен во всех выравниваниях. Так, получилось выравнивание длиной 350 а.о. (файл в формате fasta). На рисунке 4 представлено выравнивание только каспазного домена.

Рис. 4. Выравнивание каспазного домена.
Филогения последовательностей с каспазным доменом - 2
На основе полученного выравнивания каспазного домена в разных последовательностях по описанному выше (часть 1) способу было получено филогенетическое дерево. На рисунках 5-6 изображены соответственно original tree и bootstrap consensus tree.
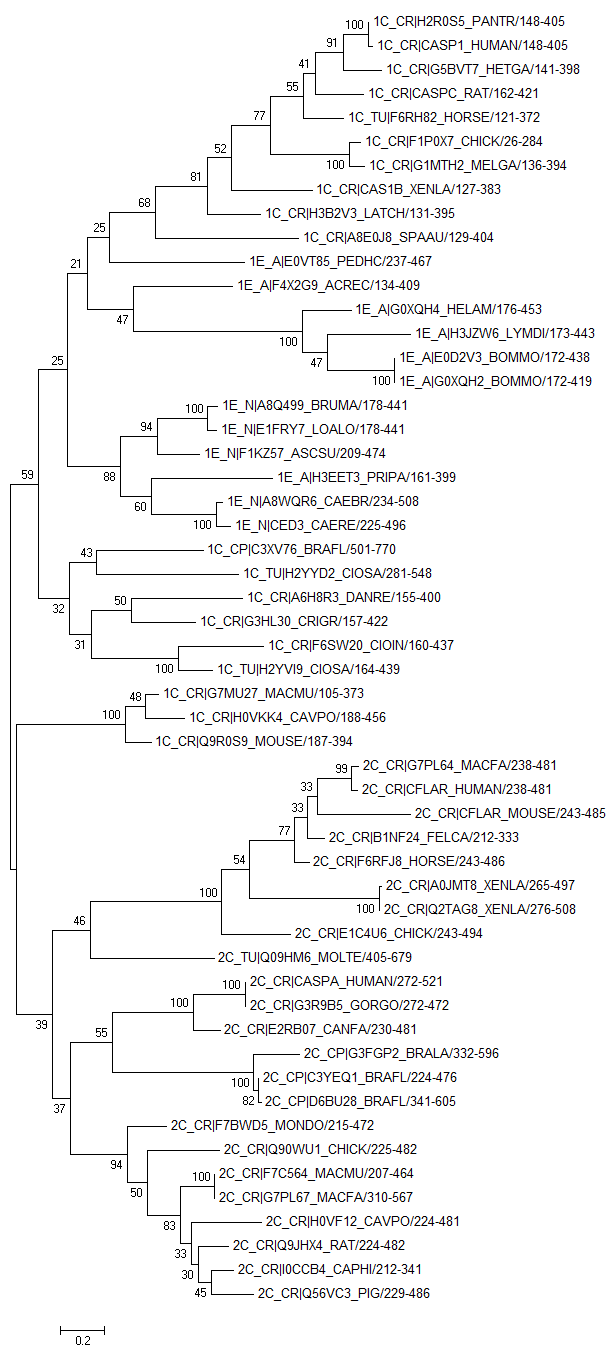
Рис. 5. Original tree.
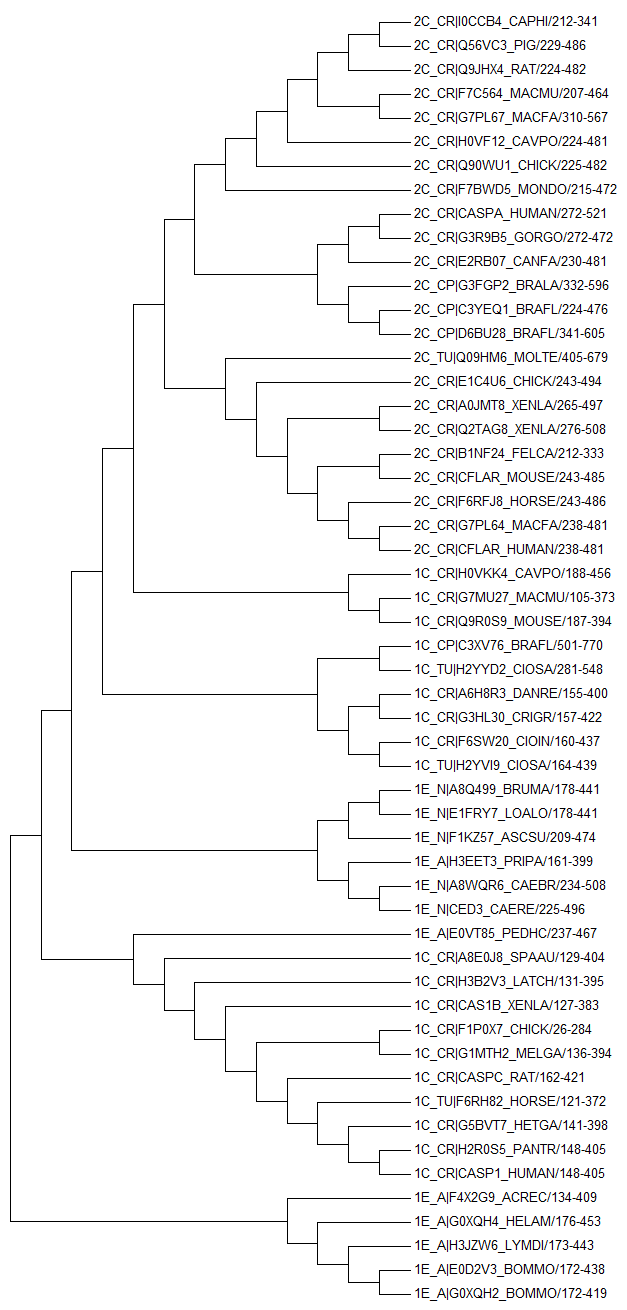
Рис. 6. Bootstrap consensus tree.
Интересно, что деревья получились практически точно совпадающими с таковыми, полученными на основе выравниваний полноразмерных последовательностей белков.
Две двудоменные архитектуры разделены на дереве на две клады. Вероятно, все три домена имелись у общего предка, или общий предок имел обе архитектуры. В большинстве случаев разделение на таксоны прослеживается достаточно четко.
Ветви, разделяющие большие группы последовательностей, имеют слабую поддержку, в то время как большую поддержку имеют ветви, которые разделяют последовательности, имеющие домены, сходные по последовательности. К последним относятся в основном последовательности организмов, принадлежащих одному таксону.