Функциональная роль гена в подсистеме. GO, SEED, String
Система секреции, к которой принадлежит ген
Данная работа посвящена белку spiA (АС: Q8Z6L1), принадлежащему организму Salmonella typhi, и подсистеме, включающей ген spiA.
В данном разделе ипользуется информация из базы данных GO (Gene Ontology)
Белок относится к системе protein secretion (секреция белков), является белком внешней мембраны, обладает транспортной активностью, принадлежит к секреторная системе III типа. На рисунке 1 приведена схема, иллюстрирующая работу данной системы.
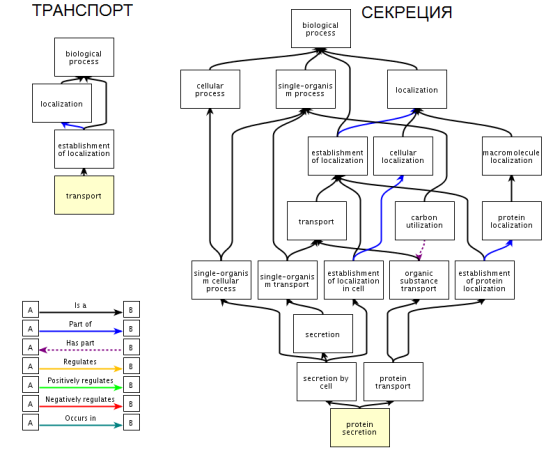
Рис. 1. Схема работы систем транспорта и секреции (к которым относится spiA).
Ген spiA имеет OrderedLocusNames - t1262. С его помощью была найдена система гена в БД KEGG. Там же была найдена схема системы бактериальной секреции.
Белок принадлежит бактерии из типа протеобактерий, которые являются грамм-отрицательными. У грамм-отрицательных бактерий 2 мембраны, секреция топологически более сложна и существует по меньшей мере 6 специализированных секреторных систем.
Секреторная система III типа ответственна за одноэтапный транспорт эффекторных молекул патогенности из цитоплазмы бактерии в цитозоль эукариотической клетки макроорганизма. Также система отвечает сборку супермолекулярных транспортных структур на поверхности клетки. Секреция эффекторных белков данной системой происходит непосредственно после контакта возбудителя с клеткой хозяина, поэтому ее называют контакт-зависимой системой секреции.
Функция интересуюещего нас белка spiA на данный момент не описана (он относится сразу ко многим классами: Environmental Information Processing; Membrane transport; Bacterial secretion system). Тем не менее, есть информация о его близком гомологе spiC. Еще в 1999 году вышла работа (PMID: 10406797), в которой показали, что spiC выходит наружу клетки через систему секреции III и входит в цитозоль других клеток (макрофагов) и подавляет в них транспортные системы.
Термины GO, ассоциированные c геном и с этой системой
Идентификаторы GO находятся в поле DR записи и бывают трех типов: F – function, P – biological process, C – cellular component.
Для spiA указаны два биологических процесса, одна структура (клеточная мембрана) и биологический процесс. Вся информация представлена в таблице 1.
Таблица 1. Идентификаторы GO для spiA и расшифровки к ним.
| GO идентификатор | GO Term Name | GO название класс | Тип | Название словаря |
| GO:0006810 | transport | транспорт | P | Биологический процесс |
| GO:0009306 | protein secretion | белки секреции | P | Биологический процесс |
| GO:0008565 | protein transporter activity | белки с транспортной активностью | F | Молекулярная функция |
| GO:0009279 | cell outer membrane | клеточная внешняя мембрана | C | Клеточные компоненты |
Ортологи белка в полных геномах бактерий. Сравнение состава генов
С помощью базы данных SEED был проведен BLAST с последовательностью белка spiA по геномам Salmonella Typhi (штаммы Ty2, CT18) и близкородственных бактерий Salmonella bongori 12149 и Salmonella typhimurium LT2. Был выбран один ген. На странице этого гена расположено графическое окно с четырьмя гомологами из разных геномов. Для получения ортологов были выставлены следующие параметры: Number of Regions – 50, Evalue cutoff – 1e-7. Затем были отобраны 18 ортологов из бактерий разных родов. Для отбора внимание обращалось на длину гена, были выбраны бактерии с соизмеримыми длинами генов. На рисунке 2 представлена карта окрестностей отобранных ортологов.
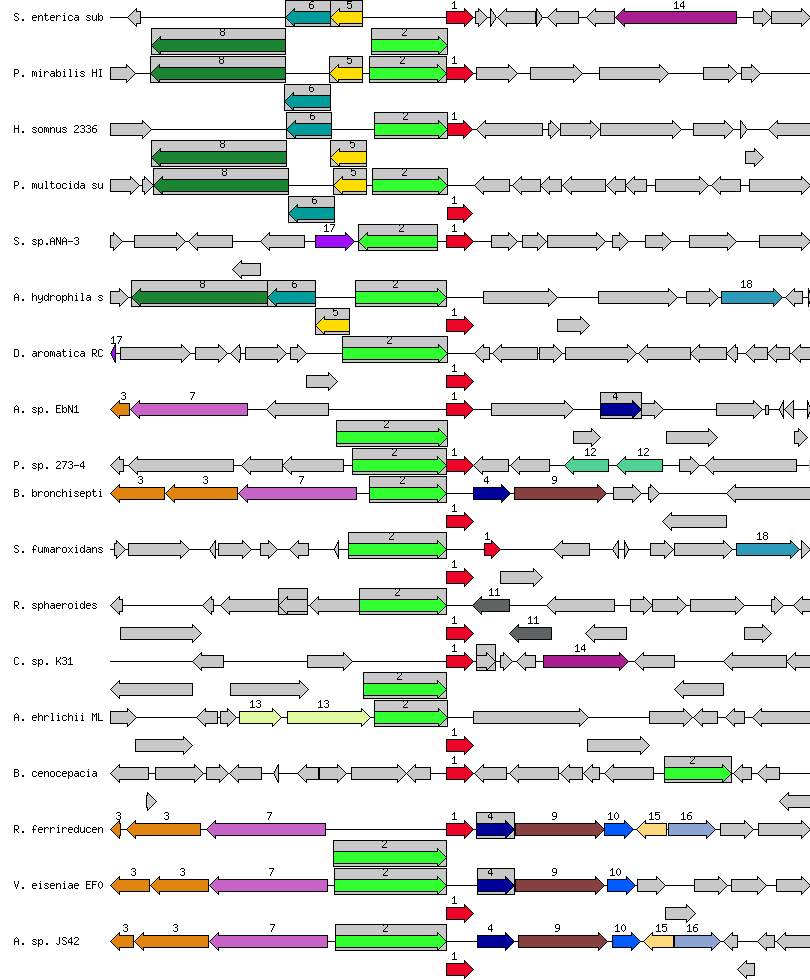
Рис. 2. Окрестности генов, кодирующих ортологи spiA в бактериях различных организмах. Параметры поиска ортологов: Number of Regions – 50, Evalue cutoff – 1e-7.
Перед обсуждением полученного результата была получена таблица всех генов окрестностей. Лист "исходная информация" содержит таблицу, полученную в SEED. На ее основне получена сводная таблица (лист "Сводная таблица"), где по строкам – геномы, по столбцам – группы ортологов, тех, которые отмечены одним цветом и имеют один номер (колонка Set). В колонке SS отмечены номера подсистем, к которым отнесен данный ген (их бывает несколько). Они были изучены автором, так как такая информация может помочь с выводами о генах определенной подсистемы.
Белок spiA обозначен на карте цифрой 1 (стрелка красного цвета). В некоторых бактериях (первые 6) можно видеть расположенные в его небольшой окрестности гены, кодирующие 3 субъединицы (А, В, С; 8, 6, 5 гены соответственно) тетратионат редуктазы, играющей роль в дыхании с использованием тетратионат-иона. Такой процесс характерен для сальмонеллы. Также в некоторых бактериях в окружении spiA встречаются гены пируват дегдирогеназы (ее комплексов, гены 7 и 3). Перечисленные гены кодируются на обратной цепи, в то время как почти во всех ортологах spiA находится на прямой цепи.
Ген NADP+ дегидрогеназы (ген 4) представлен в окружении spiA в четырех бактериях и находится на прямой цепи.
Но лишь один ген встречается почти во всех отобранных бактериях в окружении ортологов spiA (ген 2). Это позволяет сделать выывод, что лишь лишь он ко-локализован с исходным геном. Это ген, который регулирует деятельность тетратионат редуктазы, которая уже была упомянута в данном разделе.
В самом деле в аннотации SEED есть информация о ко-локализации гена белка spiA с геном, кодирующим белок с этой функцией (Set function to Tetrathionate reductase two-component response regulator based on FIGfams).
Просмотр окрестностей гена больше 16000 (30000) не показало никаких гомологичных участков и вообще ничего интересного.
Для выравнивания ортологов spiA в выбранных бактериях была использована возможности сервиса SEED в разделе Sequences для выбранных организмов: Align sequences.
Было получено выравнивание (в программе используется сервис Clustal).
Полученное вырвавнивание интегрировано в Jal-View, по ссылке можно ознакомится с проектом в формате .jar. На рисунке 3 приведено выравнивание с раскраской по Clustalx (уровень by conservation 30%).
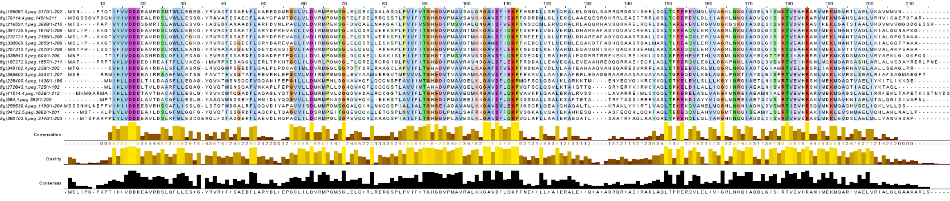
Рис. 3. Выравнивание ортологов spiA в выбранных бактериях.
Annotation Overview for fig|41514.4.peg.1526 in Salmonella enterica subsp. arizonae serovar 62:z4,z23:--: Tetrathionate reductase two-component response regulator
Гены подсистемы могут быть не колокализованы
Разумеется, из всех правил есть исключения. Существуют геномы, в которых гены подсистемы, колокализованные в одной выборке, не колокализованы.
В данном случае была обнаружена колокализация двух генов. Поиск проводился по 18 ортологов spiA из бактерий разных родов, отобранных из найденых в SEED ортологов 50 организмов.
В 17 случаях мы увидели в окрестности гена spiA второй ген. Такая подсистема существует по данным SEED. Так, мы сделали вывод о том, что гены колокализованы. Тем не менее, в 1 случае B. cenocepacia в окрестности ортолога spiA мы увидили отсутствие второго гена. Логично предположить, что spiA может встречаться еще раз в геноме бактерии. Однако, судя по всему, это не так. Был проведен сначала белковый blast для последовательности B1K4V5_BURCC внутри организма Burkholderia cenocepacia. Он выдал только одну последовательность (белок сам с собой). Никаких предсказанных по геному белков не обанаружено. Тем не менее, затем был проведен нуклеотидный blast с последовательностью, кодирующей это белок. Но и в этом случае не было новых результатов. Это означает, что ген один в геноме. Также, судя по всему, и сервис SEED показывает окружения всех генов (кодирующий данный ортолог), которые он нашел в определенном организме.
Таким образом, ген ортолога spiA в бактерии B. cenocepacia не колокализован с геном регуляции тетратионат редуктазы. Так, этот случай является примером гена подсистемы, который может быть не колокализован.
База данных String
Задачи данной работы можно решить с помощью базы данных String. Интерфейс данной базы данных более приветлив, оформление более современно и изящно. Есть хелп.
Был проведен поиск гена spiA. В String можно обнаружить новое представление окрестностей гена, оно представлено на рисуке 4.

Рис. 4. Окрестности гена spiA, база данных String.
В String можно легко переходить с белка на белок, сразу предоставляются аминокислотные последовательности, легко и быстро ищутся гомологи белка. Conserved Neighborhood View позволяет увидеть, гены, соседние данному в различных геномах (прокариотических). Здесь возможно отобразить гены, пропорционально их размеру. На мой взгляд, такой просмотр удобнее, чем в БД SEED.
Стоит отметить, что в данной БД в системе секреции типа III представлено больше белков, чем в БД SEED. БД String предоставляет и другие возможности. Можно убедиться, посмотрев в хелпе.
Интересно также посмотреть на карту evidence view, отображающую ассоциации между генами, она предствлена на рисунке 5. Данная схема создает представление о других белках системы, известных для данной бактерии. Более сильные ассоциации на ней представлены более толстыми линиями.
Рис. 5. Взаимодействия между белком spiA и другими.