Реконструкция филогенетических деревьев
Таксономия: стандарт NCBI
С помощью таксономического сервиса NCBI были определены таксоны, к которым относятся отобранные для работы бактерии (см. часть I).
| Название | Мнемоника | Таксоны (домен; тип; класс; порядок; семейство; род; группа, если указана) |
| Bacillus subtilis | BACSU | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium tetani | CLOTE | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Geobacillus kaustophilus | GEOKA | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus acidophilus | LACAC | Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactobacillus delbrueckii | LACDA | Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Lactococcus lactis | LACLM | Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Listeria monocytogenes | LISMO | Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus aureus | STAA1 | Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
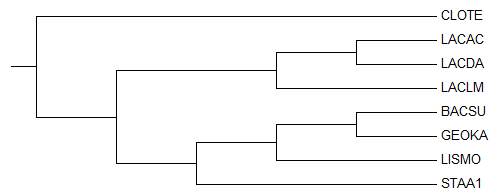
Видим, что на дереве отобранных бактерий есть ветви, выделяющие различные таксоны.
Все представленные бактерии относятся к типу Firmicutes, из 7 из 8 бактерий относятся к классу Bacilli. Бактерия Clostridium tetani относится к классу Clostridia - верхняя ветвь дерева CLOTE.
4 из 7 бактерий класса Bacilli относятся к порядку Bacillales - нетривиальная ветвь: {BACSU, GEOKA, LISMO, STAA1} против {CLOTE, LACAC, LACDA LACLM}. 3 из 7 относятся к порядку Lactobacillales - нетривиальная ветвь {LACAC, LACDA LACLM} против {CLOTE, BACSU, GEOKA, LISMO, STAA1}.
2 из 4 бактерий порядка Bacillales относятся к одному семейству Bacillaceae - нетривиальная ветвь {BACSU, GEOKA} против {CLOTE, LACAC, LACDA, LACLM, LISMO, STAA1}. Другие 2 бактерии относятся к разным семействам порядка Bacillales - ветви LISMO и STAA1.
Бактерии порядка Lactobacillales принадлежат 2 разным семействам: {LACAC,LACDA} - ветвь бактерий сеейства Lactobacillaceae, LACLM - бактерия семейства Streptococcaceae.
Реконструкция филогенетического дерева по белкам одного семейства
Для реконструкции филогенетического дерева по белкам была выбрана функциональная группа шапероновых белков: семейство шаперонин - HSLO.
Из банка Swiss-Prot были получены последовательности белков семейства HSLO из отобранных бактерий (hslo_8.fasta, в названиях последовательностей оставлена только мнемоника вида).
Было получено выравнивание последовательностей с помощью программы muscle (aligment_hslo_8.fasta). В программе JalView получено изображение выравнивания в "блочной" форме (Format - Wrap) с раскраской по проценту идентичности:

Диагностические позиции выравнивания
Диагностические позиции выравнивания - это позиции, по которым можно судить о том, относится ли та или иная последовательность выравнивания к некоторому таксону.
Была попытка выявить такие позиции в данном вырававнивании. Результат представлен на изображении выравнивания ниже.
Для удобства последовательности в порядке, соответствующем филогенетическому дереву. Диагностические позиции были заключены в рамки в графическом редакторе (автор данной работы считает, что это удобнее, чем использование JalView).
При поиске интересующих позиций внимание обращалось на свойства аминокислот и их консервативность для последовательностей белков, принадлежащих организмам разных таксонов. Так, считаем позицию консервативной для класса, если она консервативна в соответствующих последовательностях. Но если, например, консервативна позиция лейцина, а в остальных отобранных последовательностях встречается изолейцин, то эту позицию из диагностических позиций убираем. Аналогично с другими аминокислотами с близкими свойствами. Возможно, такая позиция и является диагностической, но, с другой стороны, она может быть и не достоверной. Для проверки необходимо увеличивать объем выборки бактерий.
На выравнивании ниже красным отмечены консервативные позиции таксона Clostridiales, зеленым - Lactobacillales, оранжевым - Bacillales.
Большее количество диагностических позиций даже в этом выравнивании можно найти для бактерий отдельных семейств (Lactobacillaceae, Bacillaceae), но в данной работе мы ограничимся порядками.

Реконструкция филогенетического дерева с помощью JalView
Была проведена реконструкция дерева 4 методами, доступными из JalView (меню Calculate > Calculate tree). Каждое дерево было сохранено в Newick-формате для дальнейшего экспорта в программу Mega. На рисунках 1-4 приведены изображения деревьев, созданных Mega (метод JalView указан в названии рисунка). Произведено сравнение топологии с исходным деревом (на рисунке выше), новые и отсутствующие ветви указаны в тексте.
Рис 1. Average distance tree using PID (% Identity)
Новые ветви:
{LACLM, STAA1, LISMO, GEOKA, BACSU}
{STAA1, GEOKA, BACSU}
Отсутствующие ветви: нет.
{LACLM, LACAC, LACDA}
{LISMO, GEOKA, BACSU}
Рис 2. Neigbour joining tree using PID (% Identity)
Новые ветви:
{STAA1, GEOKA, BACSU}
Отсуствующие ветви:
{LISMO, GEOKA, BACSU}
{LACLM, LACAC, LACDA}
Рис 3. Average distance tree using BLOSUM62
Новые ветви:
{LACAC, LACDA, STAA1, LISMO, GEOKA, BACSU}
{STAA1, GEOKA, BACSU}
Отсуствующие ветви:
{LISMO, GEOKA, BACSU}
{LACLM, LACAC, LACDA}
Рис 4. Neigbour joining tree using BLOSUM62
Полученный результат совпадает с таковым для метода Neigbour joining tree using PID (% Identity). Дело в том, что методы похожи. Оценка по BLOSUM62 адекватна, потому что белки семейства HSLO в основном цитоплазматические, к ним применима эта матрица весов. Получается оценка, похожая на ту, что по параметру идентичености.
Новые ветви:
{STAA1, GEOKA, BACSU}
Отсуствующие ветви:
{LISMO, GEOKA, BACSU}
{LACLM, LACAC, LACDA}
Реконструкция филогенетического дерева методом "Maximum Parsimony"
Dыравнивание (в fasta-формате) было импортировано в программу Mega ("Analyze"). Дерево реконструируировано методом "Maximum Parsimony" (Phylogeny).
Дерево укоренено в наоболее правильную ветвь (Subtree > Root). Изображение приведено на рисунке 5. Снова было проведено сравнение топологии дерева с исходным.
Рис. 5. "Maximum Parsimony"
Новые ветви:
{LACALM, STAA1, LISMO, GEOKA, BACSU}
{STAA1, GEOKA, BACSU}
Отсуствующие ветви:
{LACLM, LACAC, LACDA}
{LISMO, GEOKA, BACSU}
Наблюдения. CLOTE во всех деревьях отделяется от других организмов и имеет с ними некого общего предка, что говорит о том, что в данной выборке по семействую белков HSLO легко выделяется соответствующий таксон. GEOKA и BACSU, а также LACAC и LACDA, аналогично, присутствуют во всех деревьях. Это свидетельстует о близости бактерий и белковых последовательностей. К сожалению, ни один метод по данным последовательностям не восстановил оригинальное дерево.