Реконструкция деревьев по нуклеотидным последовательностям. Деревья, содержащие паралоги
Построение дерева по нуклеотидным последовательностям
Данная работа посвящена постоению филогенетического дерева взятых для изучения бактерий при помощи последовательностей РНК малой субъединицы рибосомы (16S rRNA).
В Uniprot находили записи о некоторых (принципиально - любых) белках данных бактерий и находили полногеномные записи EMBL, на которые ссыллаются в аннотациях к найденным белкам. В записях EMBL найдем соответствующие"особенности" (FT), с ключом (FTkey) "rRNA", "16S rRNA". Координаты интересующих последовательностей записиывались для последующего вырезания в отдельные файлы командой:
Все необходимые данные о взятых последовательностях приведены в таблице 1.
| Название | Мнемоника | AC записи EMBL | Координаты 16S рРНК | Цепь |
| Bacillus subtilis | BACSU | AL009126 | 9810-11364 | Прямая |
| Clostridium tetani | CLOTE | AE015927 | 8715-10223 | Прямая |
| Geobacillus kaustophilus | GEOKA | BA000043 | 30790..32343 | Прямая |
| Lactobacillus acidophilus | LACAC | CP000033 | 59255-60826 | Прямая |
| Lactobacillus delbrueckii | LACDA | CR954253 | 45160..46720 | Прямая |
| Lactococcus lactis | LACLM | CP002365 | 470580-472133 | Прямая |
| Listeria monocytogenes | LISMO | AL591981 | 99187..100732 | Обратная |
| Staphylococcus aureus | STAA1 | CP003808 | 540419-542060 | Прямая |
Таблица 1. Данные о последовательностях.
Нуклеотидные последовательности были собраны в одном файле (указаны названия организмов), после чего они были выровнены программой muscle:
Результат представлен в файле выравнивания.
Дерево реконструировалось в программе MEGA6 с помощью метода максимальной правдоподобности (Maximum Likelihood). Полученное дерево приведено на рисунке 1.
Рис.1. Реконструированное дерево (пояснения в тексте)
На рисунке 2 для сравнения приведено правильное дерево.
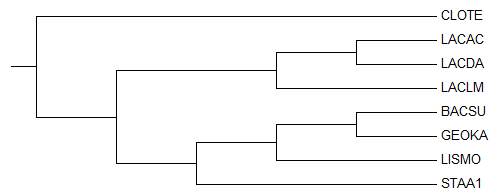
Рис.2. Правильное дерево
Сравнение деревьев. Нетривиальная ветвь дерева с сохраненной структурой можно рассматривать как отдельное дерево. Поэтому опишем реконструированное дерево через "поддеревья", которые в нем появились и которых в нем не хватает. В реконструированном дереве:
- присутствует поддерево (((STAA1,LISMO),BACSU),GEOKA);
- вместо правильного (((BACSU,GEOKA),LISMO),STAA1) (отсуствует в реконструкции).
Оценка реконструкции по нуклеотидным последовательностям. Реконструированное дерево оказалось достаточно близким к правильному. Тем не менее, результаты могли быть и лучше, т.к. вероятность случайного совпадения при анализе нуклеотидных последовательностей меньше, чем при анализе белков. Это связано с тем, что стандартных нуклеотидов в 5 раз меньше, чем аминокислот, и многие аминокислоты кодируются несколькими кодонами (до 6). Т.е. нуклеотидные выравнивания позволяют увидеть более реальную картину гомологии. Вероятно, 16S РНК является с одной стороны достаточно консервативной, а с другой стороны и имеет специфические позиции для разных таксонов. Так что выбор этой последовательности для анализа можно назвать удачным.
Построение и анализ дерева, содержащего паралоги
Эта часть работы произведена с целью построение дерево гомологов CLPX, принадлежащих вырбранным бактериям.
С помощью blastp записей банка UNIPROT(с разумным порогом на E-value, скажем, 0,001) были найдены достоверные гомологи белка CLPX_BACSU. Всего было обнаружено 32 гомолога. Последовательности белков можно увидеть в файле clpx_homol.fasta (данные о базах данных и AC белков в файле clpx_homol.txt). Реконструкция проведена в MEGA6 при помощи метода Neighbour Joining c использованием Bootstrap, чтобы было ясно, какие ветви вообще интересно рассматривать для опиания гомологов. Дерево гомологов представлено на рисунке 3.
Рис.3. Дерево гомологов CPLX изучаемых бактерий.
Два гомологичных белка - ортологи, если они а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Примеры: CLPX_BACSU и CLPX_GEOKA, HSLU_STAA1 и HSLU_LISMO.
Два гомологичных белка из одного организма - паралоги.
Примеры: Q8Y8B1_LISMO и Q8YAB6_LISMO, Q891B9_CLOTE и Q899H3_CLOTE.