
ФББ 2013-2014

ФББ 2013-2014
Построение множественных выравниваний. Pfam.
BLAST и PSI-BLAST являются удобными инструментами для поиска семейств гомологичных белков.
Проект Jalview, содержащий все выравнивания из данного практикума, можно скачать здесь.
В задании 1 требовалось собрать выборку последовательностей, гомологичных белку с идентификатором NP_661963.1. Я сделала это, используя средства BLAST. В выборку включались последовательности с покрытием 75-90% и ID = 50-65%. Всего мной было отобрано 8 белков, из них с двумя белками я уже работала в практикуме 11 (задания 1 и 3). Большинство белков из выборки имеют в описании "thiol:disulfide interchange protein DsbE", но также я взяла 1 "hypotetical protein". Всего получилось 8 штук белков, выравнивание которых я буду строить.
Рис.1. Результаты поиска BLAST, галочками отмечены выбранные белки.
Задание 2 было выполнено на сервере kodomo, на который я зашла с помощью Putty. Я использовала команду MUSCLE (MUltiple Sequence Comparison by Log-Expectation), полный синтаксис выглядел так:
muscle -in seqdump.txt -out mus_align.fasta
Полученное выравнивание, открытое в Jalview, показано на рисунке 2. Очевидно, что белки действительно входят в одно семейство, поскольку в выравнивании есть несколько консервативных блоков. Скачать выравнивание в формате fasta можно скачать здесь.
Рис.2. Общий вид выравнивания, сделанного с помощью MUSCLE, в Jalview. Раскраска BLOSUM62, консервативность выше 80%.
Задание 3 также было выполнено с помощью пакета EMBOSS на kodomo. Mafft (Multiple Alignment using Fast Fourier Transform), так же, как и MUSCLE, является популярным инструментом построения множественных выравниваний. Обе программы считаются самыми оптимальными для использования на обычном домашнем компьютере, поскольку их алгоритмы позволяют не сильно загружать память. Они уступают в аккуратности некоторым другим программам (например, T-coffee), зато используют мало вычислительной мощности. Выравнивание было получено командой:
mafft seqdump.txt > mafftalign.fasta
На рисунке 3 - участок выравнивания, полученный с помощью Mafft. Скачать его в формате fasta можно здесь.
Рис.3. Общий вид выравнивания, сделанного с помощью Mafft, в Jalview. Раскраска BLOSUM62, консервативность выше 80%.
Задание 4 я выполняла на сервере kodomo с помощью следующей команды:
muscle -profile -in1 mus_align.fasta -in2 mafftalign.fasta -out comparison.fasta
На вход было подано 2 составленных ранее выравнивания, на выходе было получено их сравнение в формате fasta. Общий вид окошка Jalview со сравнением можно увидеть на рисунке 4. Сравнение выравниваний в формате fasta можно скачать здесь.
Рис.4. Сравнение выравниваний в Jalview. Серым выделены последовательности, выровненные с помощью mafft. Раскраска BLOSUM62, порог консервативности - 80%.
Как видно из сравнения выравниваний, на участке, где должен располагаться консервативный домен и на концах последовательностей выравнивания одинаковые. На тех участках, где гомология очевидна, оба алгоритма дали одинаковый результат. Однако на начальных участках выравнивания отличаются. Это различие возникло в результате того, что я включила в выборку эукариотический белок клещевины, который длиннее, чем остальные белки в выборке. Поэтому некоторые начальные фрагменты оставшихся 7ми белков выравнивались относительно этого "хвоста" по-разному при использоватнии разных алгоритмов.
Я считаю, что обе программы хорошо справились с задачей, выравнивания гомологичных участков совпадают, а выравнивание негомологичных участков не имеют биологического значения. Однако филогинетические деревья, построенные по этим двум выравниваниям, отличаются довольно сильно. Оба дерева представлены на рисунке 5.
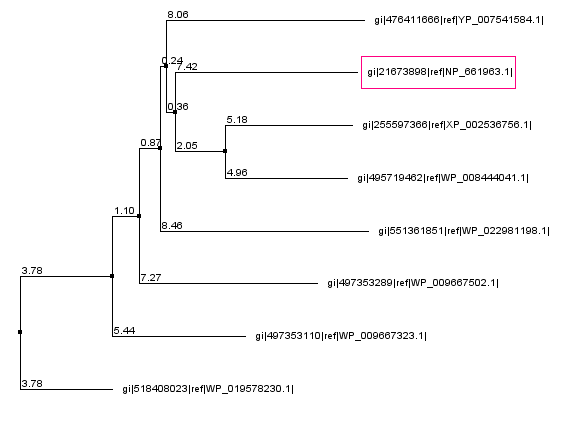

Рис.5. Дерево выравнивания, построенное по muscle-выравниванию - слева, по mafft-выравниванию - справа. В розовом прямоугольнике - исходный белок.
Чтобы определить, какое из построенных деревьей ближе к истине, я решила построить множественное выравнивание данных последовательностей, используя алгоритм T-coffee. Этот алгоритм работает дольше, но и выравнивает точнее. Скачать множественное выравнивание, полученное с помощью T-coffee можно здесь. Построенное по этому выравниванию дерево - на рисунке 6. T-coffee дерево, на мой взгляд, больше похоже на mafft-дерево, поэтому я думаю, что в данном случае mafft-алгоритм сработал лучше.

Рис.6. Дерево выравнивания, построенное по t-coffee выравниванию.
Для выполнения задания 5 я осуществила поиск по базе данных доменов Pfam. Результат поиска: в белке с
Refseq идентификатором есть 1 домен - redoxin.
Очевидно, что домен изучен плохо, потому что почти нет никакой информации о нём. Из статьи в Pfam и InterPro стало понятно, что
семейство редоксинов включает тиоредоксины (сюда относится мой белок), глютаредоксины, пероксиредоксины. Они играют важную роль в
защите от антиоксидантов и регулируют кадмий-чувствительные белки. Из 9684 белков с этим доменом 8782 обладают только им. То есть
чаще всего это довольно короткие белки. Намного реже этот домен встречается в сочетании с другими доменами (dbsD, glutaredoxin - самые
популярные).
Также с помощью Pfam я построила круговую схему, отобращающую разнообразие таксонов с этим доменом (рисунок 7). Из неё становится очевидно, что домен встречается у всех живых организмов (кроме вирусов), является универсальным. Видимо, так случилось, потому что антиоксидантная защита нужна всем.
Рис.7. Схема разнообразия таксонов с redoxin-доменом.