
ФББ 2013-2014

ФББ 2013-2014
Описание пространственной структуры белка дисульфидного обмена Chlorobium tepidum TLS
Информация была получена из базы данных pdb-структур белков. Идентификатор - 3GL3. Некоторая информация, полученная из файла:
TITLE CRYSTAL STRUCTURE OF A PUTATIVE THIOL:DISULFIDE INTERCHANGE TITLE 2 PROTEIN DSBE FROM CHLOROBIUM TEPIDUM SOURCE 2 ORGANISM_SCIENTIFIC: CHLOROBIUM TEPIDUM TLS; COMPND MOL_ID: 1; COMPND 2 MOLECULE: PUTATIVE THIOL:DISULFIDE INTERCHANGE PROTEIN COMPND 3 DSBE; COMPND 4 CHAIN: A, B, C, D; COMPND 5 FRAGMENT: UNP RESIDUES 30-170; COMPND 6 ENGINEERED: YES HETNAM MSE SELENOMETHIONINE FORMUL 1 MSE 16(C5 H11 N O2 SE) FORMUL 5 HOH *201(H2 O) EXPDTA X-RAY DIFFRACTION REMARK 2 REMARK 2 RESOLUTION. 2.09 ANGSTROMS.
Расшифровка структуры проводилась методом рентгеноструктурного анализа кристаллов с длиной волны 2 ангстрема. Этот метод основан на том, что анализируется рассеянное атомами кристалла рентгеновское излучение, благодаря чему удаётся воссоздать распределение электронной плотности в кристалле ( более подробно ).
Название белка, структуру которого расшифровывали - белок дисульфидного обмена. Организм, в котором обнаружили этот белок - Chlorobium tepidum TLS. Представлена информация о подсемействе, в которое входит белок (dsbE). Он состоит из 4х цепей (A, B, C, D). В его состав входит необычная аминокислота - селенметионин. Однако возможно, что эта аминокислота оказалась из-за метода, с помощью которого расшифровывалась структура. В качестве лигандов выступают многочисленные (201) молекулы воды.
На рисунке 1 представлена третичная структура данного белка. Мы действительно видим 4 цепи и окружающие глобулу молекулы воды.
Обозначения, принятые на рисунке:
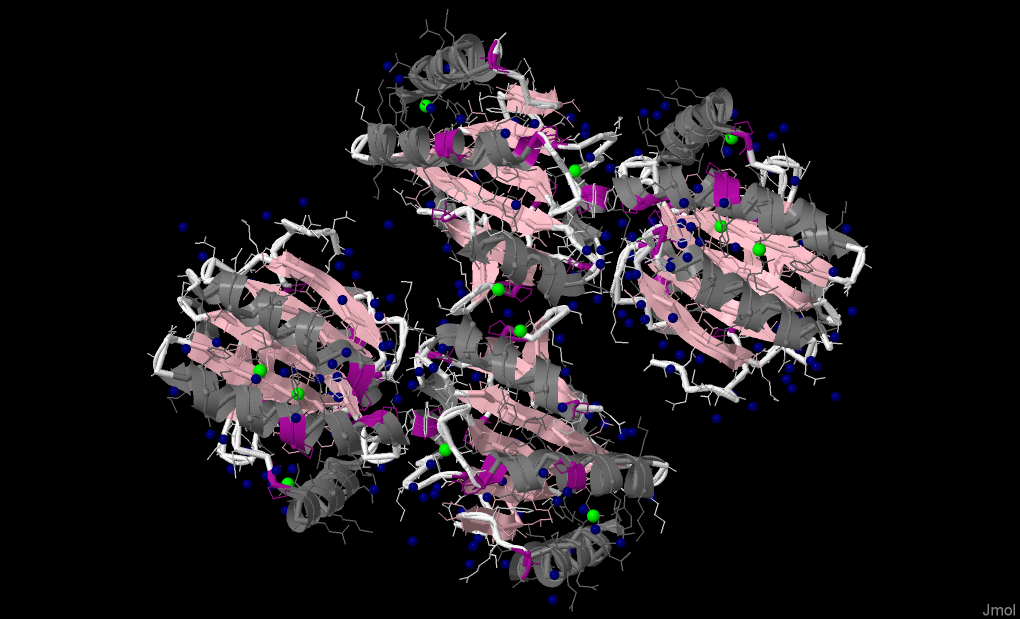
Рис. 1. Третичная структура белка дисульфидного обмена Chlorobium tepidum TLS.
Скачать оригинальный pdb-файл: 3GL3.pdb
Скачать скрипт, изменяющий оригинальное изображение на то, что представлено на рисунке 1: Скрипт Jmol
Для корректной работы скрипт и pdb-файл должны находиться к одной директории.
Также я сделала другое изображение третичной структуры белка, показывающее разнообразие составяющих его аминокислот. На рисунке 2 каждая аминокислота имеет собственный цвет. Получилось красиво.
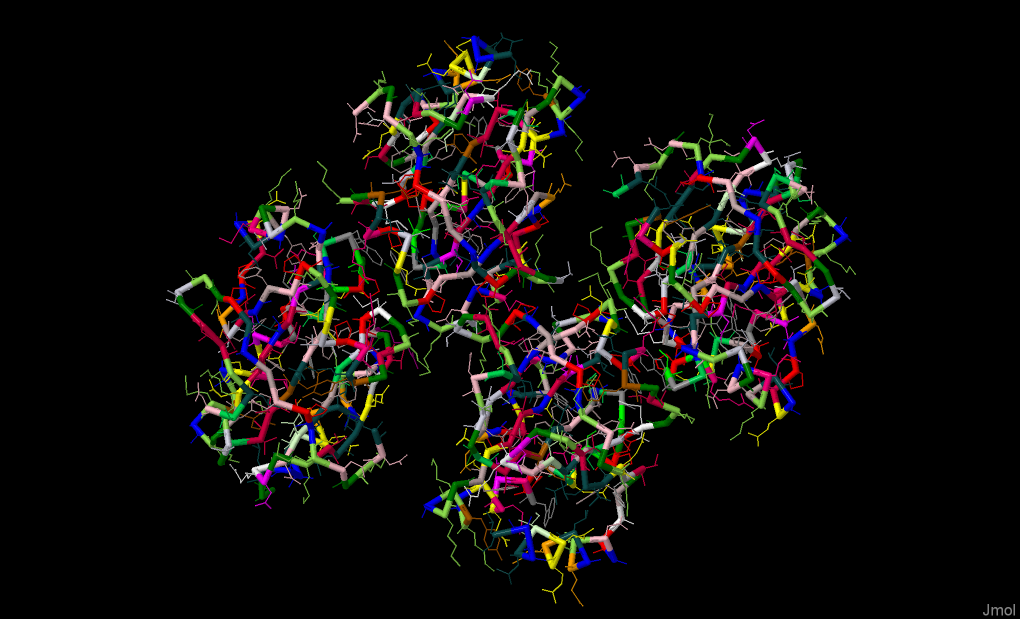
Рис.2. Третичная структура того же самого белка.
Скачать скрипт - Скрипт Jmol №2
Сравнение исходного белка с его биологической единицей (biological assembly/unit).
В базе данных pdb-структур хранятся также pdb файлы с информацией о функциональной форме этих белков в клетке. В случае белка дисульфидного обмена Chlorobium tepidum TLS эти структуры никак не отличаются (внешний осмотр).
Чтобы удостовериться в этом, я решила скачать программу, которая умеет сравнивать 2 pdb файла (Calculate structure alignment от PDB database). На рисунках 3, 4, 5 показаны результаты этого сравнения.
Рис.3. Начало работы программы
Введём в поля для сравнения имена двух файлов - 3GL3_biounit.pdb (содержит структуру биологической единицы) и 3GL3.pdb (содержит ранее обсуждавшийся белок).
Рис.4. Результаты выравнивания
Как мы видим, цепи совпадают. Это подтверждается также и графической интерпретацией сравнения, где голубым показаны совпадающие части.
Рис.5. Результаты выравнивания (графическая версия)