
ФББ 2013-2014

ФББ 2013-2014
Совмещение полипептидных цепей белков
На этой страничке речь пойдёт о структурных гомологах исследуемого белка со всё ещё неизвестной функцией. Структурными гомологами называются белки, пространственная структура которых совпадает. Как правило, это свидетельствует об их общем происхождении. С помощью структурных гомологов можно сделать некоторые выводы о лигандах белка и его функции в клетке.
Параметры, по которым оценивается структурная гомология:
| Q-score | [Quality of alignment] - комплексная характеристика, учитывающая длину
структурного выравнивания и показатель RMSD. Вычисляется по специальной формуле:
Nalgn*Nalgn
Q = -------------------------
(1+(RMSD/R0)2)*Nres1*Nres2
Максимальное значение - 1, достигается для полностью идентичных белков. Показатель становится выше
при уменьшении RMSD и увеличении длины выравнивания. Обычно по этому показателю сортируют результаты. |
| P-score | -lg от значения P, которое измеряет качество сопоставления. Оно учитывает количество гэпов, длину выравнивания, число совпавших участков со вторичной структурой. Чем больше P-score, тем более статистически значимо данное сопоставление. Даёт приблизительную оценку. Если этот показатель меньше 3, это плохо. |
| Z-score | Показывает значимость сопоставления с точки зрения гауссовой статистики. |
| RMSD | [root-mean-square deviation] среднее квадратичное. Хорошо характеризует большие отклонения.
Рассчитывается между С-альфа атомами совпавших остатков. Результат показывается в ангстремах.
Формула для расчёта:  |
| Nalign | Длина выравнивания, количество совпавших остатков |
| Ng | Количество гэпов |
| %seq | Процент совпавших аминокислотных остатков остатков.
Рассчитывается как: %seq = Nident / Nalign |
| %sse | Процент совпадений элементов вторичной структуры |
Гомология полипептидных цепей белков с pdb-идентификаторами 3GBY и 3SL7
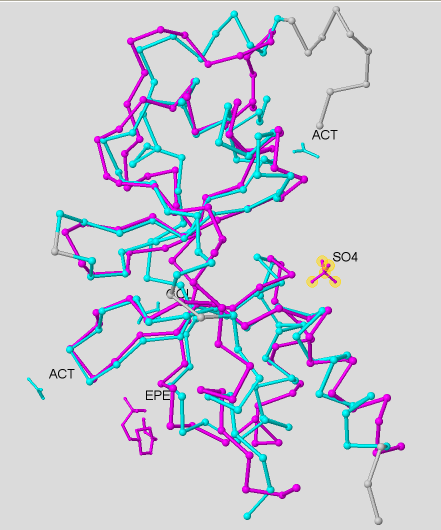
Рис.1. Совмещение двух полипептидных цепей. Розовым показана цепь A моего белка (идентификатор pdb - 3gby), голубым показана цепь B белка с pdb-идентификатором 3sl7 из Arabidopsis thaliana. Рисунок справа - выделенное геометрическое ядро.
При помощи сервиса PDBeFold я провела поиск структурных гомологов белка 3GBY. Программа нашла 97 совпадений. Как мне кажется, это много, но этого можного было ожидать, так как CBS-домены распространены очень широко. Совпадающие белки, как оказалось, принадлежат совершенно разным организмам. Много сходств среди прокариот-экстремофилов, чего и следовало ожидать, но также есть и белки растений, дрожжей, человека. Для выполнения задания я выбрала хлоропластный белок из Arabidopsis thaliana с pdb-идентификатором 3SL3.
Я выбрала именно этот белок, так как у него самый высокий процент совпавших аминокислот, один из самых низких RMSD. Также интересно, что два сравниваемых белка из двух очень неродственных организмах, живущих в разных условиях. Ещё один фактор - для этого белка известен лиганд - АМФ, что частично подтверждает мою догадку, сделанную насчёт лиганда в прошлый раз.
Информация о выравнивании:
| Номер совпадения | 9 |
| Название | CRYSTAL STRUCTURE OF CBS-PAIR PROTEIN, CBSX2 FROM ARABIDOPSIS THALIANA |
| RMSD | 1.73 |
| Идентификатор и цепь | 3sl7:B |
| Nalign | 119 (из 127) |
| Ng | 5 |
| %seq | 21 |
| %sse(Query) | 80 |
| Q-score | 0.59 |
Функция белка из Arabidopsis thaliana довольно любопытна. Белки, содержащие CBS-домены стабилизируют окислительно-восстановительный баланс клетки и влияют на развитие растения путём регуляции тиоредоксиновой системы. Они являются сенсорами, которые определяют уровень аденозин-сождержащих метаболитов в клетке. У растений есть 2 тиоредоксиновые системы - митохондриальная (НАДФ-тиоредоксиновая) и хлоропластная (ферредоксин-тиоредоксиновая) - и обе регулируются белками, содержащими CBS-домены. Данный белок работает в хлоропласте и вместе с другим CBS-содержащим белком (CBSX1) регулирует его тиоредоксиновую систему. Оба белка CBSX1 и CBSX2 попали в структурное выравнивание с исследуемым белком, они очень похожи.
CBSX2 - антипараллельный гомодимер, поэтому скорее всего он связывает сразу 2 аденозин-содержащих лиганда. Лиганд связывается главным образом за счёт положительно заряженных аминокислот, находящихся в сайте связывания, что в целом совпадает со связыванием лиганда в белке 3GBY. [источник ]
В качестве первых объектов я взяла совмещение бета-поворотов (рисунок 2): совмещённые слева цепи идентичны по своей структуре, а совмещённые справа - имеют небольшое отклонение.
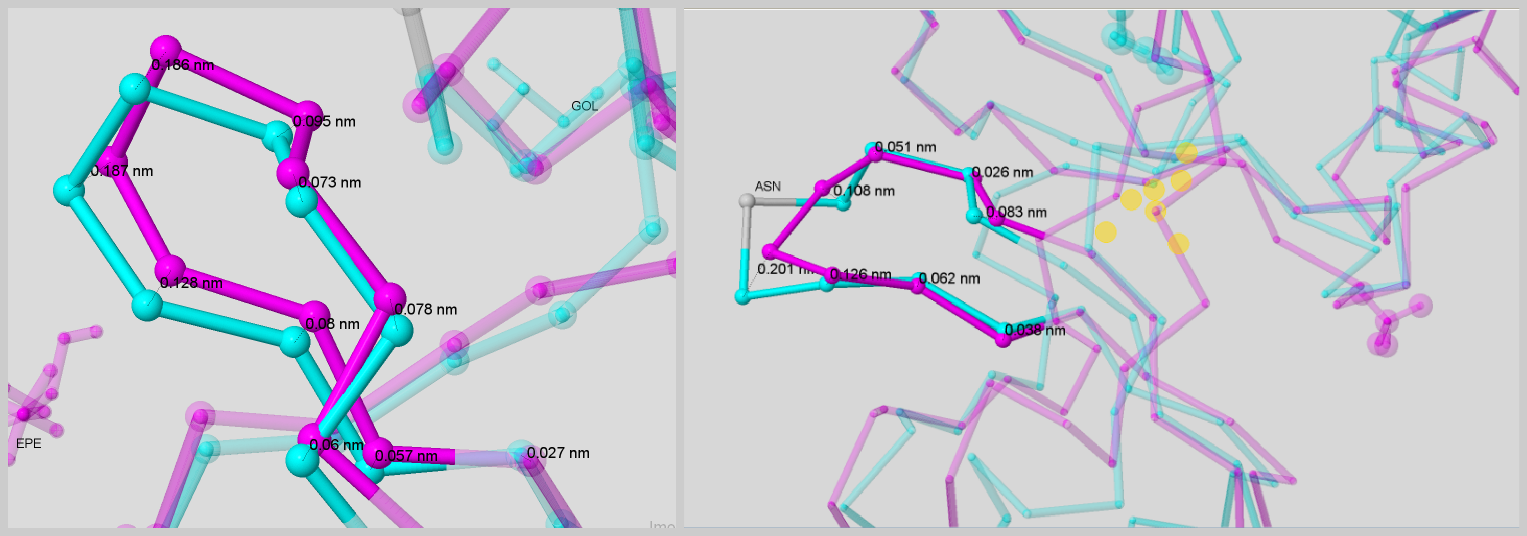
Рис.2. Совмещение двух бета-поворотов. Подписаны расстояния между CA-атомами. Слева - выравнивание аминокислотных остатков 101-109 из цепи А (3gby) и 207-215 из цепи B (3sl7). Cправа - выравнивание аминокислотных остатков 38-45 из цепи А (3gby) и 115-123 из цепи B (3sl7), подписан несовпавший участок.
Также я решила сравнить области контакта с лигандом в этих белках. Результат этого представлен на рисунке 3. Структуры сайтов связывания у этих белков одинаковы по топологии, также высоко сходство и самих аминокислотных остатков. Отклонения мы наблюдаем на конце альфа-спирали.
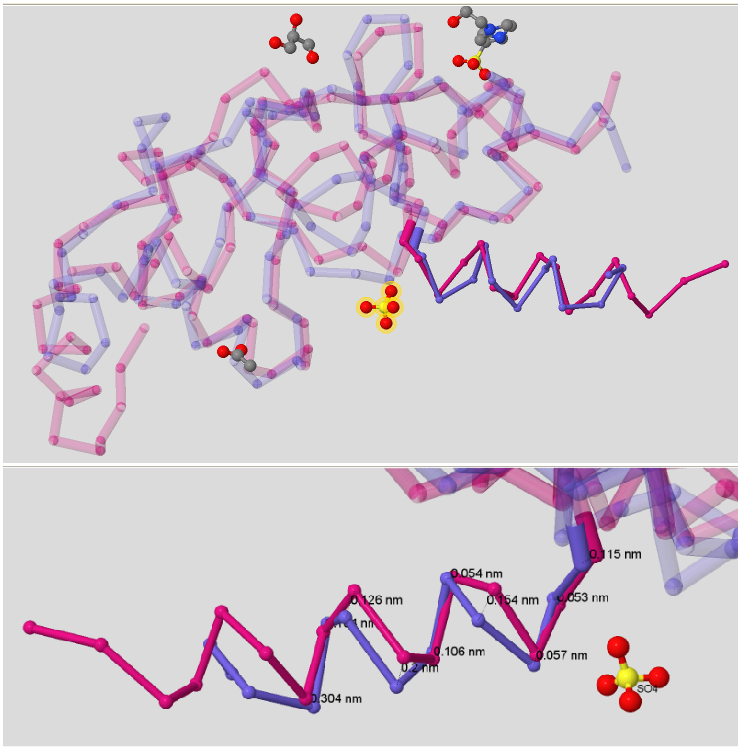
Рис.3. Совмещение альфа-спиралей. Фиолетовым обозначена цепь А моего белка (идентификатор pdb - 3gby), розовым - цепь B белка с pdb-идентификатором 3sl7 из Arabidopsis thaliana. Верхняя картинка показывает расположение данной альфа-спирали в белках, на нижней картинке показаны аминокислотные остатки, которые образуют эти спирали (38-45 из цепи А (3gby) и 115-123 из цепи B (3sl7)). Между совпадающими фрагментами показано расстояние.