
ФББ 2013-2014

ФББ 2013-2014
Множественное выравнивание последовательностей
Выравнивание последовательностей - это метод, который позволяет сделать некоторые выводы о гомологии совмещаемых последовательностей. В множественных выравниваниях последовательности белков или других биологических молекул размещаются друг под другом, при этом совпадающие буквы помещаются друг под другом. Для того, чтобы совместить последовательности, в них часто вставляются гэпы - условные символы, которые нужны для лучшего совмещения. Они не обозначают разрывы последовательности или делеции, а являются чисто формальными символами. Работа с множественными выравниваниями осуществляется с помощью специальных программ, одна из них - Jalview.
Для работы с множественным выравниванием в редакторе Jalview я выбрала специально подготовленное для этого года выравнивание, которое можно скачать здесь. В данном множественном выравнивании - 14 белков, принадлежащих высшим наземным позвоночным - птицам и млекопитающим, есть даже 2 белка человеческого происхождения. Также все белки имеют отношение к иммунной системе. Для большинства из них в аннотациях uniprot указано, что это белки главного комплекса гистосовместимости.
Для выполнения первого задания я установила консервативность 70% и выше, раскраска по типу BLOSUM62. Мне удалось найти 2 участка выравнивания, на которых высок процент консервативных и функционально консервативных аминокислот. Найденные мной вертикальные блоки расположены на участках: 365-406, 329-346 . Несмотря на то, что найденные мной блоки находятся довольно близко, я не считаю, что их можно объединять в кластер, поскольку их разделяют недостаточно консервативные участки.
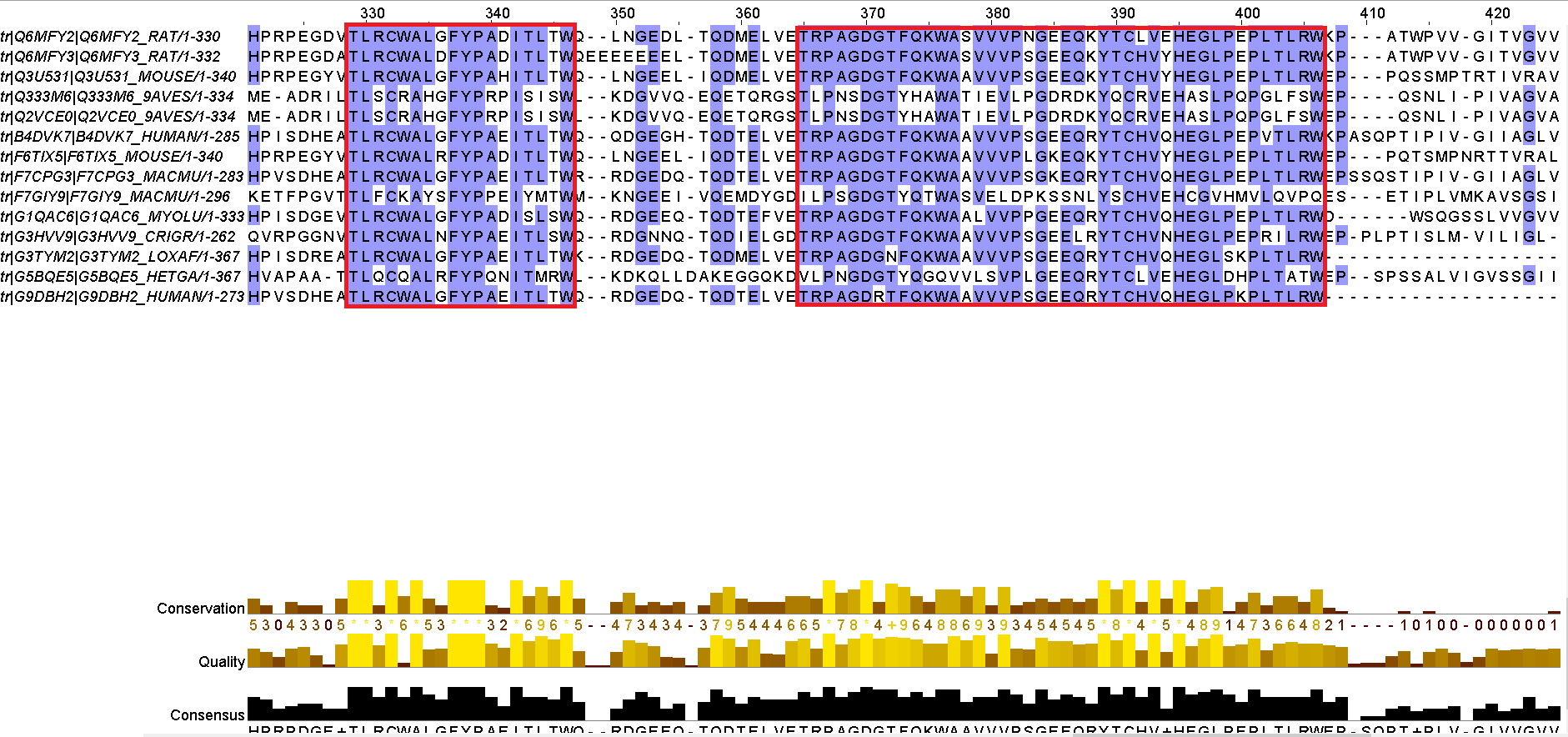
Рис.1. Общий вид проекта в Jalview, вертикальные блоки выравнивания показаны красными прямоугольниками. Как видно, в обоих блоках довольно высок процент абсолютно консервативных позиций (например, в блоке слева это позиции 329, 330, 332, 334, 337-339, 342, 346.
Также я сохранила каждый блок в формате .mfa: первый блок можно скачать здесь, второй блок можно скачать здесь.
Затем необходимо было найти участок, на котором гомологичны только 2 последовательности. Для этого, очевидно, нужно выбрать 2 наиболее близкие между собой последовательности. Сделать это можно, исходя из соображения, что 2 белка представителей одного таксона будут наиболее близки между собой в этом выравнивании. Так мне удаловь обнаружить участок из последовательностей белков птиц, которые явно гомологичны друг другу, и не гомологичны остальным последовательностям. Второй участок гомологии между теми двумя последовательностями был найден мной на участке с плохой консервативностью для всего множественного выравнивания. Видно, что блок очень протяжённый и последовательности на этом участке идентичны.

Рис.2. Блок выравнивания, на котором 2 последовательности белков птиц гомологичны друг другу, но негомологичны остальным последовательностям из множественного выравнивания. На этом рисунке показан участок с неплохой консервативностью по всему выравниванию.

Рис.3. Ещё один блок выравнивания, на котором 2 последовательности белков птиц гомологичны друг другу. Здесь показан участок с плохой гомологией других последовательностей множественного выравнивания.
Другим способом определить последовательности, у которых стоит искать более выраженную гомологию, является построение дерева. Это можно сделать в самом Jalview, поэтому я решила не пренебрегать данной возможностью. Дерево показало нам также, что две раннее сравниваемых последовательности птиц на самом деле - одна и та же последовательность, поэтому говорить о гомологии здесь как-то неуместно. Поищем 2 другие последовательности, которые удовлетворяют поставленной задаче.
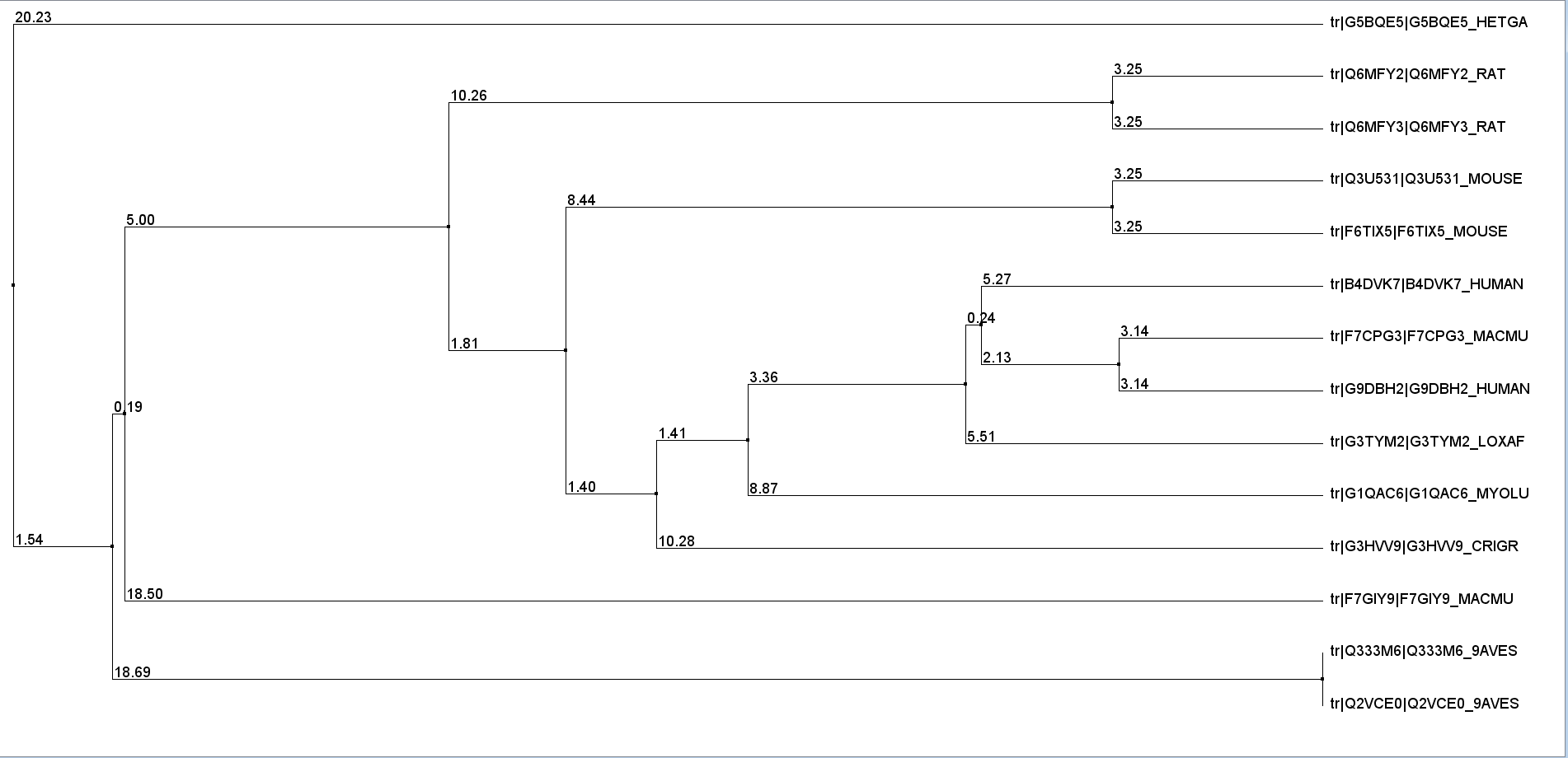
Рис.4. Дерево выравнивания, построенное в Jalview. Из него можно получить информацию о родственных связях белков. Например, наглядно видно, что последовательность белка голого землекопа (G5BQE5_HETGA)наименее гомологична всем остальным, поэтому на дереве она показана как бы одтельной ветвью.
Ориентируясь на данные дерева, я нашла 2 последовательности - B4DVK7_HUMAN и F7CPG3_MACMU. Они имеют участок, на котором их гомология очевидна, в то время как их сходство с другими последовательностями не настолько хорошее. Этот участок показан на рисунке 5.
Рис.5. В зелёных прямоугольниках показан участок выравнивания последовательностей B4DVK7_HUMAN и F7CPG3_MACMU, на котором очевидна их гомология.
Для выполнения второго задания, в котором надо посчитать процент консервативных позиций я выбрала второй блок, показанный на рисунке 1 (блок справа). Всего в нём 42 колонки.
Абсолютно консервативная позиция - это такая колонка, в которой во всех выравниваниях стоит одинаковая аминокислота. В этом блоке их 6, в процентном соотношении 14,28%.
Абсолютно функционально консервативная позиция - это такая колонка, в которой во всех выравниваниях стоят аминокислоты одной и той же группы. Таких колонок 12, их доля в процентах - 28,57%.
Консервативная на 70% позиция - это такая колонка, в которой в 70% выравниваемых последовательностей и более стоит одна и та же аминокислота. Таких колонок в блоке 36, что составляет 85,71% от всех колонок.
Функционально консервативных на 70% колонок в этом блоке 37, что составляет 88% от всех колонок блока.
Между двумя блоками на рисунке 1 находится 18 позиций, из которых 3 - это гэпы. Тогда процент гэпов в этой части выравнивания (блок 1 + блок 2 + расстояние между ними) - 3,85%.
В задании 4 необходимо было вручную выровнять последовательность относительно какого-либо из блоков множетвенного выравнивания. Я решила выравнивать относительно блока 2 с рисунка 1. Результат получился неплохой, как и ожидалось, ведь в описании добавленной последовательности сказано, что она относится к предполагаемому белку комплекса гистосовместимости домашней курицы (Gallus gallus). На рисунке 6 показано множественное выравнивание с добавленной последовательностью. Как видно, совпадений по абсолютно консервативным позициям и функционально консервативным позициям есть, из данного выравнивания можно сделать вывод о гомологии добавленной последовательности и остальных последовательностей на этом участке.
Рис. 6. Выравнивание новой последовательности относительно блока 2 из начального множественного выравнивания. Бывший блок 2 обведён красным прямоугольником.
В задании 5 необходимо было получить консенсусную последовательность всего выравнивания и отдельного блока. Консенсусная последовательность - это обобщённая последовательность, которая получена на основе сравнения букв в колонках выравнивания. Этот термин используется как для выравниваний последовательностей белков, так и нукленовых кислот. Консенсусная последовательность всего выравнивания была получена с помошью инструментов Jalview:
>Consensus/1-383 Percentage Identity Consensus ----MRMGVMVPGTLLLLLSGALALTQTKNQSWAGSHSLRYFYTAV-SRPGRGEPRFIAVGYVDDTQFVRFD SD--------------------GATQRMEPRAPWMEQEVGPEYWLRETQNA-EGGSHTFRENLGTDLGYYNQ SD-GE----------------------------------------------------G-G----G-----G- ---SHT-Q---GCEVGPD----RGFDQHAYDGKDYIALNEDLRSWTAADTAAQITKRKWEAAGTVAEQRRAY LEGTCVEWLRRYLENGKETLQRADPPKTHVTHHPRPDGEATLRCWALGFYPAEITLTWQ--RDGEEQ-TQDT ELVETRPAGDGTFQKWAAVVVPSGEEQRYTCHVEHEGLPEPLTLRWEP-SQPTIPLV-GIVVGVVLLGAVVI GALVGSFVVWRKKQAGGKGDRYLVAPGSDGEHLSLSAGSEPSALGVEAG-K-LP-APPLV-
Также данную последовательность можно скачать в формате fasta. Консенсусная последовательность блока 2 с рисунка 1:
>Consensus/1-42 Percentage Identity Consensus TRPAGDGTFQKWAAVVVPSGEEQRYTCHVEHEGLPEPLTLRW
Для построения LOGO для блока 2 мной был использован сервис WebLogo. LOGO - это наглядное графическое представление множественного выравнивания. Величина буквы в каждой позиции на рисунке говорит о консервативности аминокислоты в этом положении.

Рис. 7. LOGO для блока 2.
В последнем задании предлагается посмотреть, как выглядит выравнивание заведомо негомологичных белков. Для этого я взяла несколько белковых последовательностей, с которыми работали мои однокурсники. Их аминокислотные последовательности я "выравнивала" с помощью сервиса MUSCLE. Файл, который я получила в итоге, можно скачать здесь. Это и есть тот файл, который я подала на вход редактору JalView.
Как можно видеть на рисунках 8 и 9, мне не удалось найти хоть сколько нибудь удачный блок "выравнивания", по которому можно было бы судить о гомологии. В обоих блоках практически нет совпадающих аминокислот, а также много гэпов. По такому "выравниванию" сразу можно заключить, что белки негомологичны.
Рис. 8. Первый "блок" из выравнивания заведомо негомологичных белков.
Рис. 9. Второй "блок" из выравнивания заведомо негомологичных белков.
Проект данного "выравнивания" в формате .jar можно скачать здесь.