
ФББ 2013-2014

ФББ 2013-2014
Предсказание генов у прокариот
Открытая рамка считывания - это последовательность ДНК, начинающаяся со старт-кодона и не содержащая в себе стоп-кодона, которая возможно кодирует белок. В задании 1 требовалось найти все открытые рамки считывания заданного контига. Этот контиг взят из микробиома кардиального отдела желудка валлаби (маленькое австралийское сумчатое). То есть работа будет проводиться с бактериальным геномом. Для нахождения открытые рамки считывания с помощью программы ORF finder на сервере NCBI.
На вход программе подаётся последовательность в фаста-формате (или её идентификатор). Параметр, который можно задать - таблица генетического кода. Представлено целых 25 вариантов, т.к. у некоторых организмов может отличаться старт-кодон и другие особенности. По умолчанию стоит стандартная таблица, в которой начало трансляции допускается с AUG (стандартный), GUG, UUG. Также существует таблица ген. кода для бактерий, архей, пластид и вирусов прокариот. При её использовании трансляция допускается с AUG, GUG, UUG, CUG, AUU. Запустим Getorf два раза с разными таблицами. Получившиеся результаты можно видеть на рисунке 1. Как видно, отличия есть, но они незначительны.
Рис.1 Открытые рамки считывания, полученные с помощью программы GetOrf. Слева использовался стандартный ген. код, справа - ген. код для прокариотических организмов.
Из получившихся результатов выберем те, длина которых в нуклеотидах больше 180 пар оснований. Таких результатов 4, но возьмём мы только первые 3, т.к. четвертая находка по длине сильно меньше, чем другие три, а также она перекрывается с первой. Первая и третья находка сильно перекрываются, но на мой взгляд достовернее первая, т.к. она длиннее. Проверим это, запустив blastp. Выбранная база данных - swissprot.
Что-то похожее на гомологи было найдено для двух находок Getorf - первой и второй, то есть для самых длинных. Результаты представлены в таблице 1.
Таблица 1. Найденные открытые рамки считывания и их возможные белковые гомологи
| Начало | Конец | Длина | Цепь | Описание | Организм, из которого взят гомолог | E-value находки | Покрытие | Сходство |
| 140 | 1321 | 393 | - | АТФаза ААА | Paraprevotella clara, Paraprevotella xylaniphila, Bacteroides fluxus | 0.0 | 100% | 70% |
| 1723 | 2592 | 289 | - | Агматиновая деиминаза | Bacteroides coprosuis | 4e-113 | 97 | 58% |
Организмы, принадлежащие родам Bacteroides и Paraprevotella, входят в состав микробиома зверей, что логично. Это дополнительное доказательство того, что найденные гомологи достоверны.
GeneMark
Программа GeneMark также используется для предсказания генов. Она работает по эвристическому алгоритму. На вход даём тот же контиг, что и в первом задании. В итоге GeneMark предсказала два возможных гена, оба из которых находятся на обратной цепи. На выходе было получено 2 файла: PDF-файл с результатами и и файл с результатами в таблице. График кодирующего потенциала из PDF-файла можно увидеть на рисунке 2.
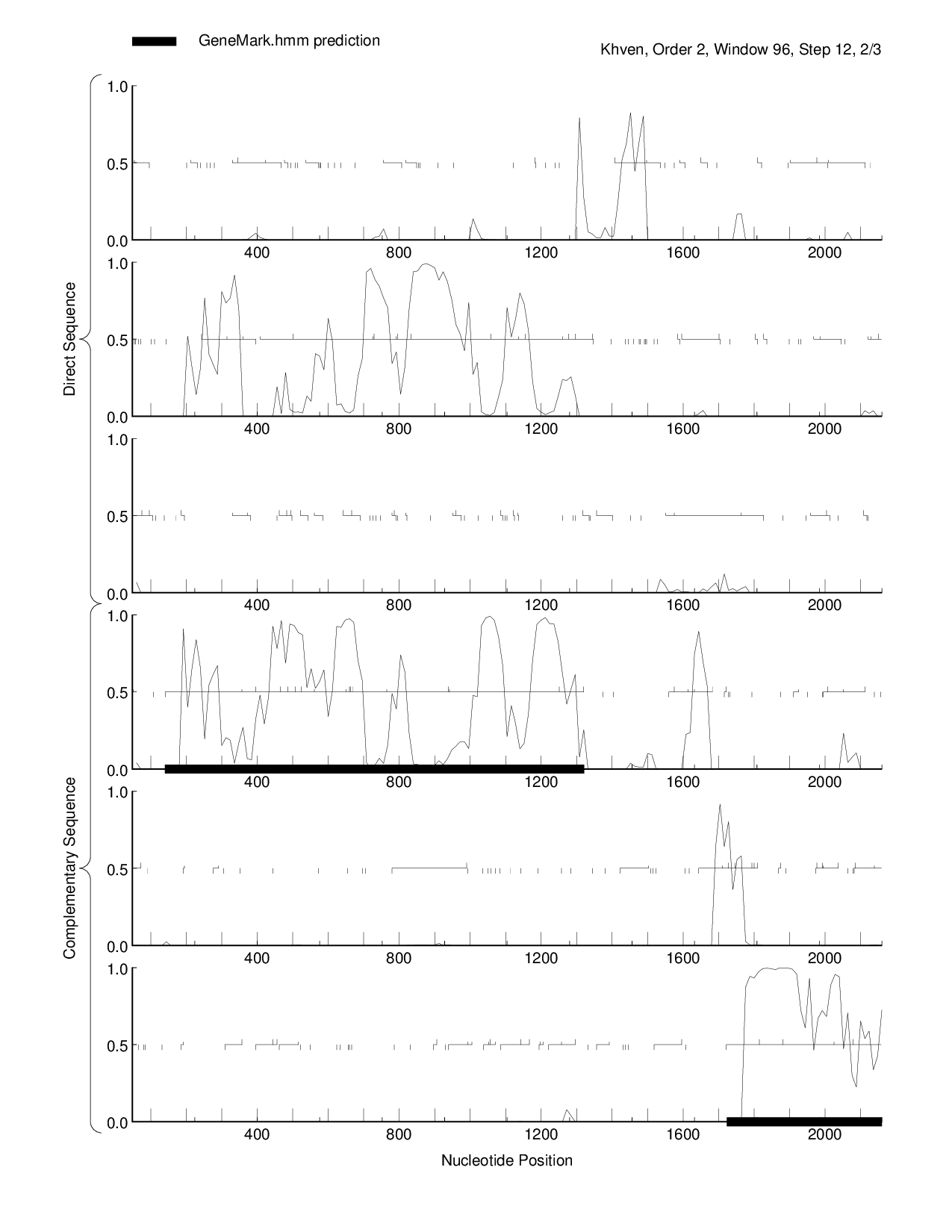

Рисунок 2 График кодирующего потенциала в данной последовательности. Жирные горизонтальные линии обозначают найденный ген.
В таблице 2 описаны найденные с помощью GeneMark гены.
Таблица 2 Характеристика генов, предсказанных с помощью программы GeneMark.
| Начало | Конец | Длина | Цепь |
| 140 | 1321 | 393 | - |
| 1723 | 2592 | 289 | - |
Сравнение предсказаний
Как можно видеть из таблиц 1 и 2, оба подхода смогли выявить 2 гена, которые с большой долей вероятности действительно существуют. В поиске с помощью GetOrf появляется больше побочных результатов, но, что понравилось лично мне, есть возможность выбрать таблицу генетического кода, в то время как в GeneMark нет такой тонкой настройки. В то же время, предсказание с помощью GetOrf + Blast более трудоёмкое и долгое, так как приходится вручную отбирать нужные результаты и бласт долго работает. Предсказанные с помощью GeneMark гены необходимо также необходимо было бы проверять с помощью поиска гомологов, если бы изначально использовалась только эта программа.
Запуск GeneMark с другими эвристическими параметрами
В 2010 году были разработаны другие эвристические параметры для работы с метагеномами. Выберем их для поиска генов в том же самом контиге. Полученные файлы: здесь и здесь. В результате были предсказаны те же два гена, что и ранее, но было и новое предсказание. Как видно из файла таблицы с результатами, программа посчитала, что с <3 до 74 пары оснований может быть также закодирован какой-то продукт.
Для того, чтобы разобраться, действительно ли там что-то закодировано, воспользуемся бластом по нуклеотидной последовательности и по белковой последовательности (просто трансляция с нуклеотидной). Несмотря на маленький размер, результат вполне может быть, если будем требовать очень большого совпадения (больше 95%) и очень маленького e-value. Используемы последовательности только что обнаруженного гена:
>gene_1|GeneMark.hmm|72_nt|-|3|74 >Khven ATGGCTAAGAAATTTATCACTTGTGATGGTAATACCGCTGCAGCACATATTGCTTATATG TTCTCAGAGGTA >gene_1|GeneMark.hmm|24_aa|-|3|74 >Khven MAKKFITCDGNTAAAHIAYMFSEV
Запуск бласта по нуклеотидам (blastn) выдал несколько полных геномов бактерий, в которых были похожие участки. Лучшее сходство - 91%, однако никакой полезной информации это не дало. Запустим бласт по белковой последовательности. Здесь обнаружился интересный, на мой взгляд, результат: самая лучшая находка имеет 96% совпадений, e-value 3e-06. Естественно, выровнялся только маленький кусок этого белка. Находка принадлежит симбиотической бактерии Prevotella sp. oral taxon 473, которая содержится в микробиоте человека и других млекопитающих, что может служить дополнительным аргументом за её достоверность. Найденный белок - пируват-синтетаза, возможно, что и в изучаемом контиге закодирован кусочек пируват-синтетазы.