
ФББ 2013-2014

ФББ 2013-2014
Предсказание генов у эукариот
Genscan
Genscan - это онлайн сервис, который позволяет установить экзон-интронную структуру последовательности ДНК. В этом задании работа будет проведена для фрагмента генома человека. Подадим эту последовательность на вход, на выходе получим список экзонов, для которых определены координаты и типы. Возможные типы экзонов: Init – начальный, Intr – внутренний, Term – конечный; PlyA – сайт полиаденилирования (не является экзоном), Prom - промотор (не является экзоном).
Сначала поставим параметр "Suboptimal exon cutoff" на максимальное значени (1.0). Это означает, что программа будет искать максимально вероятные экзоны. Результаты работы программы представлены в таблице 1.
Таблица 1. Экзоны, найденные с помощью Genscan.
| Начало | Конец | Цепь | Тип |
| 2497 | 2388 | - | Внутренний |
| 3948 | 3769 | - | Внутренний |
| 13254 | 13107 | - | Внутренний |
| 14575 | 14486 | - | Внутренний |
| 15571 | 15395 | - | Внутренний |
| 16403 | 16369 | - | Внутренний |
| 26891 | 26606 | - | Внутренний |
| 32696 | 32565 | - | Внутренний |
В таблице координаты конца меньше координат начала, потому что экзоны расположены на - цепи. Помимо экзонов Genscan предсказал возможный белковый продукт. В моём куске генома был предсказан белок каспаза-9 (протеаза, расщепляющая белки после аспарагиновой кислоты), которая закодирована на первой хромосоме и играет важную роль в регуляции апоптоза.
Также можно изменить параметр "Suboptimal exons сutoff", уменьшить его. Это делается для того, чтобы найти больше возможных экзонов, которые менее вероятны в строгой математической модели, но возможны в жизни. Снизив значение этого параметра до 0.5, на выходе получим ещё и список т.н. "неоптимальных" экзонов.
Genome browser
Воспользуемся геномным браузером, чтобы просмотреть информацию, относящуюся к данному куску ДНК человека. Для этого запустим BLAT для выданной последовательности с параметрами Assembly - Dec.2013 (RGCh38/hg38), Query type - DNA. На выходе получается довольно много результатов, самые лучшие из них представлены на рисунке 1.
Рисунок 1. Результаты поиска BLAT по заданной последовательности ДНК человека
Очевидно, что стоит взять находку №1, т.к. она полностью покрывает поданную на вход последовательность, имеет 100% сходства. Ещё в первом задании стало понятно, что скорее всего это фрагмент первой хромосомы, что подтверждается этой находкой.
Откроем находку в геномном браузере, попросим показать мРНК и ESTs (это короткие фрагменты РНК, прочитанные однократно). Чтобы найти интроны и различные типы экзонов ESTs сравниваются между собой и накладываются на геном. Получившееся окно геномного браузера можно увидеть на рисунке 2.

Рис.2.Исследуемая часть ДНК в геномном браузере.
В данной последовательности были найдены мРНК различных каспаз-9. Основная запись - CU675417, также показаны вариации, выделенные из различных органов (например, из мозга, Т-лимфоцитов(U60521), раковых клеток печени (AB015653)). Видимо, различные виды каспаз-9 получаются путём альтернативного сплайсинга с одного и того же гена. Типы экзонов - кассетные (т.е. могут как включаться, так и не включаться). Количество экзонов - 8 (всего, это лучше видно на рисунке 3), интронов - 8. Каспаза-9-бета является урезанным вариантом каспазы-9, у неё вырезаны экзоны 3-6. Известно, что каспаза-9-бета препятствует образованию полной формы каспазы-9, ингибируя активность специального белка, ответственного за образование полной каспазы-9 из прокаспазы. Также была найдены каспаза-9-гамма, которая содержит только один домен из полной каспазы-9. Это вызвано тем, что при её образовании происходит альтернативный сплайсинг четырёх экзонов, во время которого происходит инсерция последовательности, содержащей стоп кодон. В итоге трансляция с такой мРНК обрывается рано. Известно, что каспаза-9-гамма не может функционировать как полная каспаза-9, однако она способна ингибировать её активность по тому же механизму, что и каспаза-9-бета. Различные типы каспазы-9, получаемые в результате альтернативного сплайсинга, называются изоформами. Сейчас известно, что неправильный сплайсинг играет важную роль в патофизиологии рака.
В данной части генома были найдены некоторые регуляторные элементы на интронах: сайт посадки репрессора транскрипции, сайт посадки транскрипционного фактора, несколько энхансеров, промотор и участок с открытым хроматином.
Также для визуализации участка я использовала ENSEML - ещё один геномный браузер (я нахожу его более симпатичным). На рисунке 3 в красном прямоугольнике - расположение поданной на вход последовательности на хромосоме, также показано её окружение (слева ген эластазы, справа - ген шаперона DnaJ).
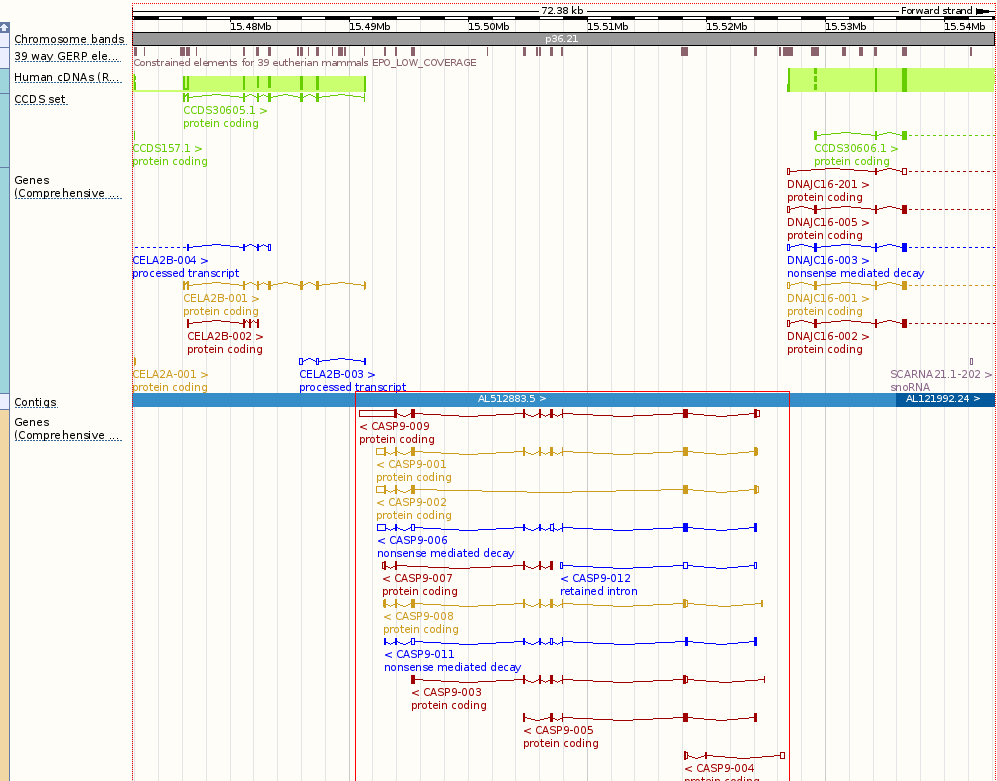
Рис.3.Исследуемая часть ДНК в геномном браузере (ENSEMBL).
Аннотирование генома с помощью BLASTX
Дана последовательность нуклеотидов из генома Actinidia chinensis, задача - проаннотировать его с помощью BlastX - это такой вид бласта, который использует геномную ДНК, транслирует её и ищет по белковым базам данных. Фрагмент, выданный мне - здесь.
Запустим BlastX по базе данных SwissProt, поиск ограничен белками растений. Было найдено 206 находок для разных мест поданной на вход последовательности. На рисунке 4 показано распределение находок по данной на вход последовательности. Видно, что есть три участка, где возможно есть гены. Участок №1 выровнялся с белками-ингибиторами диссоциации ГДФ (макс. сходство - 55%, e-value - 3e-51) и с YCF2 protein - АТФазами из стромы хлоропластов (макс.сходство - 73%, e-value - 3e-08). Второй участок выравнивается с элонгационными факторами: EF-1-alpha (сходство 97%, e-value - 2e-175), EF-Tu - хлоропластный (сходство - 37%, e-value - 3e-10). Третий участок - с рецептором ядерной оболочки, ответственный за сохранение белков на ней (сходство 83%, e-value - 5e-37).
Рисунок 4. Результаты поиска BLASTX по заданному фрагменту генома киви.
Поскольку заранее дано, что это эукариотический геном, все хлоропластные гены не рассматриваем. Также есть два участка, расположенные между первым и вторым, для которых тоже прошло выравнивание с какими-то белками, но они сомнительные из-за не очень высокого процента сходства и отсутствия гомологов. Их также не берём в расчёт.
Теперь определим экзоны на фрагменте из генома киви. Наиболее трудная ситуация - с первым геном. Для того, чтобы определить экзоны, будем смотреть не только на выравнивание с наибольшим весом, но и на другие выравнивания, т.к. структура участка, с которым выровнялся ген ингибитора участка ГДФ, содержит как бы 2 повторяющихся гена. Также будем опираться на экзон-интронную структуру гена того же белка из Arabidopsis thaliana (рисунок 5), т.к. ожидается, что она должна примерно совпадать по количеству экзонов.
Рисунок 5. Экзон-интронная структура гена ингибитора диссоциации ГДФ из Arabidopsis thaliana. Розовые прямоугольники - это экзоны, пики между ними - интроны.
Исходя из выравнивания и экзон-интронной структуры гомологичного гена, данный участок из генома киви я разделила на 14 экзонных участков. Стоит упомянуть, что экзоны 1-7, 2-8 и т.д. совпадают.
С геном элонгационного фактора 1 альфа будет проще, поскольку он обладает большой консервативностью и содержит всего лишь 1 интрон. Последний ген расположен на обратной цепи и содержит 7 экзонов. Информация о трёх этих генах собрана в таблице 1.
Таблица 1. Экзоны 3х предсказанных генов из фрагмета генома киви.
| № экзона | Координаты начала | Координаты конца | Цепь |
| Предсказанный ингибитор диссоциации ГДФ | |||
| 1 | 11507 | 11605 | + |
| 2 | 12318 | 12359 | + |
| 3 | 12640 | 12725 | + |
| 4 | 14416 | 14508 | + |
| 5 | 14597 | 14893 | + |
| 6 | 20009 | 20146 | + |
| 7 | 21066 | 21439 | + |
| 8 | 23467 | 23565 | + |
| 9 | 26563 | 26603 | + |
| 10 | 26705 | 26970 | + |
| 11 | 30398 | 30481 | + |
| 12 | 30582 | 30860 | + |
| 13 | 31372 | 31509 | + |
| 14 | 32362 | 32730 | + |
| Предсказанный элонгационный фактор 1 альфа | |||
| 1 | 72609 | 73072 | + |
| 2 | 73819 | 74666 | + |
| Предсказанный рецептор ядерной оболочки | |||
| 1 | 104098 | 103943 | - |
| 2 | 100422 | 100255 | - |
| 3 | 100158 | 100146 | - |
| 4 | 98544 | 98664 | - |
| 5 | 94568 | 94545 | - |
| 6 | 94319 | 94306 | - |