
2013-2014

2013-2014
Трансмембранные белки
Ромбоидные протеазы - класс трансмембранных белков, найденный почти у всех известных организмов. Этот белок был первоначально обнаружен у дрозофилы в результате геномного скрининга. Ромбоидные протеазы имеют одну интересную особенность - их активный сайт расположен внутри трансмембранных доменов, а главная функция этих белков - разрезать другие трансмембранные белки. У эукариот ромбоидные протеазы присутствуют в митохондриях, где они опосредуют слияние митохондриальных мембран, а далёкие гомологи участвуют в межклеточной сигнализации. У высших эукариот ромбоидные протеазы, как правило, не активны.

Рис.1. Филогенетическое дерево ромбоидных белков. Картинка взята из статьи. Не закрашенные клады имеют в своём геноме гены ромбоидных белков, однако они не активны.
Известно, что ромбоидные протеазы относятся к классу сериновых протеаз. Их отличие от других сериновых протеаз в том, что каталитическая активность опосредована не тремя аминокислотами (аспартат-серин-гистидин), а только двумя, что было показано в структурных работах. Возможно, это связано с тем, что аспартат слишком сильно заряжен, чтобы находиться внутри мембраны. На рисунке 2 представлены 3 принципиальные структуры каталитически активных ромбоидных протеаз. Как видно из рисунка, все они имеют 6 трансмембранных спиралей, но у эукариотических форм имеются дополнительные домены на N-конце или C-конце.

Рис.2. Схема расположения трансмембранных спиралей у ромбоидных протеаз. Картинка взята из статьи.
Мне была дана последовательность ромбоидной протеазы из E.coli. По ней, с помощью средств BLAST и STRING, была составлена репрезентативная выборка из 20ти белков, часть из которых - митохондриальные ромбоидные протеазы эукариот. Необходимость воспользоваться базой данных STRING была обусловлена тем, что бласт выдавал слишком много хитов из той же E.coli, даже при исключении её таксона из поиска.
Затем было составлено множественное выравнивание отобранных последовательностей с помощью программы MUSCLE. Вид полученного выравнивания представлен на рисунке 3.
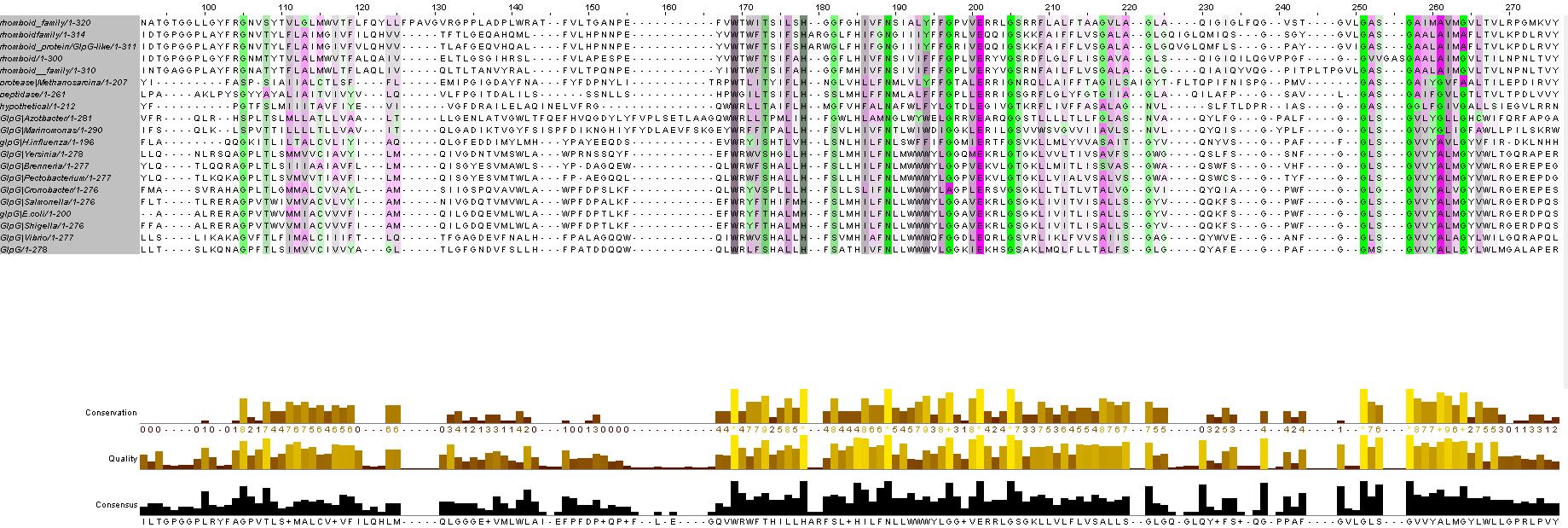
Рис.3. Множественное выравнивание отобранных последовательностей. Покраска в Jalview - Helix propensity, по консервативности (порог 30%).
Далее к последовательности E.coli была привязана пространственная структура из PDB банка. Была сделана разметка трансмембранных спиралей по этой PDB структуре (с помощью также сервиса OPM), она обозначена как TM_real. Проект Jalview можно скачать здесь. Получившиеся трансмембранные участки в большинстве случаев совпадали с самыми консервативными блоками выравнивания. Среди консервативных остатков в выравнивании нет гидрофильных аминокислот, в основном это гидрофобные аминокислоты. Однако в реальной структуре в трансмембранных участках есть заряженные остатки, что объясняется расположением активного центра этого белка внутри мембраны.

Рис.4. Общий вид проекта Jalview с разметкой трансмембранных участков. Покраска Hydrophobicity, по консервативности (порог 30%).
Далее была запущена программа TMHMM, способная предсказывать трансмембранные участки поданных ей на вход последовательностей. На рисунке 5 представлен TMHMM профиль для ромбоидного белка из E.coli. По нему была сделана разметка TM_predicted. Предсказания этого сервиса не всегда соответствуют реальной картине, его худшее и лучшее предсказание показано на рисунке 5 (вырезки из проекта Jalview). Создалось впечатление, что TMHMM в своём поиске руководствуется только наличием большого количества гидрофобных аминокислот рядом. Однако стоит отметить, что в целом число предсказанных трансмембранных участков и их примерное расположение указаны верно.


Рис.5. Лучшее и худшее предсказание сервиса TMHMM для ромбоидной протеазы E.coli.
На рисунке 6 представлен TMHMM профиль ромбоидной протеазы E.coli, красные пики обозначают предсказанные трансмембранные участки.
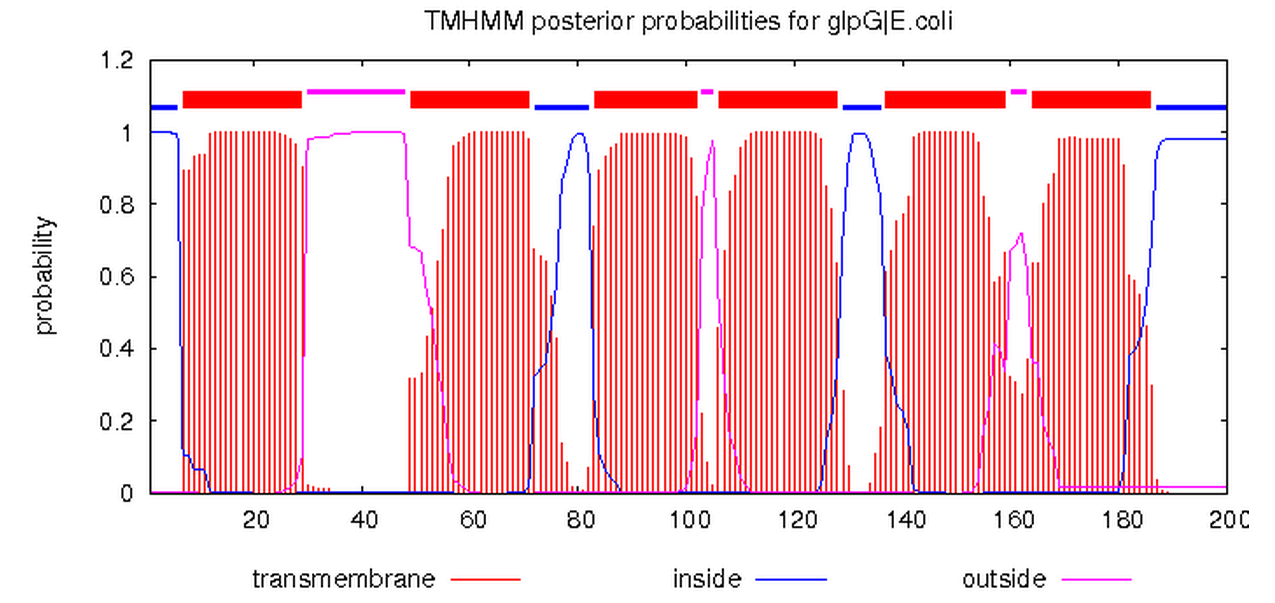
Рис.6. Предсказание трансмембранных участков с помощью TMHMM.
Предсказания TMHMM были получены для всех белков из выборки. Ожидаемо, наиболее отклоняющимися от E.coli результатами стали митохондриальные белки эукариот. Предсказание для одного из таких белков показано на рисунке 7.

Рис.7. Предсказание трансмембранных участков с помощью TMHMM для митохондриального белка.