
2013-2014

2013-2014
Реконструкция филогении по 16s рРНК
16s рРНК входит в состав малой субъединицы рибосом прокариот, выполняя важную роль в трансляции: в ней содержится последовательность, комплементарная последовательности Шайна-Дальгарно. Она участвует в посадке рибосом на мРНК. По последовательностям 16s рРНК часто проводят филогенетический анализ, поскольку они высоко консервативны.
Для отобранных ранее бактерий (STAA1, STAES, ENTFA, CLOTE, BACSU, LACLM, LACDA, STRP1) были скачаны последовательности 16s рРНК, хранящиеся на сервере NCBI в файлах с расширением .frn. Далее было построено их выравнивание (файл). Затем с помощью MEGA было построено дерево методом Neighbourhood joining, результат представлен на рисунке 1.
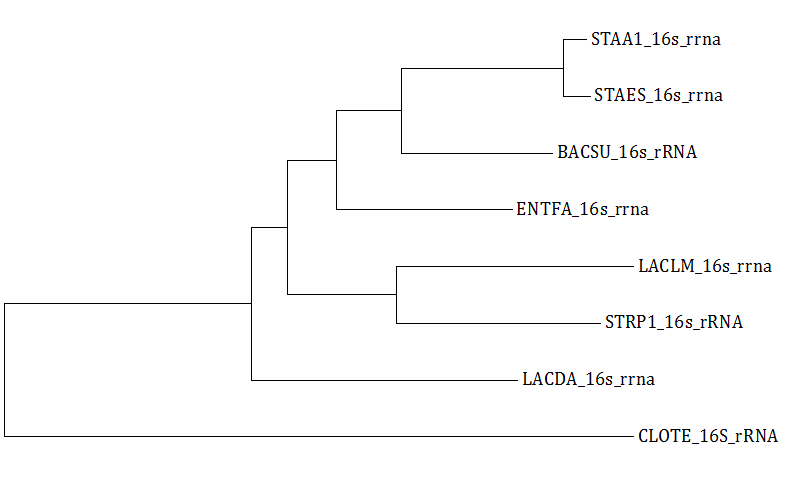
Рис.1. Дерево, построенное методом Neighbourhood joining в MEGA по выравниванию последовательностей 16s рРНК отобранных бактерий.
Данное дерево не похоже на эталонное (приведено на рис.2). ENTFA объединено в одну кладу с STAA1, STAES и BACSU, а не с LACLM и STRP1 (то есть появилась нетривиальная ветвь {STAA1, STAES, BACSU, ENTFA} vs {STRP1, LACDA, LACLM, CLOTE}). LACDA не объединена с LACLM и STRP1 (исчезла нетривиальная ветвь {LACLM, ENTFA, STRP1, LACDA} с оригинального дерева).
Рис.2. Эталонное дерево.
Вероятно, расхождения с правильной филогенией возникли из-за случайного выбора штаммов в момент скачивания последовательностей с NCBI.
Паралоги и ортологи
Паралоги - это гомологичные белки, принадлежащие одному организму. Они возникают в результате дупликации гена и могут разойтись в процессе эволюции настолько, что начнут выполнять разные функции. Ортологи - это гомологичные белки из разных организмов, разошедшиеся в процессе видообразования и выполняющие одну и ту же функцию (чаще всего).
Белок CLPX - это АТФ-связывающий домен бактериальной протеазы Clp, который использует энергию гидролиза АТФ для разворачивания белка и его транслокации в разрезающий домен. Была взята последовательность этого белка из BACSU - CLPX_BACSU. Затем был осуществлён поиск гомологов этого белка в ранее отобранных бактериях с помощью BLAST на сервере NCBI, однако нашлось слишком много одинаковых последовательностей под разными названиями и далёкие гомологи не удалось найти даже с помощью пси-бласта.
Протеомы отобранных бактерий были скачаны с kodomo и объединены в 1 файл. Из них была сделана база типа prot для дальнейшего BLAST. Затем был запущен Standalone BLAST командой blastp -query CLPX_BACSU.fasta -db strp1.fasta -evalue 0.001 -outfmt 7 -word_size 3 -out table. Из полученной таблицы были извлечены ID найденных белков, их последовательности были найдены в Uniprot, скачаны и выровнены с помощью MUSCLE (выравнивание).
В MEGA с помощью алгоритма Neighbour Joining было получено дерево, приведённое на рисунке 3.
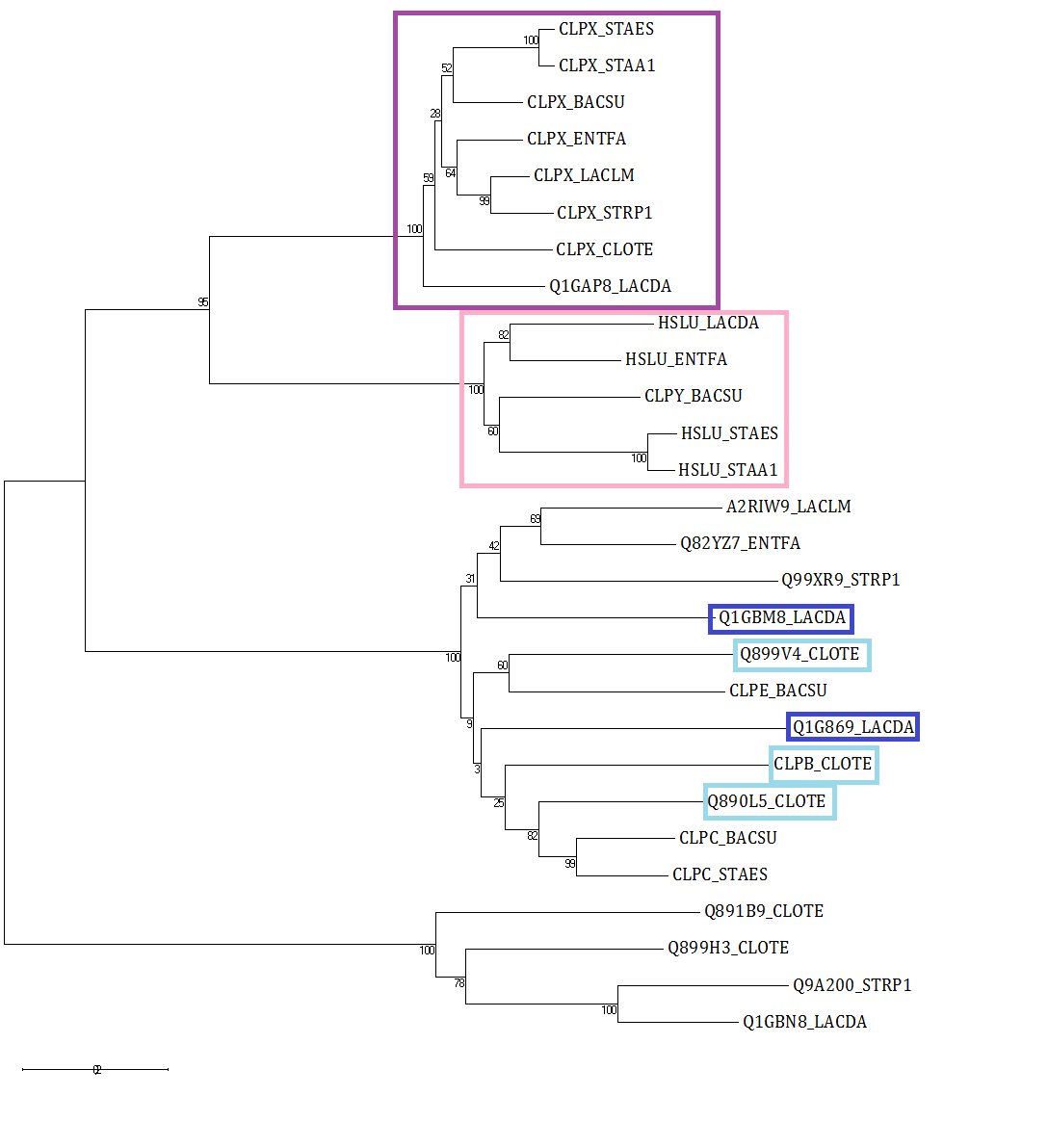
Рис.3. Полученное дерево
На дереве фиолетовым и розовым отмечены две группы ортологов. Первая группа - белки CLPX из всех 8ми бактерий, вторая - HSLY (ClpY - второе название). HSLY - это АТФ-зависимый шаперон, входящий в состав той же протеазы. Голубым и синим показаны две группы паралогов.