Мини-обзор генома Moraxella ovis
Петренко Павел
Факультет биоинженерии и биоинформатики, Московский Государственный Университет имени М.В.Ломоносова
1. Введение
Moraxella ovis - это грамотрицательная бактерия, выделенная из случаев конъюнктивита овец (и других животных-представителей крупного рогатого скота (IBK)). Данная бактерия относится к филуму Pseudomonadota (таблица 1) и имеет множество близких видов, также вызывающих патогенные заболевания IBK [1].
| Superkingdom | Bacteria (eubacteria) |
|---|---|
| Phylum | Pseudomonadota |
| Class | Gammaproteobacteria |
| Order | Moraxellales |
| Family | Moraxellaceae |
| Genus | Moraxella |
| Species | Moraxella ovis |
Moraxella ovis (как и близкие к ней виды Moraxella bovis, Moraxella bovoculi) способны вызывать патогенез (инфекционный кератоконъюнктивит) IBK. Однако за последний год удалось создать антисыворотку на основе данных штаммов, которая показала хорошие результаты в борьбе против родственных штаммов M.ovis, M.bovis и M.bovoculi. Так, было доказано, что Moraxella ssp. обладает множеством имунногенных антигенов, что может помочь в разработке поливалентной вакцины, обеспечивающей сильную и широкую защиту от всех штаммов, участвующих во вспышках IBK [3].
2. Материалы и методы
Данные по геному исследуемой бактерии были взяты с сайта Национального Центра Биотехнологической информации (NCBI). Для расчета нуклеотидного состава использовался язык программирования Python и сервис Google collab. Для расчета параметра GC-skew использовался сервис Webskew. Для анализа данных использовались электронные таблицы Google Sheets.
3. Результаты
3.1 Данные о геноме Moraxella ovis
Геном бактерии Moraxella ovis состоит из хромосомы длиной 2265515 пар нуклеотидов и плазмиды длиной 40582 нуклеотида (таблица 2). Благодаря вычисленному GC составу хромосомы и плазмиды бактерии, можно сделать вывод о температуре роста Moraxella ovis [4]. Данная бактерия не является термофильной, так как в составе её генома не много GC пар. Данная теория подтверждается тем, что Moraxella ovis обитает в организме животных (крупного рогатого скота), соответственно оптимальной температурой для неё является 37°C.
| Репликон | Длина, п.н. | РGC состав |
|---|---|---|
| Хромосома | 2265515 | 45.4% |
| Плазмида | 40582 | 39.8% |
Более подробный анализ нуклеотидного состава Moraxella ovis показал, что для генома бактерии выполняется правило Чаргаффа (таблица 3). У плазмиды будет большая статистическая погрешность ввиду маленького размера.
| Репликон | A | T | G | C |
|---|---|---|---|---|
| Хромосома | 618284 (27.3%) | 619676 (27.4%) | 517017 (22.8%) | 510538 (22.5%) |
| Плазмида | 12021 (29.6%) | 12403 (30.6%) | 7747 (19.1%) | 8411 (20.7%) |
3.2 Длины белков, закодированных в геноме бактерии Moraxella ovis
Для протеома бактерии было определено распределение белков по длине, составлена гистограмма (Рисунок 1), а также подсчитаны некоторые статистические показатели (Таблица 2). Больше всего белков с длиной в пределах 50-350 а.о., что соответствует, скорее всего, несложным одно- или двудоменным белкам. Длина самого короткого белка составляет 29 а.о.(NZ_CP011158.1_cds_WP_256594033.1_1323), в то время как самый длинный белок имеет длину 4430 а.о. (type III pantothenate kinase).
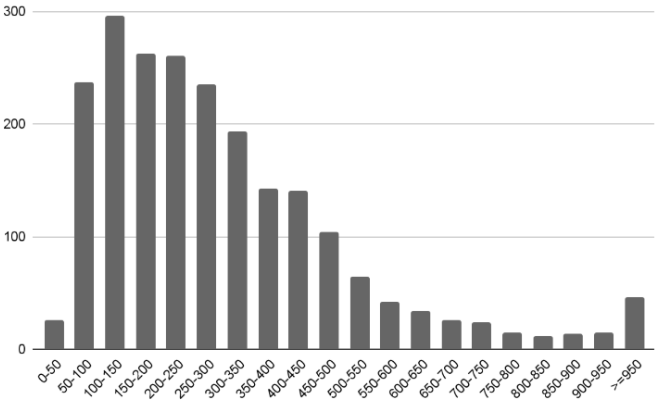
| Минимальная длина белка | 29 |
|---|---|
| Максимальная длина белка | 4430 |
| Средняя длина белка | 306,8 |
| Медиана | 253 |
3.3 Длина (в нуклеотидах) между кодирующими последовательностями (CDS)
Для генома бактерии была построена гистограмма расстояний между последовательными кодирующими последовательностями (CDS) на хромосоме (Рис.2). Расстояние, равное нулю соответствует расположенным вплотную генам, расстояние, меньшее нуля - пересекающимся генам. Было выявлено наличие довольно большого числа пересекающихся или расположенных на небольшом расстоянии друг от друга кодирующих последовательностей. Это свидетельствует об оперонной организации генома, характерной для прокариот [5].
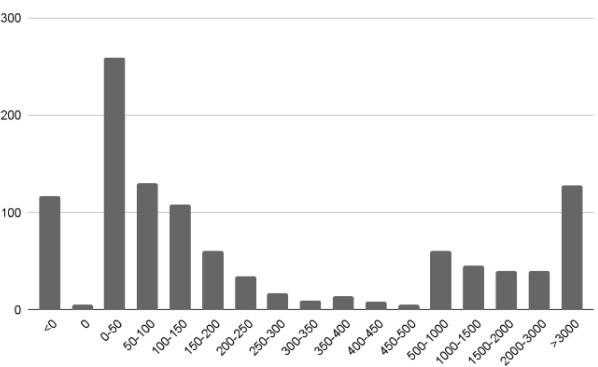
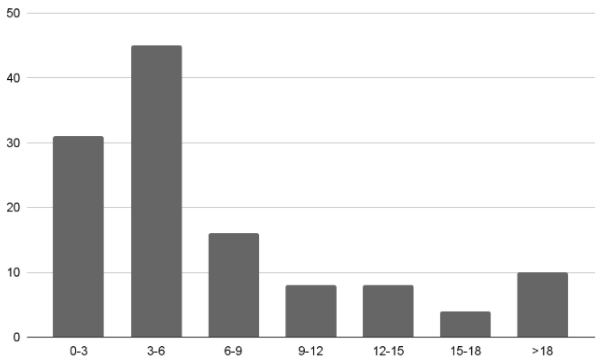
При более подробном рассмотрении длин пересечений CDS видно, что большая часть пересечений составляет от 0 до 6 нуклеотидов, что как раз характерно для генома бактерий из-за его быстрого развития [6].
3.4 Предсказание точки начала (oriC) и терминации (ter) репликации
При вычислении соотношения между нуклеотидами G и C (GC skew) было вычислено место начала репликации (origin, далее oriC) и её конца (terminator, далее ter). Место минимального значения GC skew соответствует oriC, а место максимального значения GC skew - ter. Действительно, малая доля GC пар позволяет упростить расплетение молекулы ДНК, что обеспечивает формирование репликационных вилок. Высокий состав же будет способствовать терминации [7].
При расчёте GC skew на определённом участке генома берётся интеграл от полученных значений ,рассчитанных по формуле: GC skew = (C - G)/(C + G), что образует линию графика, минимум и максимум которого соответствует расположению oriC и ter.
Так,у бактерии Moraxella ovis положение oriC находится в окрестности нулевого нуклеотида, а положение ter определено в окрестности 1127970 нуклеотида.
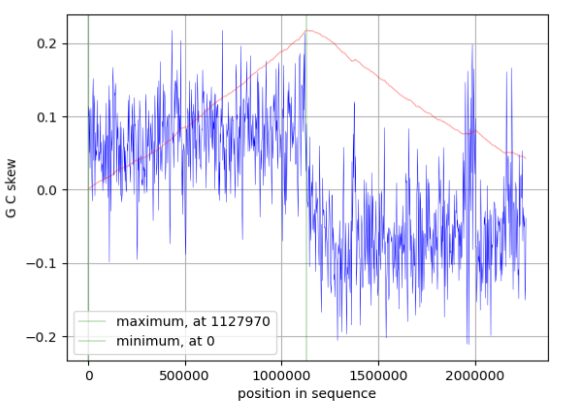
| Хромосома | Положение oriC | Положение ter |
|---|---|---|
| Окрестность нуклеотида | 0 | 1127970 |
3.5 Распределение белок-кодирующих последовательностей между + и - цепями
Исходя из полученных диаграмм (рис.4 и рис.5), видно, что гены на прямой и обратной цепочках хромосомы распределены относительно равномерно, а на плазмиде большая часть генов находится на - цепочке, что подтверждает асимметричную структуру плазмиды.
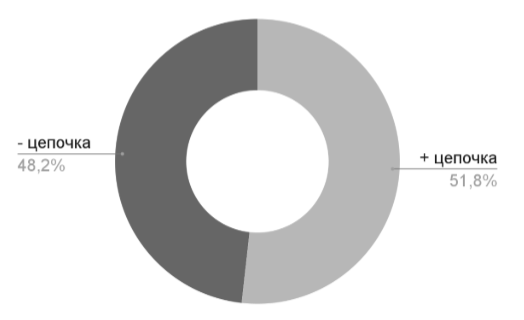
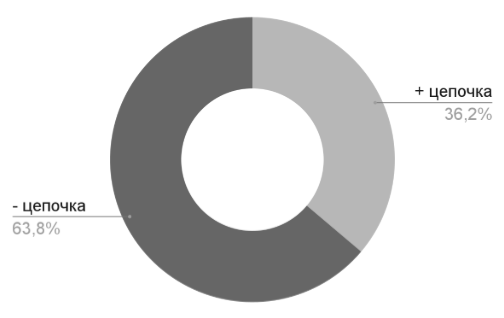
3.6 Расчёт независимого распределения генов на + и - цепочках Moraxella ovis
Для оценки случайности распределения генов на + и - цепях хромосомы и плазмиды Moraxella ovis был рассчитан критерий Пирсона (хи-квадрат). Используемый уровень значимости - 0.05. Предположим, что гены распределены на цепочках независимо. Предельный уровень значимости при наличии одной степени свободы равен 3.84. Так, расчёты показали, что значение хи-квадрата для хромосомы и плазмиды меньше предельного уровня значимости, что свидетельствует о случайном распределении генов.
Исходя из результатов исследования 3.5 и 3.6 видно, что гены на плазмиде распределены неравномерно и случайно. Это позволяет сделать вывод о том, что плазмида - это структура бактериальной клетки, подвергающаяся наиболее сильному эволюционному давлению. То есть некоторые гены могут обеспечивать выживаемость или адаптацию бактерии, что влияет на их распределение по плазмиде. В процессе эволюции происходит рекомбинация генов (гены перемещаются внутри плазмиды), что приводит к их случайному расположению, а также на расположение генов в плазмиде могут оказывать влияние различные вещества, находящиеся в среде обитания бактерии, или элементарные вставки - встраивание мобильных генетических элементов, таких как транспозоны, в плазмиду.
| Количество генов (ожидаемое) | Количество генов (наблюдаемое) | хи-квадрат | |
|---|---|---|---|
| Хромосома | 2,623 | ||
| + цепочка | 1044 | 1081 | |
| - цепочка | 1044 | 1007 | |
| Плазмида | 3,596 | ||
| + цепочка | 23,5 | 17 | |
| - цепочка | 23,5 | 30 |
Заключение
Таким образом, был исследован геном бактерии Moraxella ovis. Получена гистограмма длин белков и выявлены длины между кодирующими последовательностями на + цепи хромосомы бактерии. К тому же были предсказаны точки начала (oriC) и терминации (ter) репликации по параметру GC skew, а также было изучено распределение белок-кодирующих последовательностей и независимое распределение генов на + и - цепочках Moraxella ovis.
4. Сопроводительные материалы
- Google sheet
- Cкрипты, написанные на языке питон, использующиеся для изучения генома Moraxella ovis
- Сервис для расчёта GS-skew
- Данные NCBI по геному Moraxella ovis
5. Литература
- [1] Kosala S Ravikumaran, Rebecca M King, Anna Notaro, Antonio Molinaro, Cristina de Castro, Jennifer C Wilson, I Darren Grice, Ian R Peak. Moraxella ovis and Moraxella bovoculi lipooligosaccharide biosynthesis genes, and structural characterisation of oligosaccharide from M. ovis 354T. Carbohydrate Research, Volume 536, February 2024, 109043
- [2] Taxonomy Browzer [NCBI]: Moraxella ovis Taxonomy / Schoch CL, et al, Database (Oxford), 2020
- [3] Ananda Paula Kowalski, Grazieli Maboni, Letícia Trevisan Gressler, Julia Pires Espíndola, Cláudia Balzan, Caiane Tasca, João Antônio Guizzo, Fabricio Rochedo Conceição, Rafael Frandoloso, Agueda Castagna de Vargas. Antigenic characterization of Moraxella bovis, Moraxella bovoculi and Moraxella ovis strains with potential use in vaccines. Veterinary Microbiology, Volume 210, October 2017, Pages 56-63
- [4] En-Ze Hu, Xin-Ran Lan, Zhi-Ling Liu, Jie Gao & Deng-Ke Niu. A positive correlation between GC content and growth temperature in prokaryotes. BMC Genomics, volume 23, Article number: 110 (2022)
- [5] Zackary I Johnson, Sallie W Chisholm. Properties of overlapping genes are conserved across microbial genomes. Genome Res. 2004 Nov;14(11):2268–2272
- [6] Miguel M Fonseca, D James Harris, David Posada. Origin and Length Distribution of Unidirectional Prokaryotic Overlapping Genes. G3 Genes|Genomes|Genetics, Volume 4, Issue 1, 1 January 2014, Pages 19–27
- [7] A Grigoriev. Analyzing genomes with cumulative skew diagrams. Nucleic Acids Research, Volume 26, Issue 10, 1 May 1998, Pages 2286–2290