Нуклеотидный BLAST
Петренко Павел
Факультет биоинженерии и биоинформатики, Московский Государственный Университет имени М.В.Ломоносова
1. Ищем в геноме эукариота ген, кодирующий δ-субъединицу АТФ-синтазы
В предыдущем практикуме я работал с Caenorhabditis elegans, поэтому сейчас поищем в его геноме δ-субъединицу АТФ-синтазы. Поисковый запрос на NCBI:
Страница с найденной субъединицей
Сохраним последовательность этого белка в формате FASTA: δ-субъединицf АТФ-синтазы.
Идентификатор нуклеотидной записи, к которой относится ген, кодирующий данный белок: NP_495286
Идентификатор второй хромосомы, на которой закодирован белок (нужен для поиска участка ДНК с геном, кодирующим белок): NC_003280.
Локализация гена: 6.383.645-6.384.692
Последовательность ДНК, непосредственно содержащая кодирующую белок часть гена δ-субъединицы АТФ-синтазы: NC_003280.10
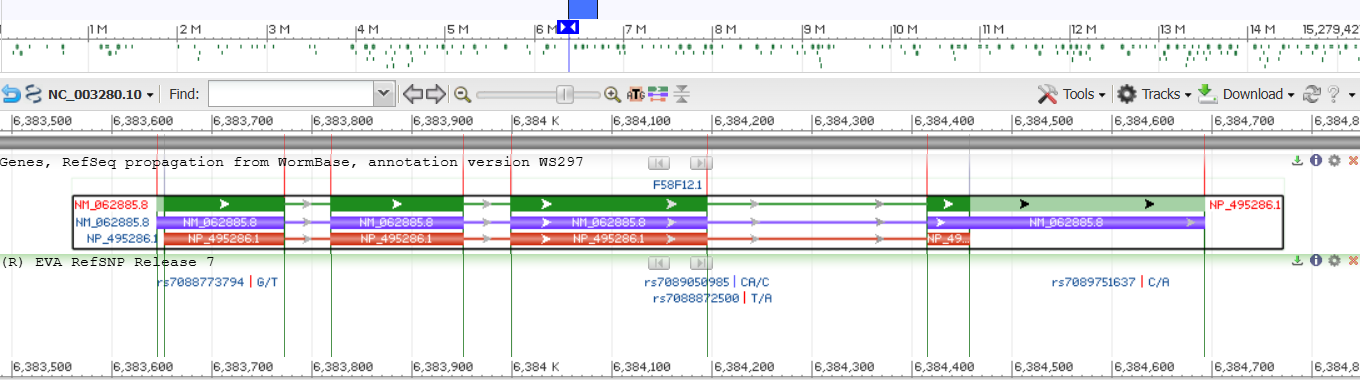
Верхнее поле - карта всей хромосомы, зелёные участки - гены на ДНК (ярким цветом обозначены интроны и экзоны, светло-зелёным обозначена нетранслируемая область), синие участки - гены на мРНК, красные участки - сам белок, стрелочки вправо указывают на то, что у нас (+)цепь, снизу указаны образованные неканонические пары нуклеотидов (как я понял).
2. Пробуем разные варианты BLAST для фрагмента ДНК
Следущей задачей стало найти в относительно отдаленном таксоне последовательности, гомологичные гену δ-субъединицы АТФ-синтазы из Caenorhabditis elegans. Так как круглые черви относятся к первичноротым, в качестве таксона для поиска я взял семейство Canidae (это клада собак, к которым относятся домашние собаки, волки, койоты, енотовидные собаки, лисы, шакалы и некоторые другие виды.). База данных, которая использовалась - refseq_genomes.
Поиск гомологов с помощью blastn дал всего два результата. Это неудивительно, так как гены эукариот содержат интроны, на которые слабо действует отбор (а значит они сильно вариабельны в далеких таксонах, что делает поиск по нуклеотидным последовательностям эукариот плохим способом поиска гомологов). Также, blastn не учитывает вырожденность генетического кода (одна аминокислота может кодироваться несколькими кодонами). А значит множество синонимичных мутаций, меняющие нуклеотидную последовательность, но слабо подверженные отбору, будут ухудшать работу blastn.

Дальше я решил использовать метод tblastx, так как мне хотелось давать на вход нуклеотидную последовательность, а не белковую. Были использованы следующие параметры: word size - 3; threshold - 0.05. Количество находок - 13, количество гомологичных участков - 24. Было достаточно ожидаемо, что будут хорошие находки, так как АТФ-синтаза - это очень важный белок, а значит его аминокислотная последовательность должна быть довольно консервативной (то есть гомологи должны находиться без проблем). При уменьшении word_size до 2, количество находок не меняется.

3. Найдём в геноме эукариота гены основных рибосомальных РНК по далекому гомологу
Следующей задачей стало найти в сборке генома круглого червя гены рРНК по далекому гомологу - генам рРНК из E. coli, используя standalone (локальный) BLAST. Поиск проводился по последовательностям генов 23S и 16S рРНК. Обе молекулы входят в состав рибосомы (16S - малой субъединицы, 23S - большой), выполняют структурную функцию.
Для начала нужно было проиндексировать последовательности сборки, чтобы получить локальную базу данных. Команда:
Поиск производился с помощью blastn, так как, во-первых, запросом здесь является нуклеотидная последовательность, во-вторых, E.coli и C.elegans - это достаточно далекие по систематическому положению организмы, поэтому даже такие консервартивные последовательности, как рРНК будут несколько отличаться между ними (поэтому megablast не подходит). Размер слова - 7 (повысим шансы найти что-то), пороговое значение E-value - 0,05 (остальные параметры по умолчанию). Команды:
blastn -task blastn -query rRNA_23s -db GCA_000002985.3_WBcel235_genomic.fna -out blastn_23.out -word_size 7 -evalue 0.05 -outfmt 7
В результате получили 2 файла с табличным форматом выдачи: blastn_16.out, blastn_23.out.
Результаты:
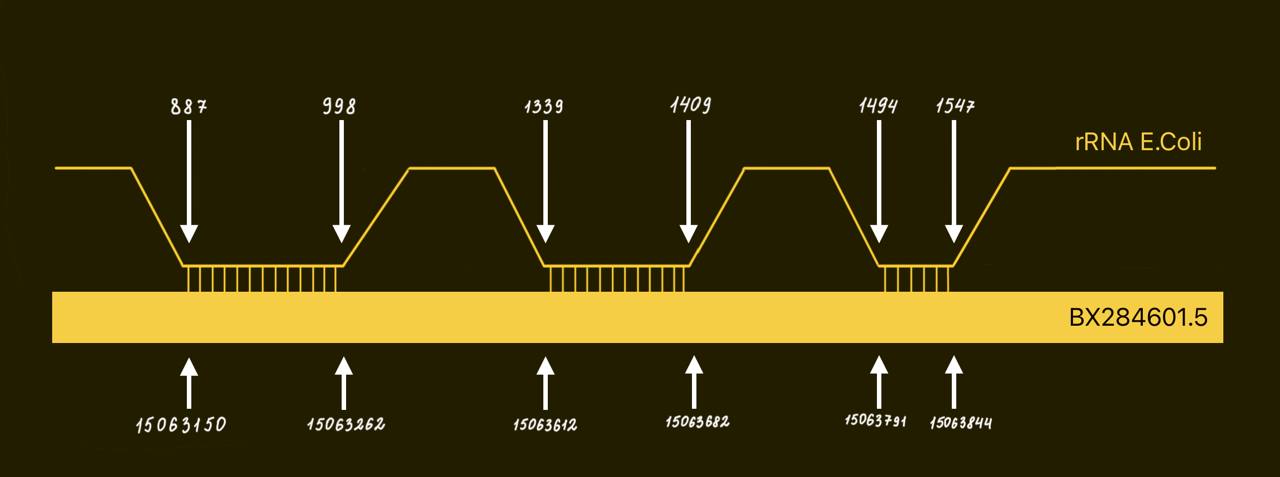
Выводы
Среди 16S рРНК получилось меньше находок. Это связано с тем, что 16S рРНК участвует в специфичных для прокариот процессах: 3′-конец содержит последовательность анти-Шайна-Дальгарно, с помощью которой 16S рРНК связывается с мРНК (которой нет у эукариот); 3′-конец связывается с факторами, участвующими в инициации трансляции (S1 и S21).
4. Подберём пару геномов и построим карты их локального сходства
Нуклеотидные последовательности выбирались для бактерий из одного семейства Enterobacteriaceae. Так мой выбор пал на плазмиды бактерий Escherichia coli и Salmonella enterica.
Поиск производился на сайте NCBI с помощью следующих запросов:
(Salmonella enterica [Organism]) AND plasmid [Title]
С помощью трёх разных алгоритмов: blastn, megablast и tblastx были построены карты локального сходства.
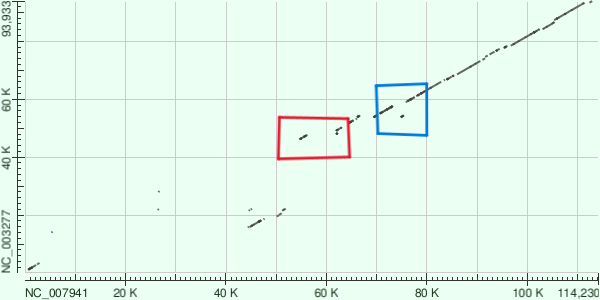
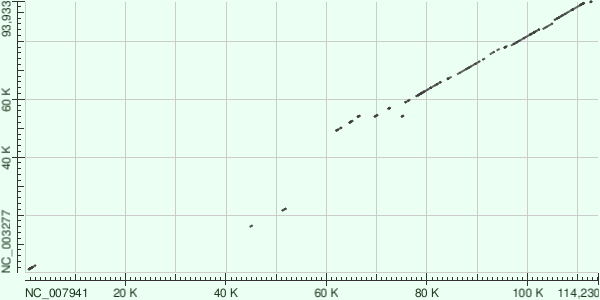
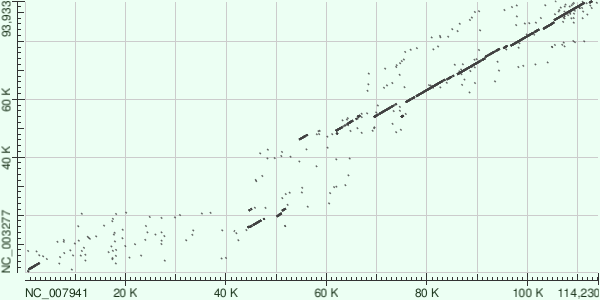
Программа megablast нужна для поиска практически идентичных участков, blastn, наоборот, более чувствительна. Поэтому, megablast может пропустить многие интересные области. Например, некоторые участки явно различимы только на графике, построенным программой blastn. С другой стороны на графике blastn гораздо больше "шума". График, построенный tblastx гораздо более цельный (то есть алгоритм более чувствительный), но при это на нём очень иного шума.