Предсказание вторичной структуры заданной тРНК и анализ НК-белкового комплекса
Петренко Павел
Факультет биоинженерии и биоинформатики, Московский Государственный Университет имени М.В.Ломоносова
Предсказание вторичной структуры заданной тРНК
В данном практикуме я работал со структурой 1pp8 (pdb).
einverted
! einverted -sequence rna.seq -gap 12 -threshold 2 -match 3 -mismatch -3 -outfile outfile -outseq seqout
В итоге нашли акцепторный стебель: 1-6 и 64-69.
ViennaRNA
seq = "GGGGUAUCGCCAAGCGGUAAGGCACCGGAUUCUGAUUCCGGCAUUCCGAGGUUCGAAUCCUCGUACCCCAGCCA"
# create fold_compound data structure (required for all subsequently applied algorithms)
fc = RNA.fold_compound(seq)
# compute MFE and MFE structure
(mfe_struct, mfe) = fc.mfe()
# rescale Boltzmann factors for partition function computation
fc.exp_params_rescale(mfe)
# compute partition function
(pp, pf) = fc.pf()
# compute MEA structure
(MEA_struct, MEA) = fc.MEA()
# compute free energy of MEA structure
MEA_en = fc.eval_structure(MEA_struct)
# print everything like RNAfold -p --MEA
print("%s\n%s (%6.2f)" % (seq, mfe_struct, mfe))
print("%s [%6.2f]" % (pp, pf))
print("%s {%6.2f MEA=%.2f}" % (MEA_struct, MEA_en, MEA))
print(" frequency of mfe structure in ensemble %g; ensemble diversity %-6.2f" % (fc.pr_structure(mfe_struct), fc.mean_bp_distance()))
GGGGUAUCGCCAAGCGGUAAGGCACCGGAUUCUGAUUCCGGCAUUCCGAGGUUCGAAUCCUCGUACCCCAGCCA
((((((..(((.........))).(((((.......))))).....(((((.......)))))))))))..... (-27.30)
(((((({,(({..,,,,...}}}.(((((.......))))).....|((((.......)))))))))))..... [-28.32]
((((((..(((.........))).(((((.......))))).....(((((.......)))))))))))..... {-27.30 MEA=59.93}
frequency of mfe structure in ensemble 0.190896; ensemble diversity 14.06
1
SVG('ggg.svg')
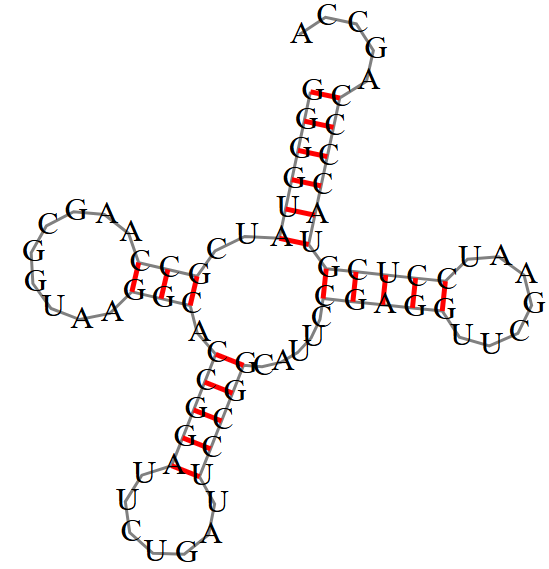
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 1-6; 65-74 | 1-6; 64-69 | 1-6; 64-69 |
| D-стебель | 9-24 | - | 9-23 |
| T-стебель | 48-64 | - | 47-63 |
| Антикодоновый стебель | 25-43 | - | 25-41 |
| Общее число канонических пар нуклеотидов | 28 | 6 | 19 |
Поиск ДНК-белковых контактов в заданной структуре
В PyMol были исследованы ДНК-белковые контакты в структуре 1pp8 (pdb), цепи M, R, Y. Использовались команды:
Где pol - для полярных взаимодействий, nonpol - для неполярных взаимодействий. Видим, что получилось много неполярных контактов и взаимодействий с остатками фосфорной кислоты. Возможно, белок в основном за счет сахарнофосфатного остова, а также за счет взаимодействия с большой бороздкой.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 1 | 14 | 15 |
| остатками фосфорной кислоты | 5 | 8 | 13 |
| остатками азотистых оснований со стороны большой бороздки | 0 | 9 | 9 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
Далее помощью программы nucplot были получена схема ДНК-белковых контактов в цепях M, R, Y комплекса 1PP8 (pdb):
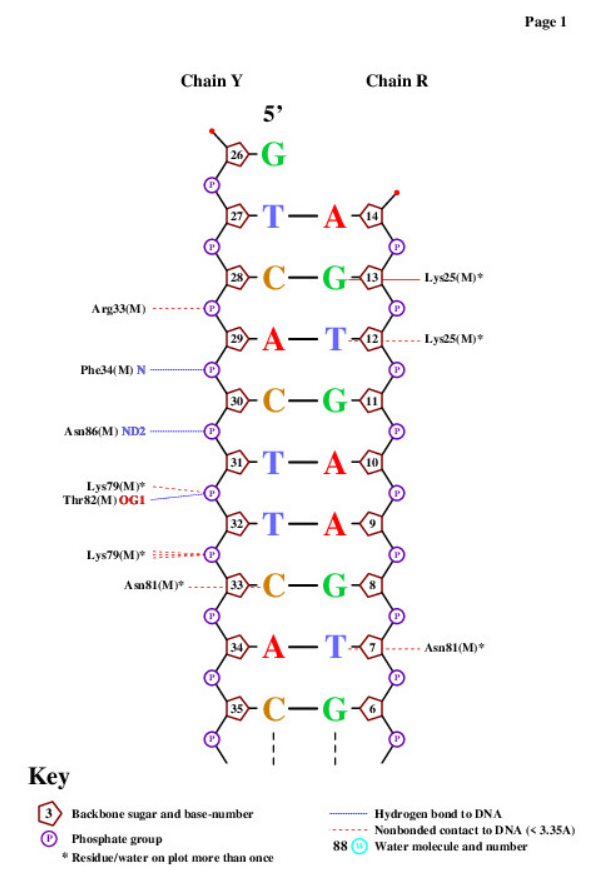
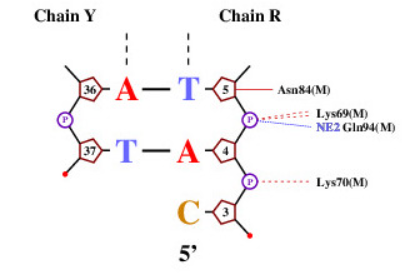
Аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК:
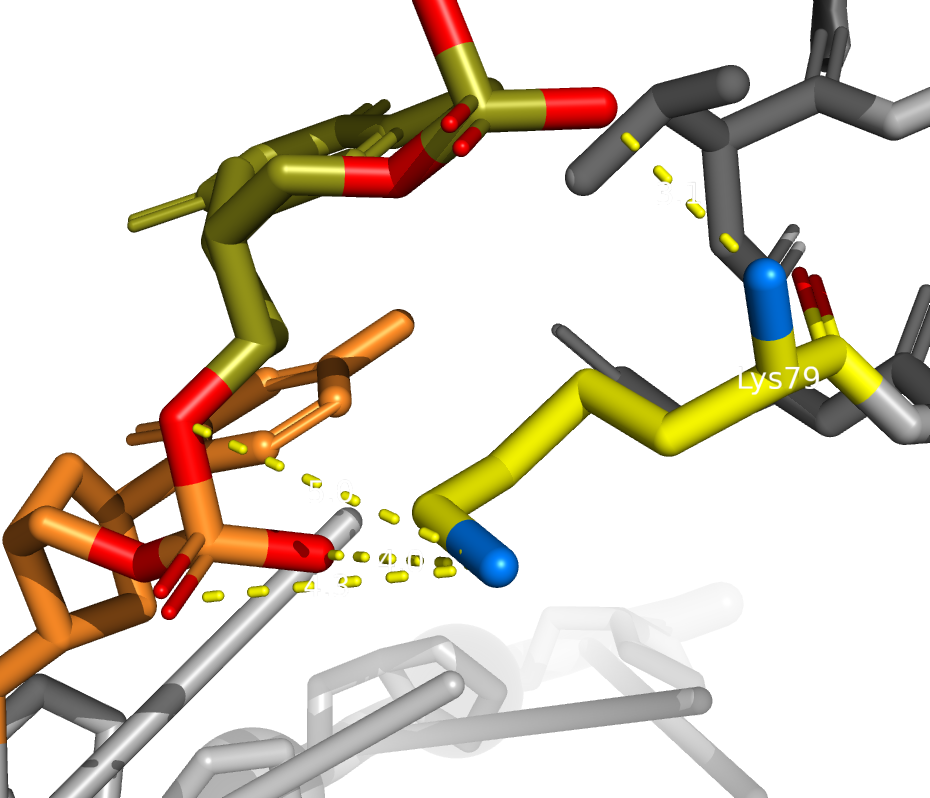
Я выбрал Lys79, который образует 4 водородные связи с фосфатами.
Аминокислотный остаток, по-вашему мнению, наиболее важный для распознавания последовательности ДНК:
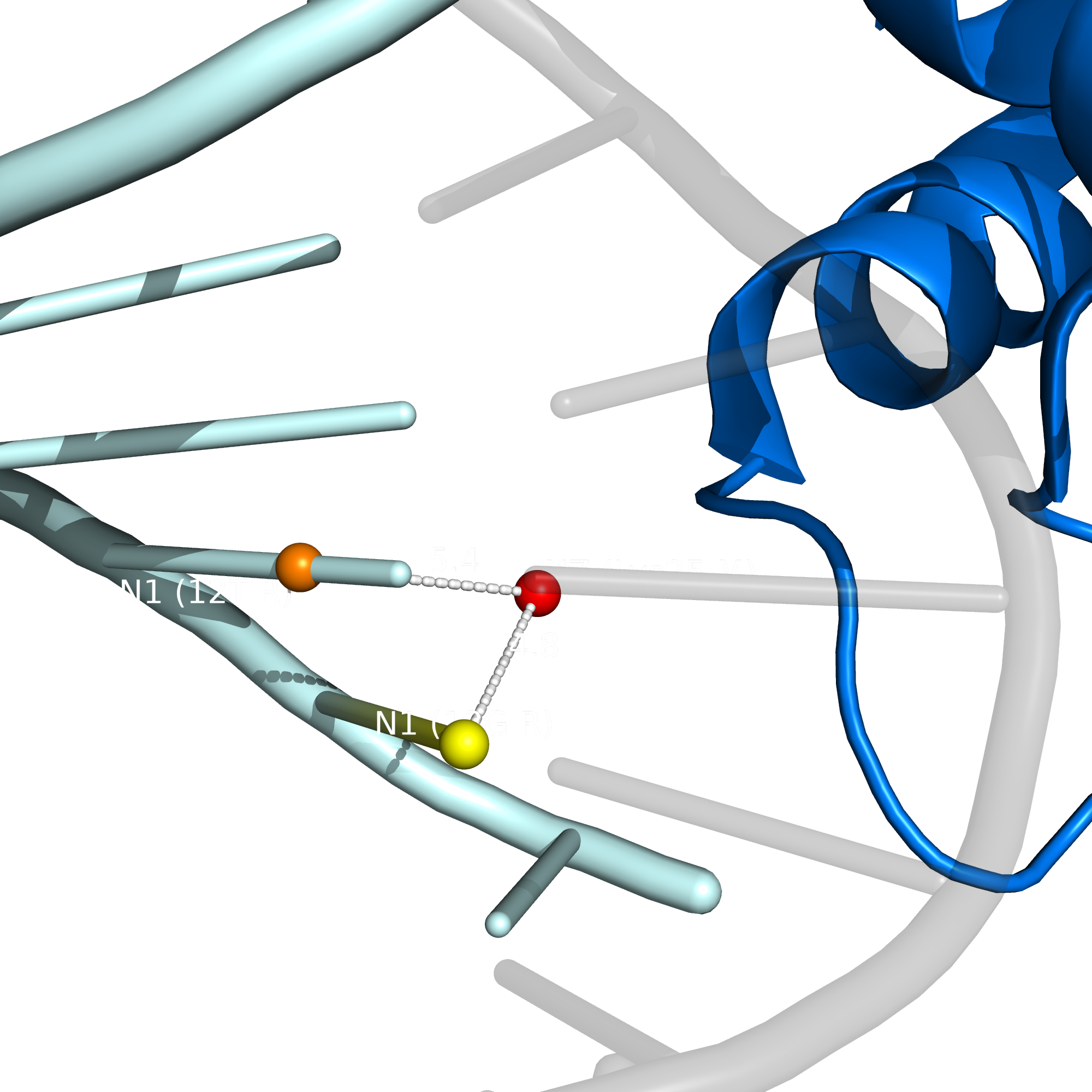
Я отбирал такой аминокислотный остаток, который в большей степени взаимодействует с азотистыми основаниями нуклеиновой кислоты. Поэтому мой выбор пал на Lys25 цепи М, который взаимодействует с 12Т, 13G цепи R.