
На сайте Uniprot был произведён поиск по CDS идентификатору белка (AJG99379.1). Оказалось, что данный белок был найден в организме Clostridium beijerinckii и описан как Sialidase(Сиалидаза/нейраминидаза). Запись о нём была включена в базу 1 апреля 2015 года авторами Little G.T. и Minton N.P. Белок имеет 755 аминокислотных остатков в длину, что имеет массу 8,763 кДА.Систематическое положение продуцента биополимера представлено в таблице 1. Статус аннотирования белка указан как 4, Predicted, что говорит о том, что доказательства о существовании белка были получены косвенным путём. В таблице 2 указаны все основные зарактеристики белка, представленные в UniProtKB записи.
Из записи можно узнать, что для сиалидазы известна полная структура. Белок имеет сигнальную часть (остатки 1-31) и цепь(32-755), которая содержит домен(41-183).Сигнальная часть, вероятно, играет роль специфического сигнала, позволяющего белку перемещаться по компартментам и/или органоидам клетки, а также, возможно, вне её. Цепь, как правило, выполняет структурную функцию, поддерживая содержащиеся в домене радикалы аминокислотных остков в опредлённом соподчинённом положении. Белок является ферментом, относимым к гидроксил-гидролазам. Запись не имела как RecName, так и PDB ID, поэтому я взял SubName и отметил прочерки в таблице соответственно.
В таблице 3 представлена аминокислотная последовательность сиалидазы. Анализ даёт информацию о среднем содержании гидрофобных аминокислот в белке(Изображение 1).
| Таксономическая единица | Название таксономической единицы |
| Отдел | Firmicutes |
| Класс | Clostridia |
| Порядок | Clostridiales |
| Семейство | Clostridiaceae |
| Род | Clostridium |
| Вид | Clostridium beijerinckii |
| Uniprot ID | Uniprot AC | Refseq ID | PDB ID | Длина белка | Молекулярная масса белка(кДа) | Рекомендуемое Unirpot название |
| A0A0B5QAX8_CLOBE | A0A0B5QAX8 | WP_041896799.1 | ---- | 755 | 8,763 | Sialidase |
MIRRNKRILS LTLSMAVFTT MFMSTSFITK AETVSLGANS EITSNASTES TAVATNIALN KPSTASSVTG GNTASLAVDG NAGTRWESAQ GSDPQWISID LGGSYNISGV KLNWETAAAK DYKIQVSTDN KNWIDAYTKT GGTGGVENIA FNSTATGRYI RMLGTTRTTQ YGYSLWEFEV YGIPDGNTVN NVDLGPNVKI FDPSMPSSDI QNTVDSVFSK METNQFGNER YAFLFKPGSY NVNVNVGFFT SVLGLGKTPD AVNITGAVRC EADWMGGNAT CNFWRSVENV AVTPTYSSNN LAPAGTLTWA VSQAAPMRRV HIKGGLSLWD PLGTNYDGAW SSGGFIADSK IDNSITSGSQ QQFFTRNSQM GSWNGANWNM VFVGNNGAPT DDNAYPSTPD TVVSQTPAIR EKPFLYIDDS GNYQVFIPDL RKNSQGITWT NGLGQGTSLS IDQFYIAKPD TSTAESINAA LSQGKNIIFT PGVYHLSDAI NVTKSNTVIL GLGLATLIPD NGTAAMNISD VDGVKVSGVL FDAGAKNSPV LLKVGQDGSS ADHSANPTSL SDLFFRIGGA AVGNADTSLK INSNNVIGDD FWVWRADHGT GVGWTVNNAK NGVIVNGNNV TLYGLFVEHF KEYQTIWNGN GGKVYFYQSE LPYDVPNQAS WMSNNGTQNG YASYKVADSV TSHQLFGSGI YSYFRDSVVS ENNGIEVPNA SGVKVHHACS VYLSGNGEIT HVVNNTGNTA KSGDMKQSVT DYPNS |

В кластере со стопроцентной идентичностью не содержится иных белков кроме сиалидазы из Clostridium beijerinckii. В кластере с девяностопроцентной идентичностью имеется фактор коагуляции и другие разновидности сиалидазы из родственных организмов. На изображении 2 показано выравнивание последовательностей белков, выполненное с помощью программы Jalview. Фильтр по консервативности и дерево на изображении 3 дают представление о родстве и изменениях последовательностей. В кластере с пятидесятипроцентной идентичностью содержится большое количество количество сходных белков, имеющих тот же, либо изменённый, домен, сигнальную часть или цепь.
| Тип кластера | Идентификатор кластера | Количество белков в кластере |
| Uniref100 | UniRef100_A0A0B5QAX8 | 1 |
| Uniref90 | UniRef90_A6LWU8 | 6 |
| Uniref50 | UniRef50_G8SCP7 | 509 |
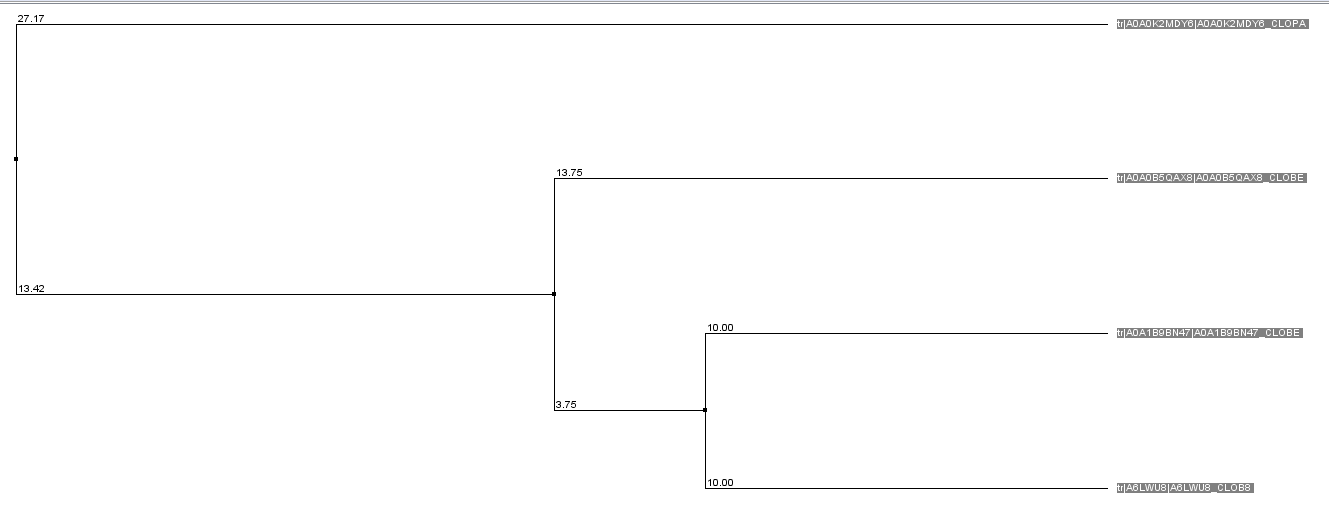
 Изображение 3. Дерево 90% идентичность
Изображение 3. Дерево 90% идентичность
| № | Текст запроса | Количество найденных белков | Количество Reviewed белков | Другое |
| 1 | name:sialidase | 5,591 | 36 | Сиалидаза есть в Swiss-Prot только для модельных организмов и человека |
| 2 | name:sialidase AND organism:"Clostridium beijerinckii (Clostridium MP) [1520]" | 4 | 0 | |
| 3 | name:sialidase taxonomy:clostridiaceae | 80 | 2 | Известна достоверная структура для белков Clostridium septicum и Clostridium perfringens |
| 4 | name:sialidase taxonomy:firmicutes | 526 | 6 | |
| 5 | keyword:homeobox | 81,898 | 1,664 | Данный домен очень важен для большинства форм живого |
| 6 | keyword:homeobox taxonomy:vertebrata | 33,015 | 1,140 | Домен преобладает у vertebrata |
| 7 | keyword:homeobox taxonomy:viridiplantae | 14,945 | 263 | Домен меньше представлен у viridiplantae |
| 4 | name:trypsin | 13,241 | 310 | |
| 4 | name:trypsin keyword:inhibitor | 347 | 196 |
Данные свидетельствуют о том, что проводить поиск по множеству критериев гораздо продуктивнее.
Была найдена запись в базе данных RefSeq, соответствующая моему белку. В записи, в основном, содержится лишь самая необходимая информация об объекте; имеется длина, идентификаторы, систематическое положение организма, ссылку на источник, разбиение последовательности на функциональные/смысловые единицы, аминокислотная последовательность. Отсутствуют перекрёстные ссылки на другие базы данных, а также рекомендованное название и статус анонсирования.
Мною был выбран гемоглобин (альфа-цепь) с идентификатором UniProtKB - P69905. С 2005 года было внесено 158 записей этого белка. На данный момент он имеет две версии: Entry version 158 (15 Feb 2017) и Sequence version 2 (23 Jan 2007).
Стоит изучить историю обогощения знаний о нём.
158 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2017_02 2017-02-15 157 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2017_01 2017-01-18 156 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_11 2016-11-30 155 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_10 2016-11-02 154 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_09 2016-10-05 153 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_08 2016-09-07 152 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_07 2016-07-06 151 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_06 2016-06-08 150 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_05 2016-05-11 149 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_04 2016-04-13 148 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_03 2016-03-16 147 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_02 2016-02-17 146 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2016_01 2016-01-20 145 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_12 2015-12-09 144 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_11 2015-11-11 143 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_10 2015-10-14 142 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_09 2015-09-16 141 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_08 2015-07-22 140 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_07 2015-06-24 139 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_06 2015-05-27 138 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_05 2015-04-29 137 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_03 2015-03-11 136 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_02 2015-02-04 135 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2015_01 2015-01-07 134 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_11 2014-11-26 133 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_10 2014-10-29 132 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_09 2014-10-01 131 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_08 2014-09-03 130 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_07 2014-07-09 129 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_06 2014-06-11 128 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_05 2014-05-14 127 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_04 2014-04-16 126 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_03 2014-03-19 125 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_02 2014-02-19 124 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2014_01 2014-01-22 123 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_12 2013-12-11 122 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_11 2013-11-13 121 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_10 2013-10-16 120 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_09 2013-09-18 119 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_08 2013-07-24 118 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_06 2013-05-29 117 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_05 2013-05-01 116 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_04 2013-04-03 115 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_03 2013-03-06 114 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2013_02 2013-02-06 113 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_11 2012-11-28 112 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_10 2012-10-31 111 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_09 2012-10-03 110 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_08 2012-09-05 109 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_07 2012-07-11 108 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_06 2012-06-13 107 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_04 2012-04-18 106 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_03 2012-03-21 105 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_02 2012-02-22 104 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2012_01 2012-01-25 103 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_12 2011-12-14 102 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_11 2011-11-16 101 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_10 2011-10-09 100 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_09 2011-09-21 99 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_08 2011-07-27 98 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_07 2011-06-28 97 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_06 2011-05-31 96 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_05 2011-05-03 95 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_04 2011-04-05 94 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_03 2011-03-08 93 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_02 2011-02-08 92 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2011_01 2011-01-11 91 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2010_12 2010-11-30 90 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2010_11 2010-11-02 89 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2010_10 2010-10-05 88 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2010_09 2010-08-10 87 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2010_08 2010-07-13 86 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2010_07 2010-06-15 85 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2010_05 2010-04-20 84 .txt 2 .fasta HBA_HUMAN Swiss-Prot 2010_04 2010-03-23 83 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.15/57.15 2010-03-02 82 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.13/57.13 2010-01-19 81 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.12/57.12 2009-12-15 80 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.11/57.11 2009-11-24 79 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.10/57.10 2009-11-03 78 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.9/57.9 2009-10-13 77 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.8/57.8 2009-09-22 76 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.7/57.7 2009-09-01 75 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.6/57.6 2009-07-28 74 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.5/57.5 2009-07-07 73 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.4/57.4 2009-06-16 72 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.3/57.3 2009-05-26 71 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.2/57.2 2009-05-05 70 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.1/57.1 2009-04-14 69 .txt 2 .fasta HBA_HUMAN Swiss-Prot 15.0/57.0 2009-03-24 68 .txt 2 .fasta HBA_HUMAN Swiss-Prot 14.9/56.9 2009-03-03 67 .txt 2 .fasta HBA_HUMAN Swiss-Prot 14.8/56.8 2009-02-10 66 .txt 2 .fasta HBA_HUMAN Swiss-Prot 14.6/56.6 2008-12-16 65 .txt 2 .fasta HBA_HUMAN Swiss-Prot 14.5/56.5 2008-11-25 64 .txt 2 .fasta HBA_HUMAN Swiss-Prot 14.4/56.4 2008-11-04 63 .txt 2 .fasta HBA_HUMAN Swiss-Prot 14.2/56.2 2008-09-23 62 .txt 2 .fasta HBA_HUMAN Swiss-Prot 14.1/56.1 2008-09-02 61 .txt 2 .fasta HBA_HUMAN Swiss-Prot 14.0/56.0 2008-07-22 60 .txt 2 .fasta HBA_HUMAN Swiss-Prot 13.5/55.5 2008-06-10 59 .txt 2 .fasta HBA_HUMAN Swiss-Prot 13.4/55.4 2008-05-20 58 .txt 2 .fasta HBA_HUMAN Swiss-Prot 13.3/55.3 2008-04-29 57 .txt 2 .fasta HBA_HUMAN Swiss-Prot 13.0/55.0 2008-02-26 56 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.8/54.8 2008-02-05 55 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.7/54.7 2008-01-15 54 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.6/54.6 2007-12-04 53 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.5/54.5 2007-11-13 52 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.4/54.4 2007-10-23 51 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.3/54.3 2007-10-02 50 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.2/54.2 2007-09-11 49 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.1/54.1 2007-08-21 48 .txt 2 .fasta HBA_HUMAN Swiss-Prot 12.0/54.0 2007-07-24 47 .txt 2 .fasta HBA_HUMAN Swiss-Prot 11.3/53.3 2007-07-10 46 .txt 2 .fasta HBA_HUMAN Swiss-Prot 11.1/53.1 2007-06-12 45 .txt 2 .fasta HBA_HUMAN Swiss-Prot 11.0/53.0 2007-05-29 44 .txt 2 .fasta HBA_HUMAN Swiss-Prot 10.5/52.5 2007-05-15 43 .txt 2 .fasta HBA_HUMAN Swiss-Prot 10.4/52.4 2007-05-01 42 .txt 2 .fasta HBA_HUMAN Swiss-Prot 10.3/52.3 2007-04-17 41 .txt 2 .fasta HBA_HUMAN Swiss-Prot 10.2/52.2 2007-04-03 40 .txt 2 .fasta HBA_HUMAN Swiss-Prot 10.1/52.1 2007-03-20 39 .txt 2 .fasta HBA_HUMAN Swiss-Prot 9.6/51.6 2007-02-06 38 .txt 2 .fasta HBA_HUMAN Swiss-Prot 9.5/51.5 2007-01-23 37 .txt 1 .fasta HBA_HUMAN Swiss-Prot 9.4/51.4 2007-01-09 36 .txt 1 .fasta HBA_HUMAN Swiss-Prot 9.3/51.3 2006-12-12 35 .txt 1 .fasta HBA_HUMAN Swiss-Prot 9.2/51.2 2006-11-28 34 .txt 1 .fasta HBA_HUMAN Swiss-Prot 9.1/51.1 2006-11-14 33 .txt 1 .fasta HBA_HUMAN Swiss-Prot 9.0/51.0 2006-10-31 32 .txt 1 .fasta HBA_HUMAN Swiss-Prot 8.9/50.9 2006-10-17 31 .txt 1 .fasta HBA_HUMAN Swiss-Prot 8.8/50.8 2006-10-03 30 .txt 1 .fasta HBA_HUMAN Swiss-Prot 8.6/50.6 2006-09-05 29 .txt 1 .fasta HBA_HUMAN Swiss-Prot 8.4/50.4 2006-07-25 28 .txt 1 .fasta HBA_HUMAN Swiss-Prot 8.2/50.2 2006-06-27 27 .txt 1 .fasta HBA_HUMAN Swiss-Prot 8.1/50.1 2006-06-13 26 .txt 1 .fasta HBA_HUMAN Swiss-Prot 8.0/50.0 2006-05-30 25 .txt 1 .fasta HBA_HUMAN Swiss-Prot 7.7/49.7 2006-05-16 24 .txt 1 .fasta HBA_HUMAN Swiss-Prot 7.6/49.6 2006-05-02 23 .txt 1 .fasta HBA_HUMAN Swiss-Prot 7.5/49.5 2006-04-18 22 .txt 1 .fasta HBA_HUMAN Swiss-Prot 7.4/49.4 2006-04-04 21 .txt 1 .fasta HBA_HUMAN Swiss-Prot 7.3/49.3 2006-03-21 20 .txt 1 .fasta HBA_HUMAN Swiss-Prot 7.2/49.2 2006-03-07 19 .txt 1 .fasta HBA_HUMAN Swiss-Prot 7.1/49.1 2006-02-21 18 .txt 1 .fasta HBA_HUMAN Swiss-Prot 7.0/49.0 2006-02-07 17 .txt 1 .fasta HBA_HUMAN Swiss-Prot 6.9/48.9 2006-01-24 16 .txt 1 .fasta HBA_HUMAN Swiss-Prot 6.6/48.6 2005-12-06 15 .txt 1 .fasta HBA_HUMAN Swiss-Prot 6.5/48.5 2005-11-22 14 .txt 1 .fasta HBA_HUMAN Swiss-Prot 6.2/48.2 2005-10-11 13 .txt 1 .fasta HBA_HUMAN Swiss-Prot 6.0/48.0 2005-09-13 12 .txt 1 .fasta HBA_HUMAN Swiss-Prot 5.8/47.8 2005-08-30 11 .txt 1 .fasta HBA_HUMAN Swiss-Prot 5.6/47.6 2005-08-02 10 .txt 1 .fasta HBA_HUMAN Swiss-Prot 5.5/47.5 2005-07-19 9 .txt 1 .fasta HBA_HUMAN Swiss-Prot 5.4/47.4 2005-07-05 8 .txt 1 .fasta HBA_HUMAN Swiss-Prot 5.2/47.2 2005-06-07 7 .txt 1 .fasta HBA_HUMAN Swiss-Prot 5.0/47.0 2005-05-10 6 .txt 1 .fasta HBA_HUMAN Swiss-Prot 4.6/46.6 2005-04-26 5 .txt 1 .fasta HBA_HUMAN Swiss-Prot 4.5/46.5 2005-04-12 4 .txt 1 .fasta HBA_HUMAN Swiss-Prot 4.4/46.4 2005-03-29 3 .txt 1 .fasta HBA_HUMAN Swiss-Prot 4.3/46.3 2005-03-15 2 .txt 1 .fasta HBA_HUMAN Swiss-Prot 4.0/46.0 2005-02-01 1 .txt 1 .fasta HBA_HUMAN Swiss-Prot 3.5/45.5 2005-01-04 |
Нестандартные аминокислоты обозначаются как NON_STD. Селеноцистеин (Selenocysteine) представлен как U или Sec, пирролизин (Pyrrolysine) как O или Pyl.
| One-letter code | Three-letter code | Amino-acid name |
|---|---|---|
| A | Ala | Alanine |
| R | Arg | Arginine |
| N | Asn | Asparagine |
| D | Asp | Aspartic acid |
| C | Cys | Cysteine |
| Q | Gln | Glutamine |
| E | Glu | Glutamic acid |
| G | Gly | Glycine |
| H | His | Histidine |
| I | Ile | Isoleucine |
| L | Leu | Leucine |
| K | Lys | Lysine |
| M | Met | Methionine |
| F | Phe | Phenylalanine |
| P | Pro | Proline |
| S | Ser | Serine |
| T | Thr | Threonine |
| W | Trp | Tryptophan |
| Y | Tyr | Tyrosine |
| V | Val | Valine |
| O | Pyl | Pyrrolysine |
| U | Sec | Selenocysteine |
| B | Asx | Aspartic acid or Asparagine |
| Z | Glx | Glutamic acid or Glutamine |
| X | Xaa | Any amino acid |
Посттрансляционные модификации обозначаются как MOD_RES.
| Modification | Description |
|---|---|
| ACETYLATION | N-terminal of some residues and side chain of lysine |
| AMIDATION | Generally at the C-terminal of a mature active peptide after oxidative cleavage of last glycine |
| BLOCKED | Unidentified N- or C-terminal blocking group |
| FORMYLATION | Generally of the N-terminal methionine |
| GAMMA-CARBOXYGLUTAMIC ACID | Of glutamate |
| HYDROXYLATION | Generally of asparagine, aspartate, proline or lysine |
| METHYLATION | Generally of N-terminal phenylalanine, side chain of lysine, arginine, histidine, asparagine or glutamate, and C-terminal cysteine |
| PHOSPHORYLATION | Of serine, threonine, tyrosine, aspartate, histidine or cysteine, and, more rarely, of arginine |
| PYRROLIDONE CARBOXYLIC ACID | N-terminal glutamine which has formed an internal cyclic lactam. This is also called 'pyro-Glu'. Very rarely, pyro-Glu can be produced by modification of a N-terminal glutamate |
| SULFATION | Of tyrosine, serine or threonine |
Варианты последовательностей, претерпевших альтернативный сплайсинг обозначаются как VAR_SEQ. Дисульфидные мостики обозначаются как DISULFID.

© Кравченко Павел
2017