Pfam — база данных семейств белковых доменов. Каждое семейство в ней представлено множественным выравниванием фрагментов белковых последовательностей и скрытой марковской моделью (HMM). На март 2017 года в Pfam содержалось 16 712 записей (семейств), объединённых в 604 клана[1].
Для выполнения задания были использованы последовательности белков, гомологичных белку ELF2 из Poeciliopsis prolifica, найденных с помощью BLAST. В таблице 1 приведены параметры найденных последовательностей.
Последовательность ELF2 Poeciliopsis prolifica
>JAO98220.1 ELF2, partial [Poeciliopsis prolifica] MATSQHEGHANQLDLLIRAVEASGYSNVHCSDKTIEAAEALLHMDSPSSLREDRSPEAFTPQSEAAPDFL HAAMRPDVIGETEVEITTEDCCEEDEEEDEEEEMVTSLEEPEPDNEPVRKKRAGRKTKAPHSSISNGAPD LSFKKKPREGKAGSTTYLWEFLLDLLQDKNTCPRYIKWTQREKGIFKLVDSKAVSKLWGKHKNKPDMNYE TMGRALRYYYQRGILAKVEGQRLVYQFKEMPKNIVIIDED
| Название белка | Coverage | E-value | Identity | ID/AC |
| spectrin beta chain, non-erythrocytic 1 isoform 2 [Mus musculus] | 1 | 4.00E-24 | 1 | NP_033286.2 |
| spectrin beta chain, non-erythrocytic 1 isoform 1 [Mus musculus] | 1 | 4.00E-24 | 1 | NP_787030.2 |
| spectrin beta chain, non-erythrocytic 1 [Rattus norvegicus] | 0,83 | 1.00E-16 | 0,93 | NP_001013148.1 |
| spectrin beta chain, non-erythrocytic 1 isoform X1 [Castor canadensis] | 0,83 | 5.00E-15 | 0,9 | XP_020040264.1 |
| spectrin beta chain, non-erythrocytic 1 [Macaca mulatta] | 0,83 | 1.00E-14 | 0,88 | NP_001253897.1 |
| spectrin beta chain, non-erythrocytic 1 isoform X1 [Odocoileus virginianus texanus] | 0,83 | 2.00E-14 | 0,88 | XP_020770781.1 |
| spectrin beta chain, non-erythrocytic 1 [Bos taurus] | 0,83 | 2.00E-14 | 0,88 | NP_001179205.1 |
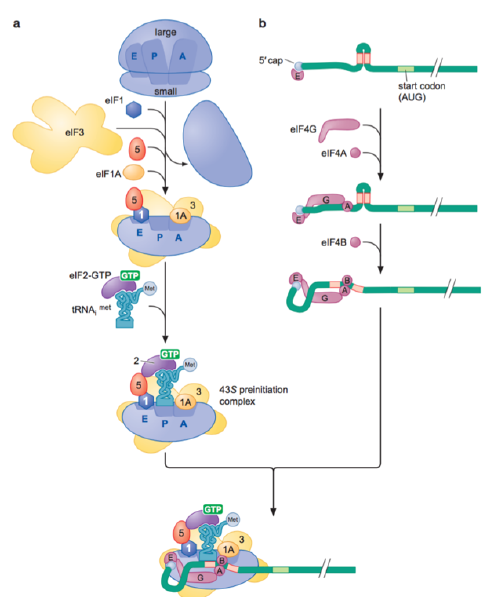
Из рисунка 1 видно, что ELF2 играет важную роль в инициации трансляции. Белки этого семейства участвуют в связывании участка двойной спирали транспортной РНК. Как известно, важной частью инициации является подготовка, обеспечивающее правильное позиционирование белков. Начало трансляции обеспечивается, в частности, затратой ГТФ, связываемого ELF2. Для анализа был выбран именно ELF2, так как он имеет оптимальный размер. Так, ELF3, ELF1 и ELF1A имеют либо слишком большой, либо слишком маленький размер.







Видно, что, в целом, программы одинаково справляются с выравниями консервативных участков. Выравнивания же сложных мест разнятся в силу отличий алгоритмов и,
скорее всего, платы за оказию штрафов за гэпы. Участки с расположенными друг над другом представителями разных групп, имеющими отрицательные веса в матрице
BLOSSUM62, выравниваются плохо. Хуже всего выравниваются длинные участки такого состава- очевидно. Разные программы/алгоритмы старются по- разному решить задачу
выравнивания за счёт, скорее всего, изменения и подбора параметнов. Из выравниваний видно, что "жадные" на гэпы/индели алгоритмы хуже справляются с выравниванием, в сравнении
с "умеренными". При выравнивании необходимо осозновать, хотим ли мы строить блоки всеми доступными средствами или же хотим максимально предсказать биологическое значение
полученного результата.
Лучшую воспроизводимость показали алгоритмы GLprobs и Probcons.
Самое быстрое выравнивание было построено с помощью Mafft.
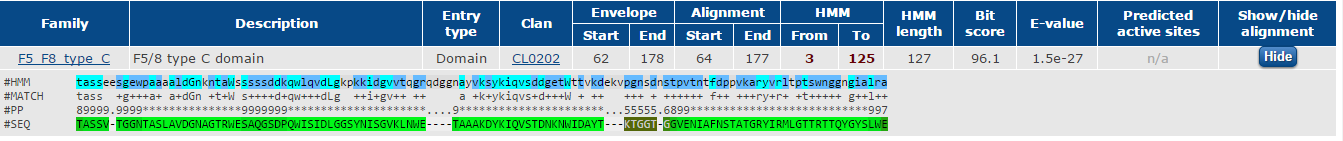
Основные параметры белка сиалидазы из бактерии Clostridium beijerinckii приведены в таблице 2. Используя сервис Pfam (Рисунок 2), был найден домен моего белка. Рисунок 2 демонстрирует единственный найденный F5/8 type C домен сиалидазы. Он представлен во многих факторах, обеспечивающих коагуляцию крови (5 и 8). Содержит два консервативных цистеина во многих белках, которые замыкают структуру посредством концевых дисульфидных связей. Сервис предлагает 1560 архитектур, содержащих данный домен. При сравнительном анализе видно, что, чаще всего, F5/8 type C домен представлен в двух экземплярах, располагающихся скраю последовательностей.
| Параметр | Значение |
| Идентификатор белка | AJG99379.1 |
| Идентификатор генома | CP010086 |
| Координаты гена в геноме | 3087698..3089965 |
| Длина гена (в парах нуклеотидов) | 2268 |
| Цепь (прямая или обратная) | Прямая |
| Длина белка (в аминокислотных остатках) | 755 |
Аминокислотная последовательность сиалидазы
>AJG99379.1 sialidase MIRRNKRILSLTLSMAVFTTMFMSTSFITKAETVSLGANSEITSNASTESTAVATNIA LNKPSTASSVTGGNTASLAVDGNAGTRWESAQGSDPQWISIDLGGSYNISGVKLNWET AAAKDYKIQVSTDNKNWIDAYTKTGGTGGVENIAFNSTATGRYIRMLGTTRTTQYGYS LWEFEVYGIPDGNTVNNVDLGPNVKIFDPSMPSSDIQNTVDSVFSKMETNQFGNERYA FLFKPGSYNVNVNVGFFTSVLGLGKTPDAVNITGAVRCEADWMGGNATCNFWRSVENV AVTPTYSSNNLAPAGTLTWAVSQAAPMRRVHIKGGLSLWDPLGTNYDGAWSSGGFIAD SKIDNSITSGSQQQFFTRNSQMGSWNGANWNMVFVGNNGAPTDDNAYPSTPDTVVSQT PAIREKPFLYIDDSGNYQVFIPDLRKNSQGITWTNGLGQGTSLSIDQFYIAKPDTSTA ESINAALSQGKNIIFTPGVYHLSDAINVTKSNTVILGLGLATLIPDNGTAAMNISDVD GVKVSGVLFDAGAKNSPVLLKVGQDGSSADHSANPTSLSDLFFRIGGAAVGNADTSLK INSNNVIGDDFWVWRADHGTGVGWTVNNAKNGVIVNGNNVTLYGLFVEHFKEYQTIWN GNGGKVYFYQSELPYDVPNQASWMSNNGTQNGYASYKVADSVTSHQLFGSGIYSYFRD SVVSENNGIEVPNASGVKVHHACSVYLSGNGEITHVVNNTGNTAKSGDMKQSVTDYPN S

Во вкладке "architectures" было найдено три белка, содержащих тот же домен.
Это неописанный белок из Caenorhabditis elegans. Он имеет 581 аминокислотных остатков в длину и два домена помимо F5_F8_type_С: BTB и BACK.

Обычно представлен как часть доменной архитектуры цинковых польцев, вирусных pox белков и белков, содержащих Klech мотивы. Индуцирует гомомерную и гетеромерную автодимеризацию. Может иметь ядерную и цитоплазматическую локализацию. Взаимодействует с убиквитин-связывающим белком.
Это фактор коагуляции пятый из Homo sapiens. Он имеет 2224 аминокислотных остатков в длину и сложную доменную архитектуру помимо F5_F8_type_С. Активирует тромбоциты. Ответственен за деградацию тромбоцитов, чувствителен к влиянию витамина К. Может влиять на кровоток, среднюю скорость тока крови. Утрата функции балка может приводить к широкому спектру заболеваний.


Это неописанный белок из Streptomyces sp.. Он имеет 1031 аминокислотных остатков в длину и несколько доменов помимо F5_F8_type_С




© Кравченко Павел
2017