Анализ крупных перестроек геномов
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
Выбор геномов
|
Для выполнения практикума были выбраны три генома бактерий, относящихся к роду [Clostridium] stercorarium из семейства Clostridiaceae. Клостридии уже ранее описывались в
- [Clostridium] stercorarium subsp. leptospartum DSM 9219 (firmicutes)
- [Clostridium] stercorarium subsp. stercorarium DSM 8532
- [Clostridium] stercorarium subsp. thermolacticum DSM 2910
Были скачаны последовательности геномов бактерий. Каждая содержит по одной хромосоме без плазмид. Найти их можно в директории
Исследование перестроек в геномах
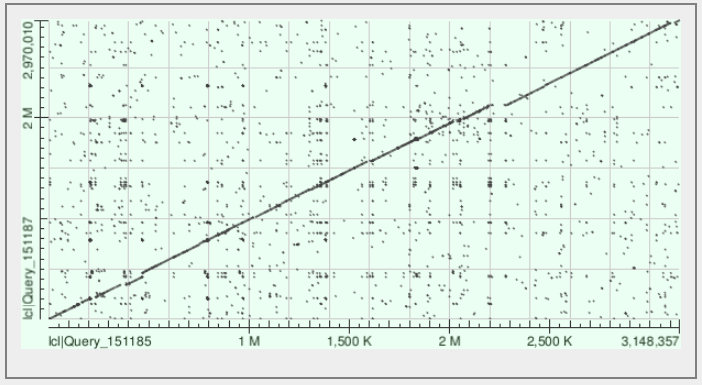
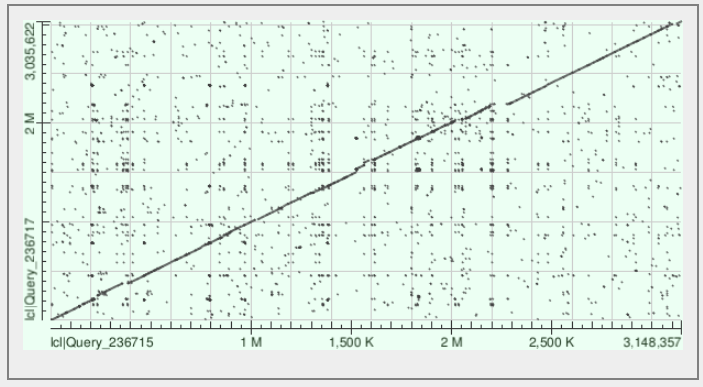
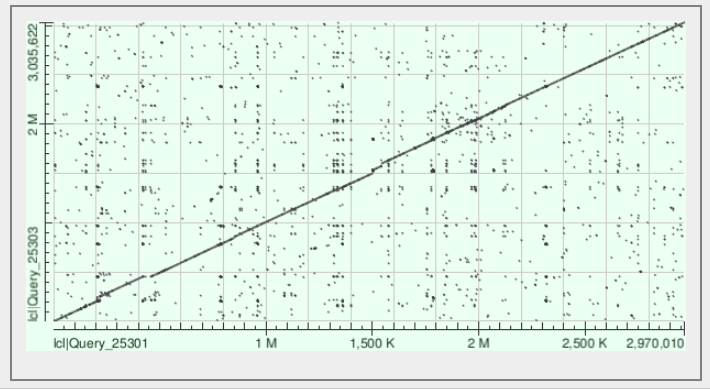
С помощью программы blast2seq был произведён поиск геномных перестроек между тремя штаммами бактерий. На рисунках 2-4 показаны получившиеся карты локального сходства. Из полученных данных следует, что выбор был произведён неудачно и поиск следует продолжить, так как крупных геномных перестроек не наблюдается.
|
|
|
Было найдено ещё несколько аналогичных близких троек последовательностей. Наибольший интерес представляли следующие ниже карты.

|
Для выполнения практикума были выбраны три генома бактерий, относящихся к другому виду -
Bacillus velezensis
из рода Bacillus.
- Bacillus velezensis strain ATR2
- Bacillus velezensis strain SCDB 291
- Bacillus velezensis strain SCGB 1
Последовательности лежат в директории
Параметры выравниваний геномов
Query cover, полученное для трёх выравниваний с помощью blast2seq было в каждом случае равно 93%.
Исследование перестроек в геномах
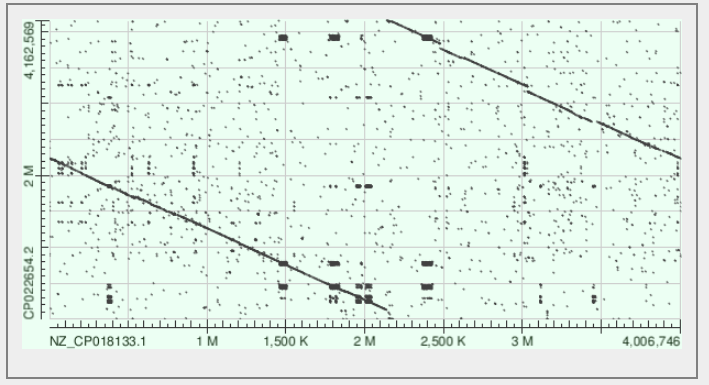
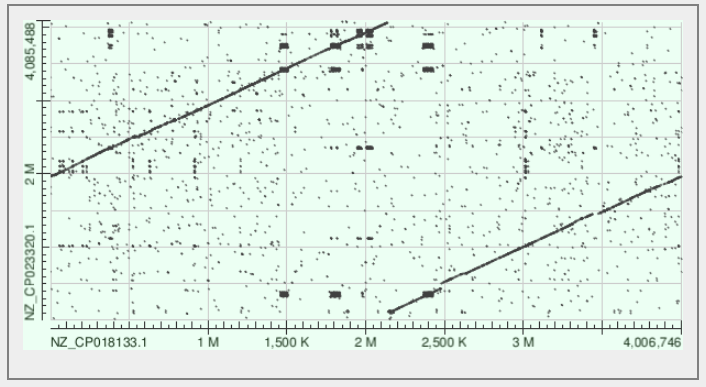
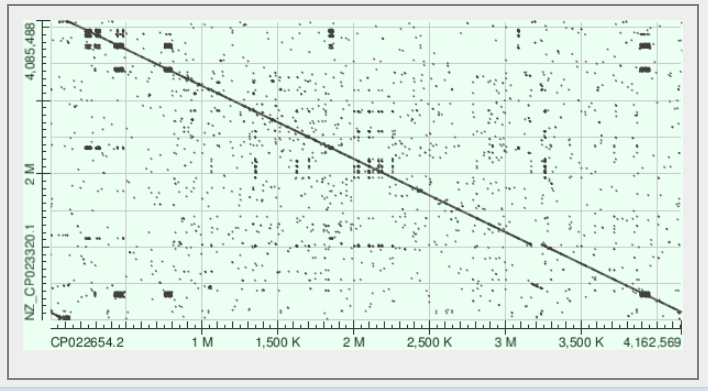
С помощью программы blast2seq повторно был произведён поиск геномных перестроек между тремя штаммами бактерий. На рисунках 5-7 показаны получившиеся карты локального сходства.
|
|
|
Карты локального сходства говорят об отсутствии крупных перестроек в геноме разных штаммов. Выравнивания говорят о том, что кольцевые хромосомы бактерий собирались и были записан, начиная с разных геномных координат. Стоит отметить наличие около десятка мелких блоков локального сходства, присутствующих на каждой из карт. Скорее всего, это участки коротких дупликаций или повторов, многократно повторённые в геномах или в одном из геновов.
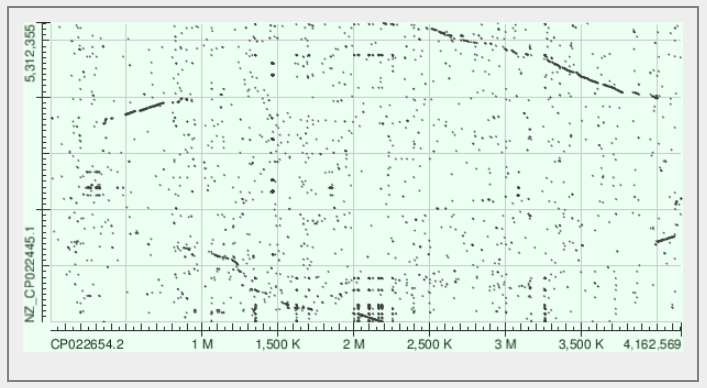
Чтобы найти и описать больше геномных перестроек и сравнить данные выравнивания заведомо неродственных штаммов, был выбран штамм из другого рода - Bacillus cereus C1L. Видно, что последовательности имеют низкое сходство. Участок в 300 пар нуклеотидов является общим для обоих штаммов и располагается примерно от 500 до 800 пар нуклеотидов по оси абсцисс. У предков данных штаммов произошла инверсия участков от 1 до 2.25 и от 2.5 и до 4.5 Мбаз. Их разбиение может свидетельствовать о сборке геномов с разных точек. Также имеется гомологичный участок от 4.5 до 4.7 Мбаз по оси х. Сравнение геномов отдалённых штаммов даёт сложно интерпретируемый результат.
|
|
С n-ой попытки всё же были найдены штаммы, представляющие некоторый интерес. Их ID:
CP009512.1
CP004144.1
CP009512.1
Параметры выравниваний геномов
Query cover, полученные для трёх выравниваний с помощью blast2seq были равны:
1-2 87% С покрытием 99%
1-3 93% С покрытикм 99%
2-3 98% С покрытием 97%
С помощью NPG-explorer были найдены параметры, оценивающие сходство трёх геномов.
Исследование перестроек в геномах
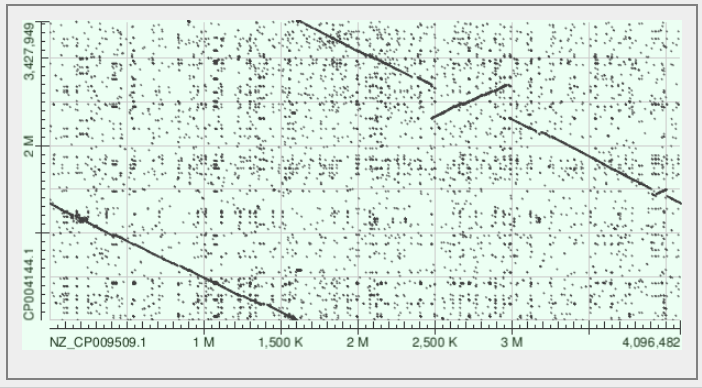
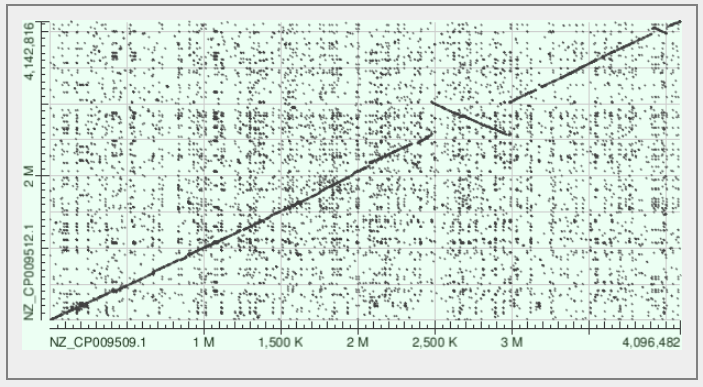
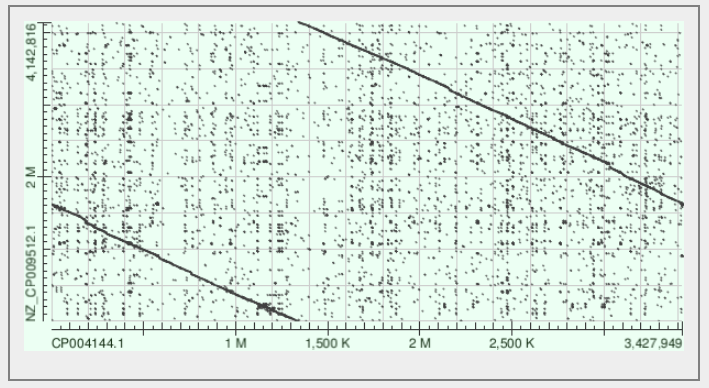
На рисунках 11-13 показаны получившиеся карты локального сходства.
|
Заметна инверсия участка длинной 1 Мбаза между 2.5 и 5 Мбазы. Также имеется небольшая инверсия ближе к 4 Мбазам. Инвертированииый участок был найден в 4 хромосоме человека с E-value 0.029.
|
Заметна, как и на рисунке 10, инверсия участка длинной 1 Мбаза между 2.5 и 5 Мбазы. Также имеется небольшая инверсия ближе к 4 Мбазам.
|
Бласт против последовательности, отсутствующей во втором геноме, но присутствующей в третьей (координаты 3.1 Мбазы) находит данную последовательность в геномах родственных бактерий с низким E-value и с очень высоким E-value (2.9) говорит, что присутствует гомология с регуляторным фактором интерферонов, что крайне маловероятно: этому гену не от куда взяться в бактериальном геноме. Данный участок присутствует в десяти близкородственных штаммах, что может свидетельствовать в пользу его делеции в штамме 2. Но возможно также, что имеется ещё большая клада, где этот ген отсутствует. Чтобы проверить это, был выполнен поиск участка с делецией с помощью blastn. Достоверная выдача была гораздо меньше, а значит, не так давно произошла делеция данного участка. Карта локального сходства 13 говорит о близком родстве штаммов 2 и 3. Также похоже, что сборка производилась, начиная с разных участков.
NPG-explorer
С помощью NPG-explorer были найдены блоки, оценивающие сходство трёх геномов. Было найдено 49 s-блоков, имеющих по одному фрагменту из каждого генома. 25 h-блоков, имеющих всего по два фрагмента из трёх геномов. 19 u-блоков - уникальных последовательностей в каждом из геномов, не встречающихся в других двух. 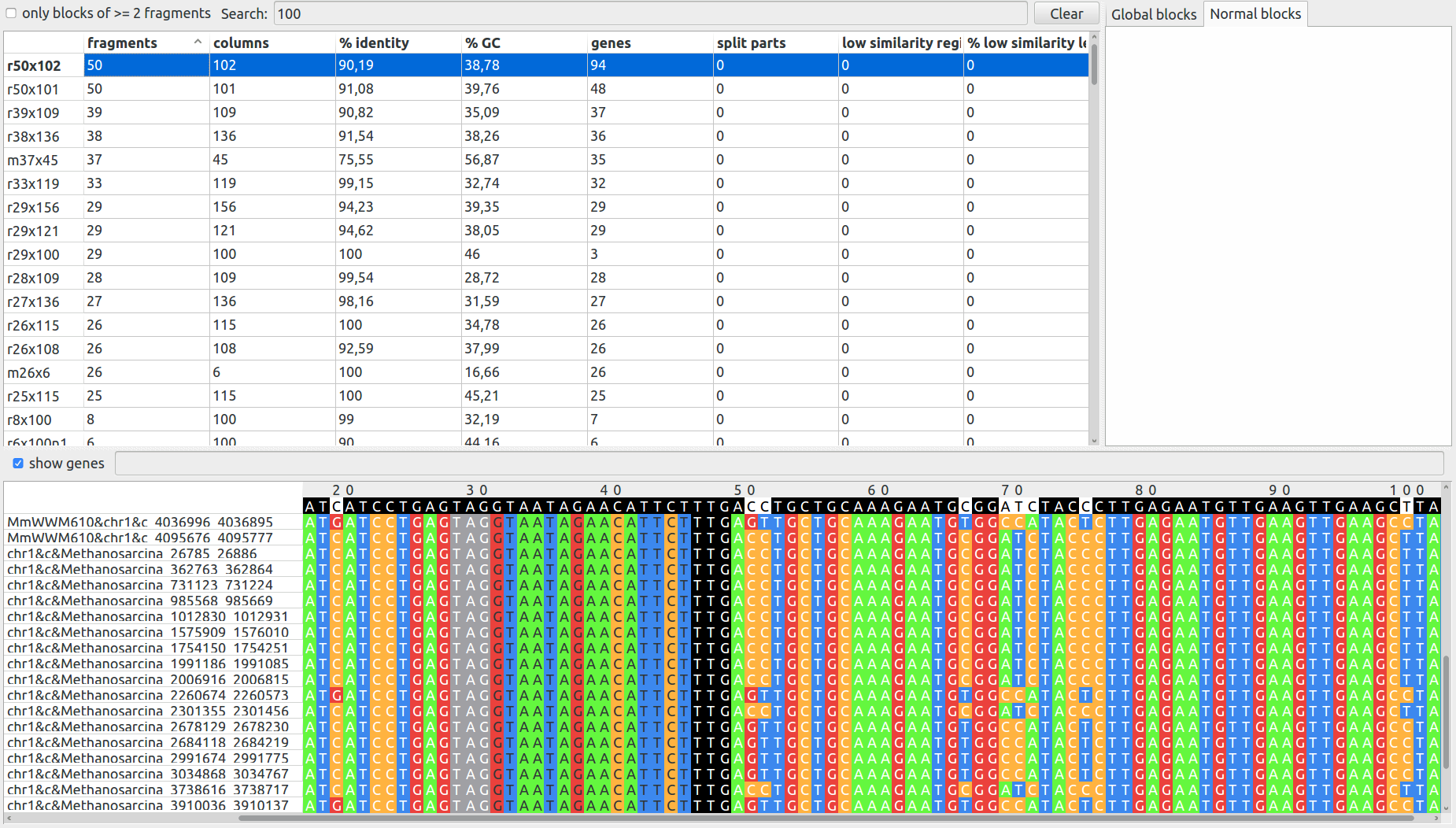
|
|
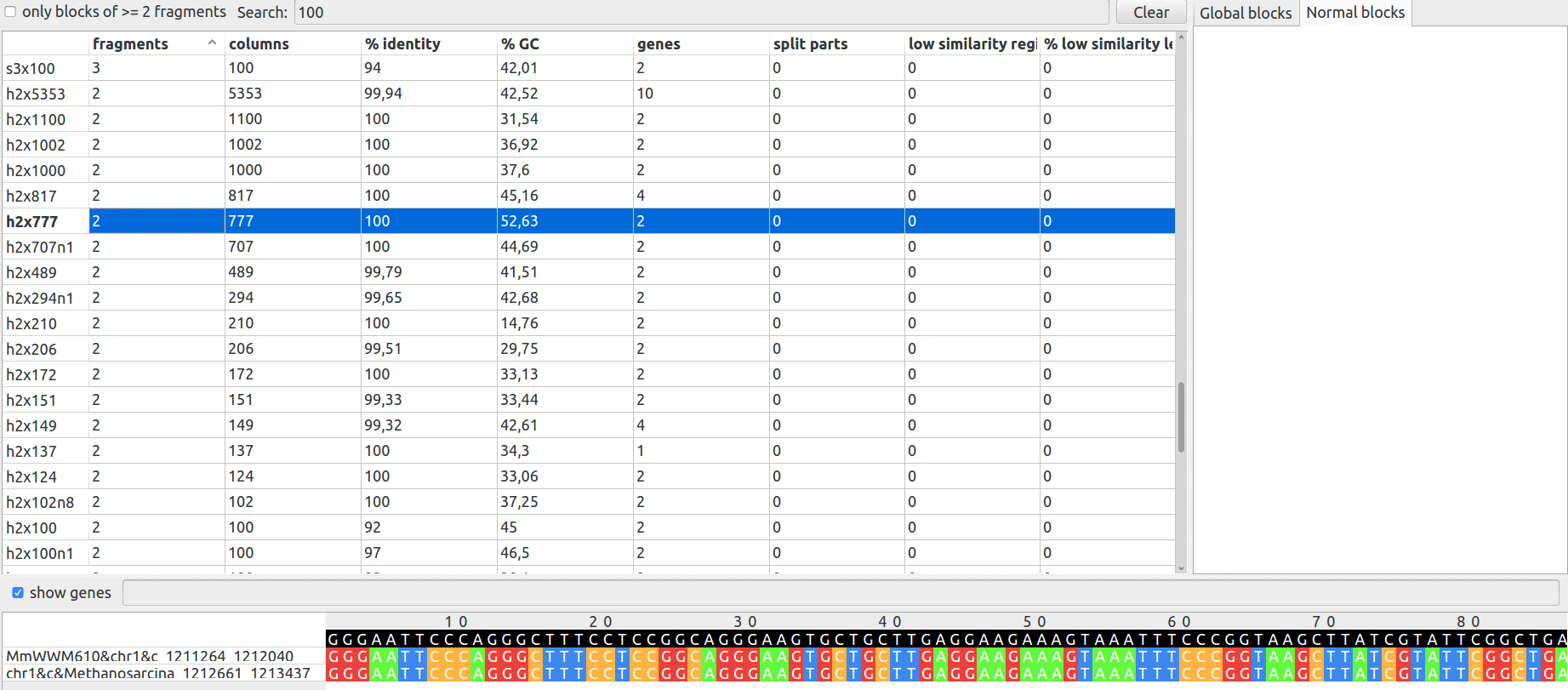
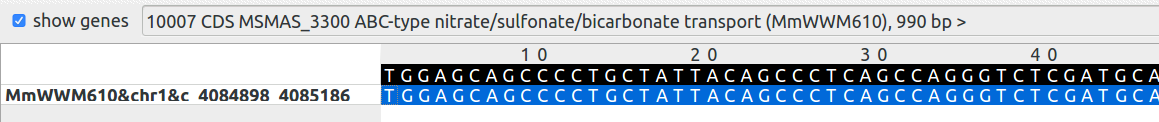
В базе данных UniProt была найдена последовательность ABC-type nitrate/sulfonate/bicarbonate transport system неродственной бактерии , обнаруженная в геноме Methanosarcina mazei WWM610. Показана на рисунке 16. В данном штамме было обнаружено больше всего инсерций. Самые интересные из них показаны ниже.
|
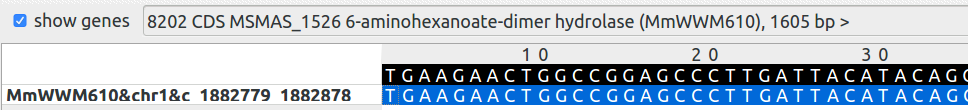
Была найдена последовательность 6-aminohexanoate-dimer hydrolase из класса гидролаз, катктализирующей реакцию гидролиза N-(6-aminohexanoyl)-6-aminohexanoate в 6-aminohexanoate.
|
Был найден фрагмент последовательности, кодирующей АТФ-зависимую ДНК геликазу, найденную только в первом штамме. Похоже, что произошла серия мутаций в этом гене, приведшая к несоответствию последовательностей.
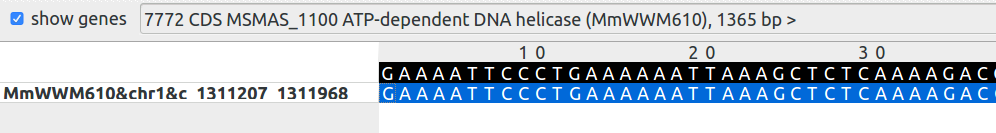
|
Для всех трёх геномов характерно наличие енолазы или фосфопируват гидратазы, относящейся к суперсемейству енолаз. Данный фермент является катализатором девятой реакции гликолиза, переводящей 2-Фосфоглицерат в фосфоенолпируват.
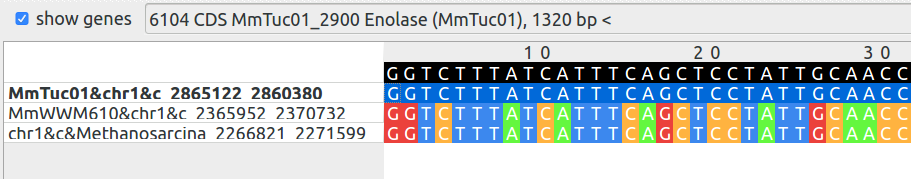
|
Также в трёх штаммах был обнаружен ген полифосфат киназы, катализирующей реакцию образования полифосфата из АТФ.
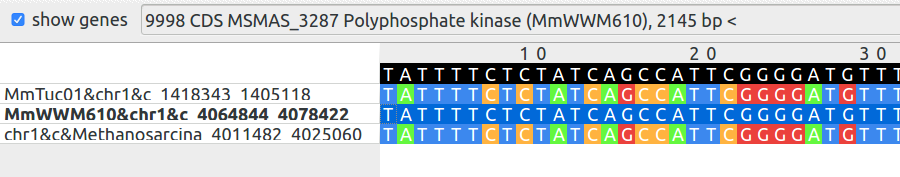
|
Был найден ген сорбитол дегидрогеназы, превращающей глюкозу в фруктозу.
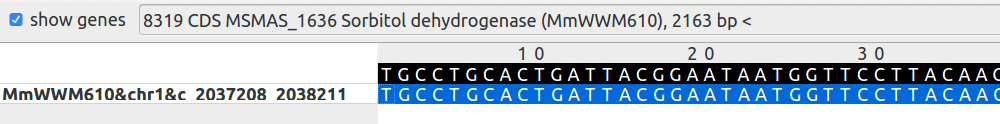
|
Также в трёх геномах присутствует Xaa-Pro aminopeptidase, катализирующая реакцию N-краевого расщепления пептидов.
|
