Чтение последовательностей по Сэнгеру
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
Упражнение 1
Прочитение последовательности ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру. Проблемы при чтении хроматограмм
Капиллярный секвенатор выдает файлы с хроматограммой и автоматически прочтённой последовательностью в формате .ab1. В данной работе анализировались два файла, соответствующих прочтению прямой и обратной цепочки ДНК.
Ссылки на исходные файлы .ab1
Прямое прочтение
Обратное прочтение
| Прочтение | Координаты 5'- конца | Координаты 3'- конца |
| Прямое | 1-31 | 690-714 |
| Обратное | 1-66 | 643-724 |
Хроматорграммы качественные, читаемые. Пики шума, в среднем, имеют малую высоту: 5% от сигнала при прямом и 10% при обратном прочтении. Обратное прочтение сильно зашумлено кравсителем или солями ближе к 600. Последовательности были избавлены от зашумлённых концов. Далее приводится нумерация относительно новых отредактированных последовательностей.
Ссылки на результат - последовательностьи в fasta формате с выделенными строчными буквами проблемными нуклеотидами и полиморфизмами
Прямое прочтение
Обратное прочтение
Таблица 2. Внесённые исправления
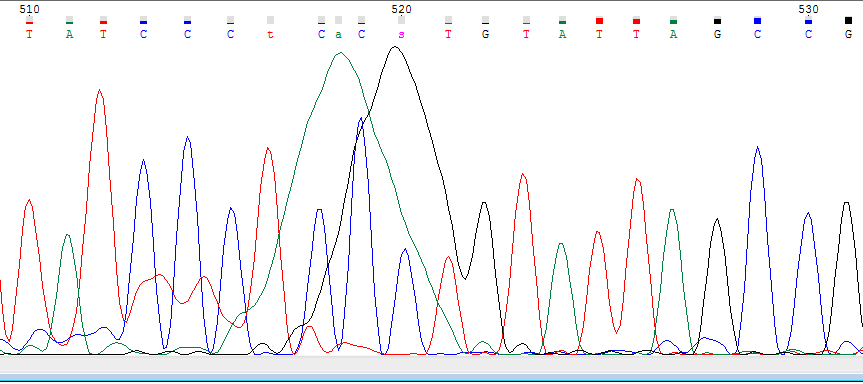
Прямое прочтение | На 38 позиции нуклеотид, не распознанный программой, был исправлен на k (то есть G или T), так как на хтоматограмме присутствуют два пика. По обратному прочтению можно восстановить, что в данной позиции находится тимин. | 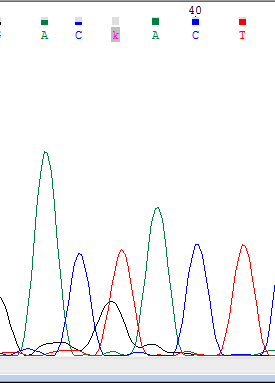
|
| Нуклеотид 642 не был прочитан однозначно. В этой позиции читаются два пика гуанина и аденина, поэтому был выбран IUPAC Code r. | 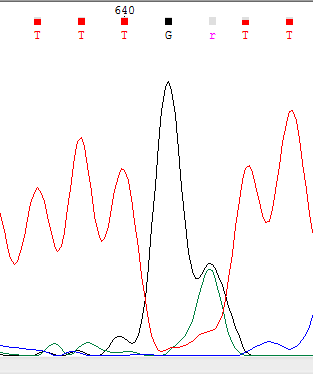
|
| Нуклеотиды 645 и 648 не были прочитаны однозначно. В этих позицииях читаются по два пика цитозина и тимина, поэтому в обоих случаях был выбран IUPAC Code y. Использовать комплементарную последовательность в данном случае не передставляется возможным, так как она оказывается сильно зашумлена и обрывается рядом с интересующим участком. | 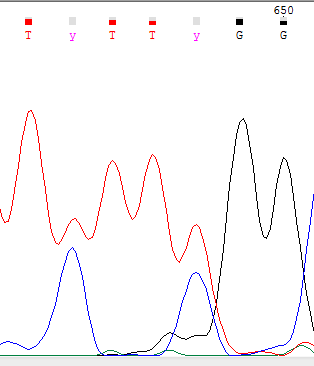
|
| Скорее всего,этот участок стоит рассматривать целиком. При движении по хроматоргамме видно, что некоторые группы пиков повторяются, что может быть вызвано ошибкой полимеразы на ранних раундах ПЦР. | 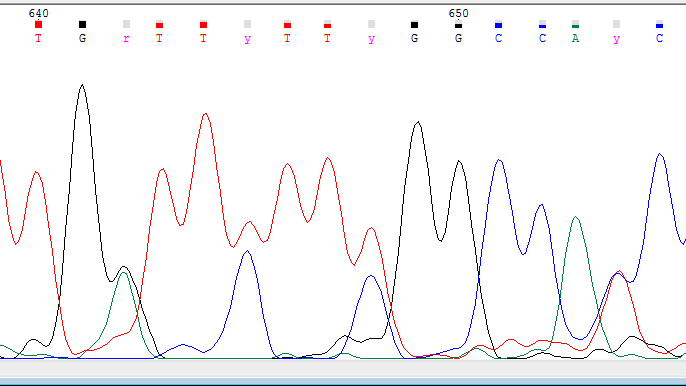
|
Обратное прочтение | |
| Для обратной последовательности на цитозин был исправлен 26 нуклеотид, а 36 на тимин. | 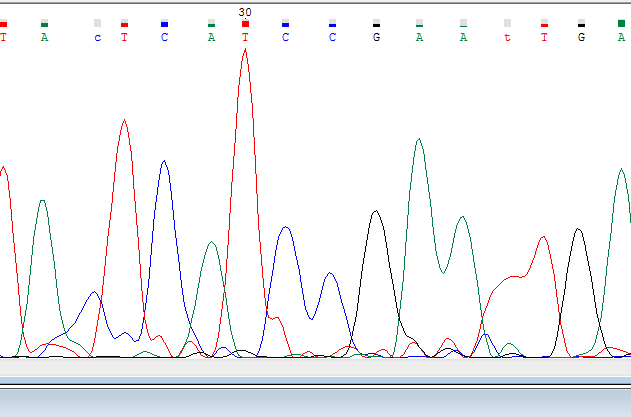
|
| 55 нуклеотид не был исправлен, но пик имеет изгиб с правой стороны, что может говорить о незначительном присутствии побочной ДНК, солей, о неровномерном движении в капиляре. | 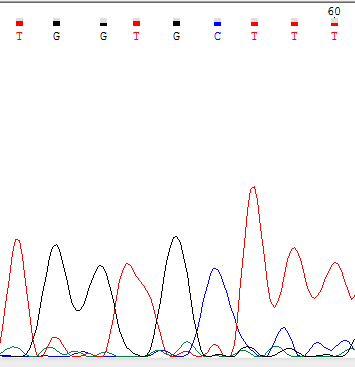
|
| 178 позиция, возможно, является полиморфизмом при обратном прочтении, однако при прямом однозначно определён аденин. | 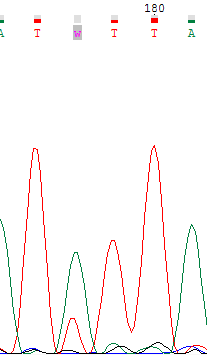
|
Упражнение 2.
Примеры нечитаемых фрагментов хроматограммы
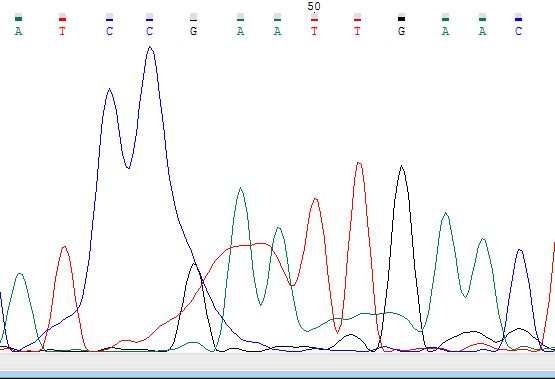
На хроматограмме есть несколько мест, загрязнённых солями или красителем, однако с них, с использованием обратного прочтения, чаще всего, удаётся прочесть последовательность (Рисунок 8, 9).
Однако на хроматограмме встречаются участки, расшифровать которые оказывается трудно. Рисунок 10 иллюстрирует пример такого участка. Вероятно, в ходе эксперимента в капиляре вместе с последовательностями ДНК разогнался флуорофор. Из рисунка 11 видно, что на последовательнось, сигнал от кторой. действительно, продолжает приниматься, наслаивается сигнал флуорофоров тимина, аденина и гуанина.
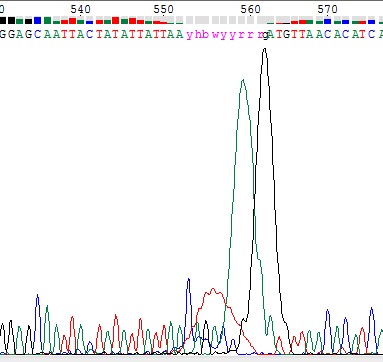
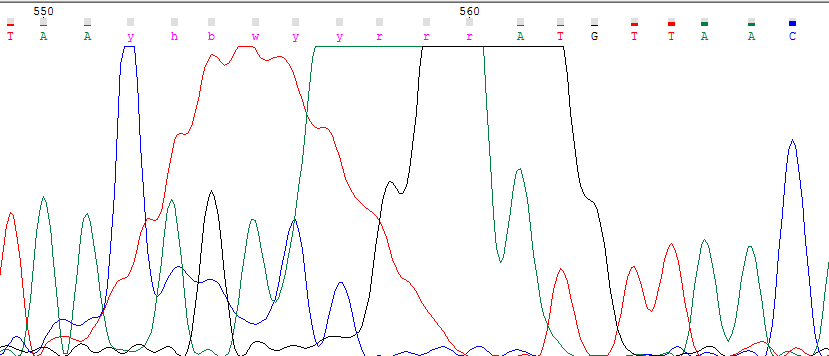
На основании проведённого анализа, была построена консенсусная последовательность.
Ссылки
IUPAC Code
Ссылка на проект Jalview
Прямое прочтение. Последовательность в Chromas
Обратное прочтение. Последовательность в Chromas
