Качество сборки генома эукариотического организма
 |
Рисунок 1. Dictyostelium discoideum [1].
|
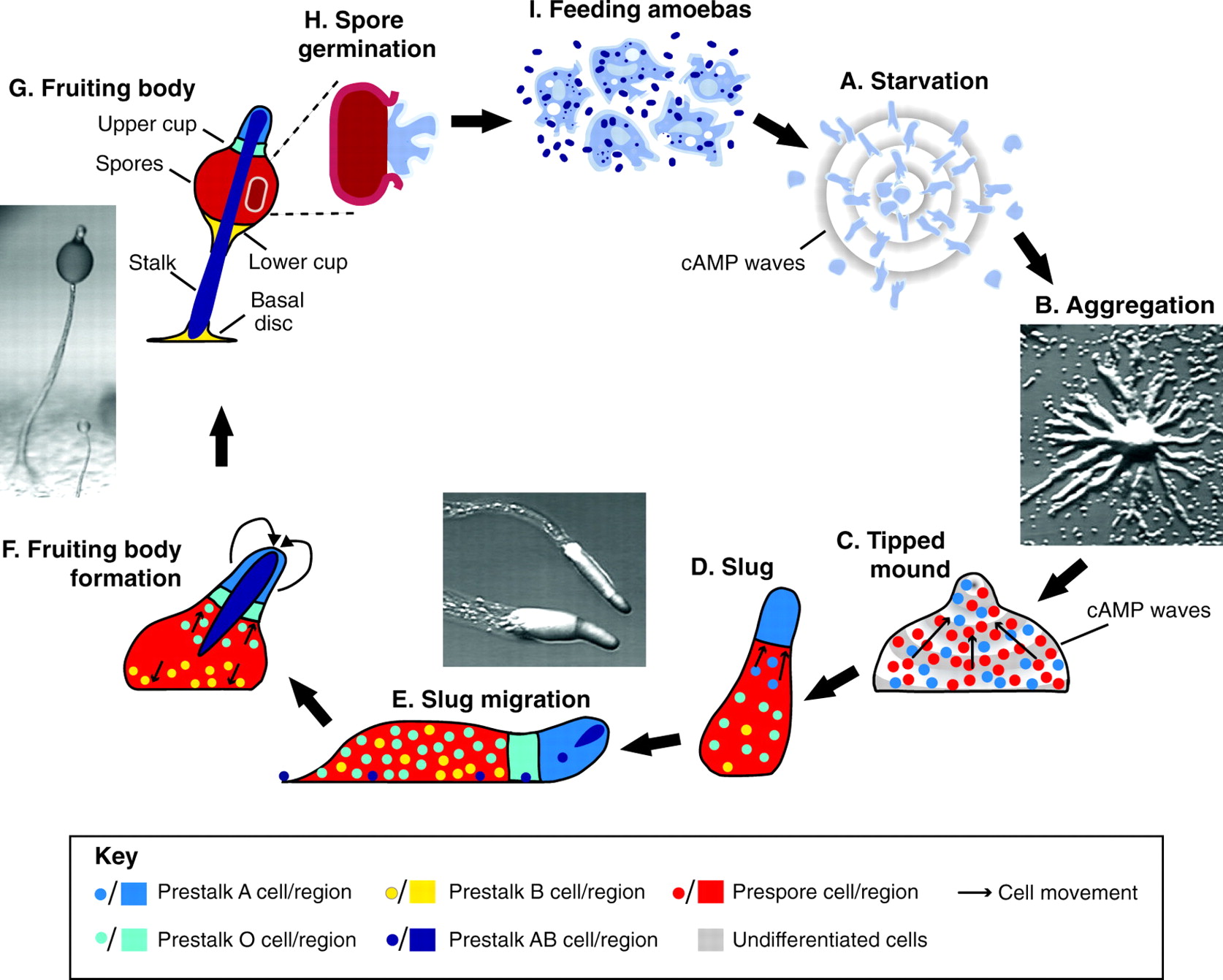 |
Рисунок 2. Жизненный цикл Dictyostelium discoideum [1].
|
Для выполнения практикума был выбран слизевик Dictyostelium discoideum (Рисунок 1), относящийся к типу Mycetozoa. Dictyostelium discoideum является модельным организмом
для изучения генетики и биологии развития.
Для него известна всего одна сборка, которая была опубликована
Northwestern University, dictyBase, Center for Genetic Medicine 6 августа 2014 года.
Для определения последовательности применялось полное геномное секвенирование методом дробовика (Whole chromosome shotgun (WCS) sequencing).
Dictyostelium discoideum имеет очень интересный жизненный цикл (Рисунок 2).
Большую часть времени диктиостелиум проводит в виде одиночных почвенных амёб, однако при определённых условиях амёбы
образуют подвижные агрегаты, а затем многоклеточные плодовые тела сложного строения.
В таблице 1 представлены результаты поиска сборок генома. Таблицы контигом найденной сборки можно найти
по ссылке. Сиквенс первой хромосомы также можно найти по ссылке
ссылке.
Таблица 1. Результаты поиска сборок генома.
| Организм: название |
Dictyostelium discoideum |
| Технология секвенирования |
Whole chromosome shotgun (WCS) sequencing |
| Число находок |
1 |
| BioSample: |
SAMN02953606 |
| GenBank assembly accession: |
GCA_000004695.1 |
| RefSeq assembly accession: |
GCF_000004695.1 |
| Покрытие
|
8.3x |
| Число контигов |
261 |
| N50
|
341,816
|
| L50
|
20
|
| Число скэффолдов
|
41
|
| Genome coverage: |
8.3x |
|
Описание ключей, используемых в таблицах особенностей
В таблице 2 представлено семь ключей, использующихся в поле "особенности" баз данных. [4]
Таблица 2. Ключи, использующиеся в поле "особенности" баз данных.
| Ключ: название (англ.) |
Ключ: описание (рус.) |
Пример из записи |
| exon |
Участок генома, кодирующий сплайсирующийся участок mRNA, rRNA или tRNA; может содержать 5' некодирующие области, белок кодирующие последовательности и 3' некодирующие области.
|
exon 4179986..4180218
/product="protein kinase-like protein"
/inference="non-experimental evidence, no additional
details recorded"
/note="CDS is reported in Acc# AP000375;
gene_id:MGH6.22"
/number=1
https://www.ncbi.nlm.nih.gov/nuccore/BA000014.8
|
| gene |
Имеющий название и представляющий интерес для изучения участок последовательности ДНК.
|
gene complement(<4090123..>4090589)
/gene="MJM20.1"
/note="similar to oxidoreductases"
https://www.ncbi.nlm.nih.gov/nuccore/BA000014.8 |
| ncRNA |
Не кодирующий белок ген, рРНК или тРНК, транскрибирующийся в функционирующую РНК последовательность.
|
ncRNA 3475..3524
/ncRNA_class="other"
/locus_tag="SM2011_c06000"
/product="putative ncRNA"
/note="corresponds to SMc06000;
based on oriented RNAseq data"
https://www.ncbi.nlm.nih.gov/nuccore/CP004140.1 |
| mRNA |
Информационная РНК, содержащая 5' нетранслируемую область, кодирующая экзонную или зрелую белковую последовательность и содержащая 3' нетранслируемую область.
|
mRNA complement(<1965195..>1965572)
/gene="F24P17.17"
https://www.ncbi.nlm.nih.gov/nuccore/BA000014.8 |
| CDS |
Зрелая не содержащая стоп кодонов белок кодирующая последовательность, содержащая информацию об аминокислотной последовательности пептидного или
белкового продукта с последующими посттрансляционными модификациями.
|
CDS complement(4090123..4090589)
/gene="MJM20.1"
/note="hypothetical protein"
/codon_start=1
/protein_id="AAF34826.1"
https://www.ncbi.nlm.nih.gov/nuccore/BA000014.8 |
| polyA_site |
Сайт последовательности РНК, к которому присоединяются адениновые остатки в процессе посттранскрипционного полиаденилирования.
|
https://www.ncbi.nlm.nih.gov/nuccore/Z24749.1 |
| mat_peptide |
Зрелая пептид или белок кодирующая последовательность, несущая информацию о зрелом или конечном пептиде/белке, получающемся при посттрансляционных модификациях. Разметка не включает в себя позиции стоп-кодонов.
|
mat_peptide 105..383
/product="propeptide"
https://www.ncbi.nlm.nih.gov/nuccore/KY785485.1 |
|
Описание массового геномного проекта
 |
Рисунок 3. Genomics England [6].
|
Для описания был выбран проект Genomics England [6]. Целью которого является секвенирование геномов 100 000 людей и их семей с редкими заболеваниями.
Проект был иницирован Английским департаментом здравоохранения, которому принадлежит Genomics England в августе 2014; В проект было инвестировано
27 миллионов фунтов стерлингов. Планируется отсеквенировать 100 000 геномов. Дата завершения ориентировочно назначена на конц 2018 года.
К настоящему моменту отсеквенировано более 36 083 генома.
Последняя статья
Таблица митохондриальных генов Saccharomyces cerevisiae
 |
Рисунок 4. Saccharomyces cerevisiae [2].
|
Для поиска были выбраны пекарские дрожжи (Saccharomyces cerevisiae) - вид одноклеточных микроскопических аскомицет,
широко используемый в производстве алкогольной и хлебопекарной продукции, а также в научных исследованиях. Удобны для моделирования процессов и биохимических систем эукариот.
С результатами поиска можно ознакомиться в таблице 3. Также была скачана таблица с информацией о всех белках, закодированных в митохондриальной ДНК дрожжей.
С ней можно ознакомиться, перейдя по
ссылке.
Таблица 3. Результаты поиска митохондриальных геномов.
| Запрос |
"Saccharomyces cerevisiae"[Organism] AND ("Complete genome"[Title] OR "Complete sequence"[Title]) AND "Mitochondrion"[Title] AND "RefSeq"[Keyword] |
| Число находок |
2 |
| АС выбранной записи |
NC_001224 |
| Число п. н. в митохондриальном геноме |
85779
|
| Число генов белков |
46
|
| Число генов РНК |
27
|
|
Таблица размеров геномов
Ознакомиться с таблицей Вы можете, перейдя по ссылке.
Таблица 4. Размеры геномов
| Название организма |
Минимальный размер (Mb) |
Типичный размер (Mb) |
Максимальный размер (Mb) |
| Вироиды |
0.000246 (Coconut cadang-cadang viroid) |
0,000360 |
0.000434 (Apple hammerhead viroid-like circular RNA) |
| Вирусы |
0.00022 (Rice yellow mottle virus satellite) |
0,003 |
2.47387 (Pandoravirus salinus) |
| Бактерии |
0.104827 (Cloacimonetes bacterium JGI 0000039-I11) |
1,08943 |
9.99787 (Lentzea guizhouensis) |
| Археи |
0.137797 (candidate divison MSBL1 archaeon SCGC-AAA385M02) |
1,278 |
6.4512 (uncultured marine crenarchaeote 'Gulf of Maine') |
| Эукариоты |
0.245805 (Amana erythronioides) |
100 |
10495 (Triticum dicoccoides) |
|
Ссылки
ID - identification (begins each entry; 1 per entry)
AC - accession number (>=1 per entry)
PR - project identifier (0 or 1 per entry)
DT - date (2 per entry)
DE - description (>=1 per entry)
KW - keyword (>=1 per entry)
OS - organism species (>=1 per entry)
OC - organism classification (>=1 per entry)
OG - organelle (0 or 1 per entry)
RN - reference number (>=1 per entry)
RC - reference comment (>=0 per entry)
RP - reference positions (>=1 per entry)
RX - reference cross-reference (>=0 per entry)
RG - reference group (>=0 per entry)
RA - reference author(s) (>=0 per entry)
RT - reference title (>=1 per entry)
RL - reference location (>=1 per entry)
DR - database cross-reference (>=0 per entry)
CC - comments or notes (>=0 per entry)
AH - assembly header (0 or 1 per entry)
AS - assembly information (0 or >=1 per entry)
FH - feature table header (2 per entry)
FT - feature table data (>=2 per entry)
XX - spacer line (many per entry)
SQ - sequence header (1 per entry)
CO - contig/construct line (0 or >=1 per entry)
bb - (blanks) sequence data (>=1 per entry)
// - termination line (ends each entry; 1 per entry)




