Поиск по сходству (нуклеотидный blast)
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
Определение таксономии и функции прочтённой нуклеотидной последовательности
Для выполнения практикума была взята последовательность, полученная на капилярном секвенаторе
Беломорской биостанции и откорректированная в предыдущем практикуме.
Для определения предполагаемого уровня таксономии была использована часть програмного пакета BLAST - алгоритм
BLASTN.
Программы пакета принимаеют на вход последовательность или её чать и осуществляют поиск по всем имеющимся в базе данным последовательностям (если другое не выбрано),
выравнивая одни против других. Выравнивание начинается с коротких затравок по 7 - 13 позиций. В программе BLASTN осуществляется выравнивание нуклеотидных последовательностей
против нуклеотидных.
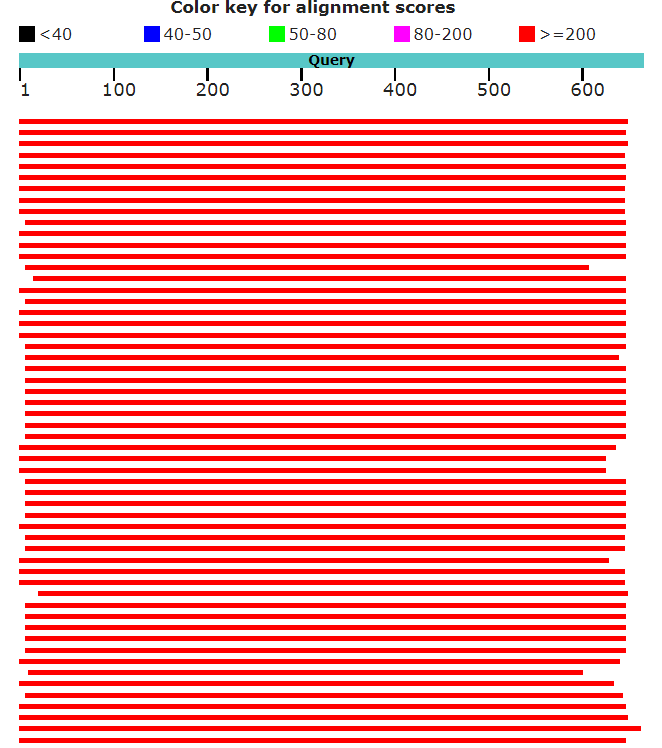
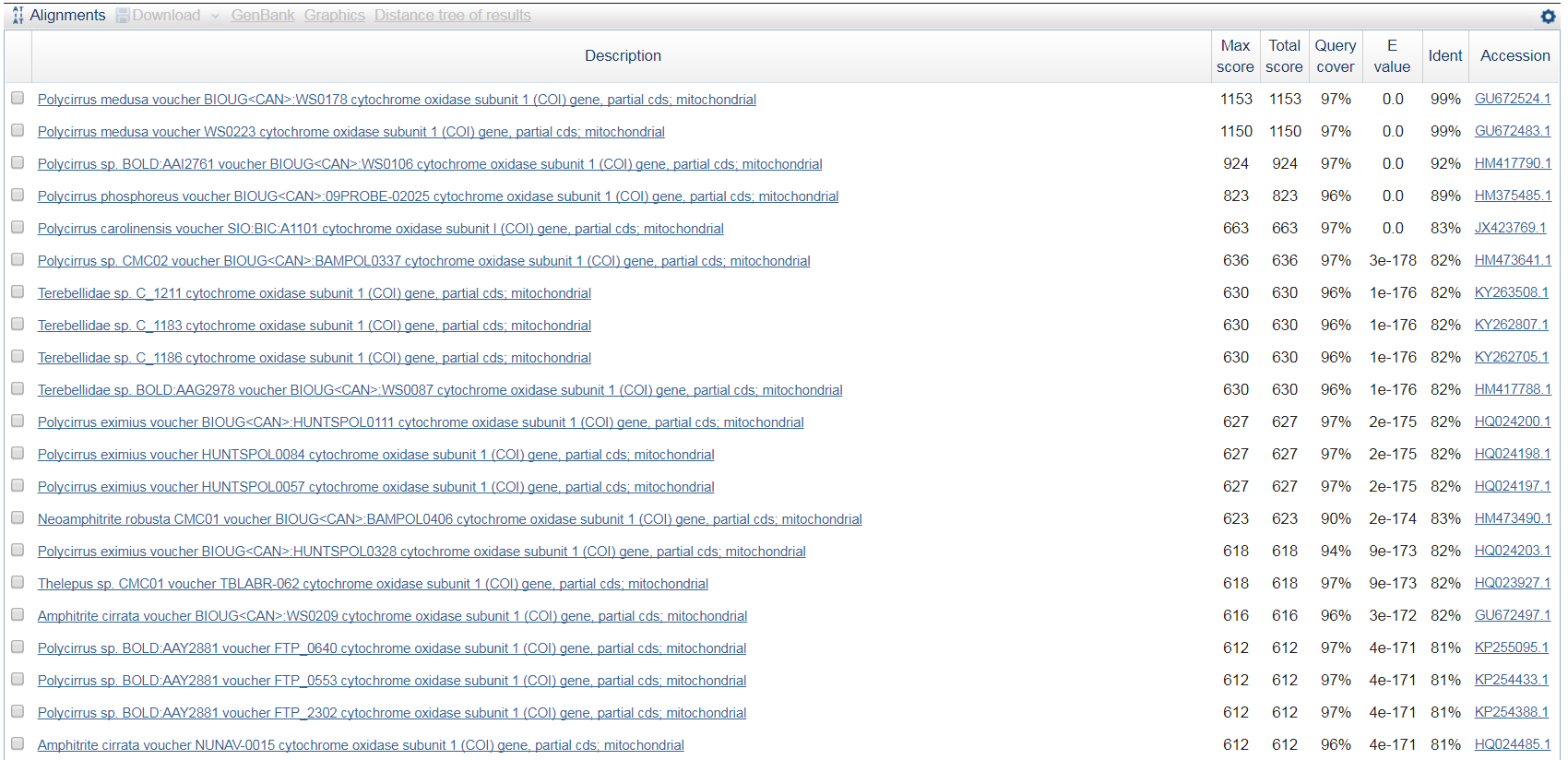
Рисунок 1 демонстрирует результат работы программы. Можно предположить, что организм, из которого был получен и и секвенирован образец ДНК, относится к виду
Polycirrus medusa, относящемуся наравне с другими 14 видами, известными на данный момент,
к роду небольших (150 мм) многощетинковых морских червей Polycirrus из семейства Terebellidae, описанного Карлом Линнеем в 1767 году.
Полная характеристика таксономического положения представлена в таблице 1.
Таксономия была определена таким образом, поскольку три первых находки с лучшими максимальным весом (1153), покрытием (97%), минимальным E-value (0.0), максимальной
идентичностью (99%) относятся к роду Polycirrus, а две первых к виду Polycirrus medusa.
Данная последовательность с высокой вероятностью кодирует cds ген субъединицы цитохромоксидазы 1 с идентификатором GU672524.1. Данная субъединица является ключевой при сборке цитохром оксидазы с.
Рисунок 0. Lanice conchilega - представитель семейства Terebellidae

|
Рисунок 1. Результаты поиска BLASTN
|
Рисунок 2. Результаты поиска BLASTN
|
Таблица 1. Характеристика таксономического положения Polycirrus medusa
| Таксономическое положение |
Таксономия |
| species |
Polycirrus medusa |
| genus |
Polycirrus |
| family |
Terebellidae |
| order |
Terebellida |
| subclass |
Scolecida |
| class |
Polychaeta |
| phylum |
Annelida |
|
Сравние списков находок по нуклеотидной последовательности 3-я разными алгоритмами blast
Производился поиск с использованием той же последовательности с использованием трёх алгоритмов: blastn, discontiguous megablast, megablast. Параметры поиска: Поиск производился внутри семейства Terebellidae (taxid:32261)
с исключением рода Polycirrus (taxid:516039). Использовалась отсечка по E-value: 1e-150. Длина слова варьировалась в зависимости от алгоритма поиска, результаты которого можно найти
в таблице 2.
Результаты поиска blastn
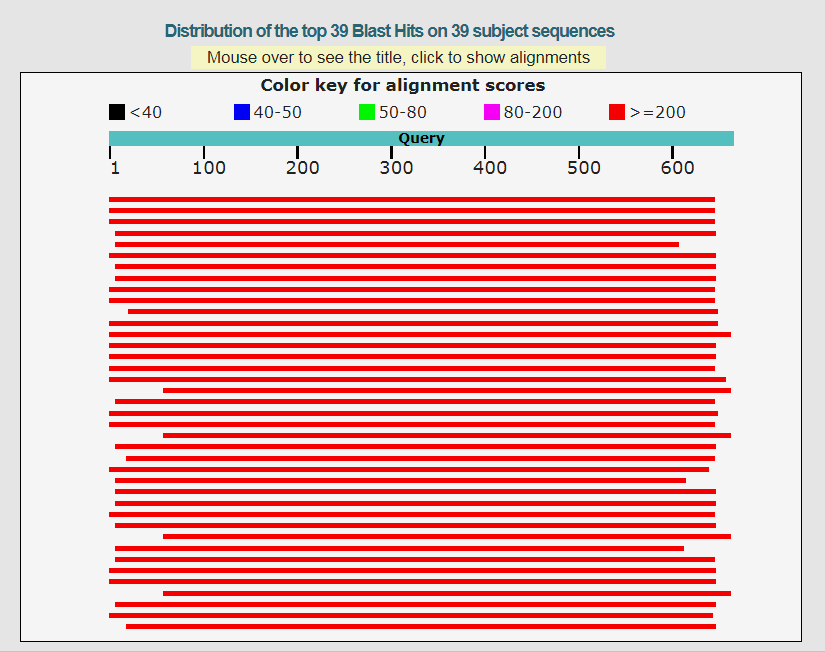
Рисунок 3. Результаты поиска blastn
|
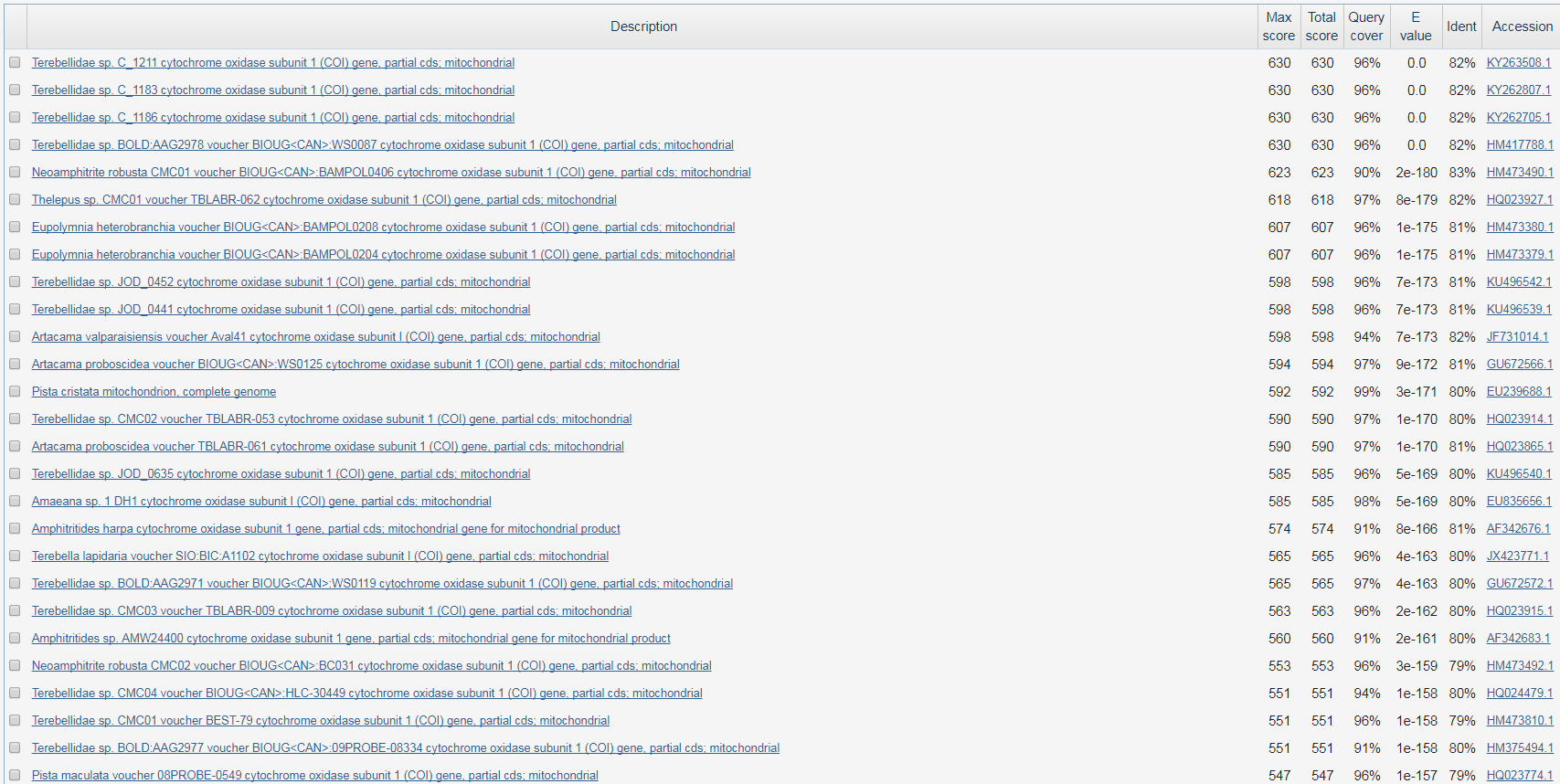
Рисунок 4. Результаты поиска blastn
|
Результаты поиска discontiguous megablast
Рисунок 5. Результаты поиска discontiguous megablast
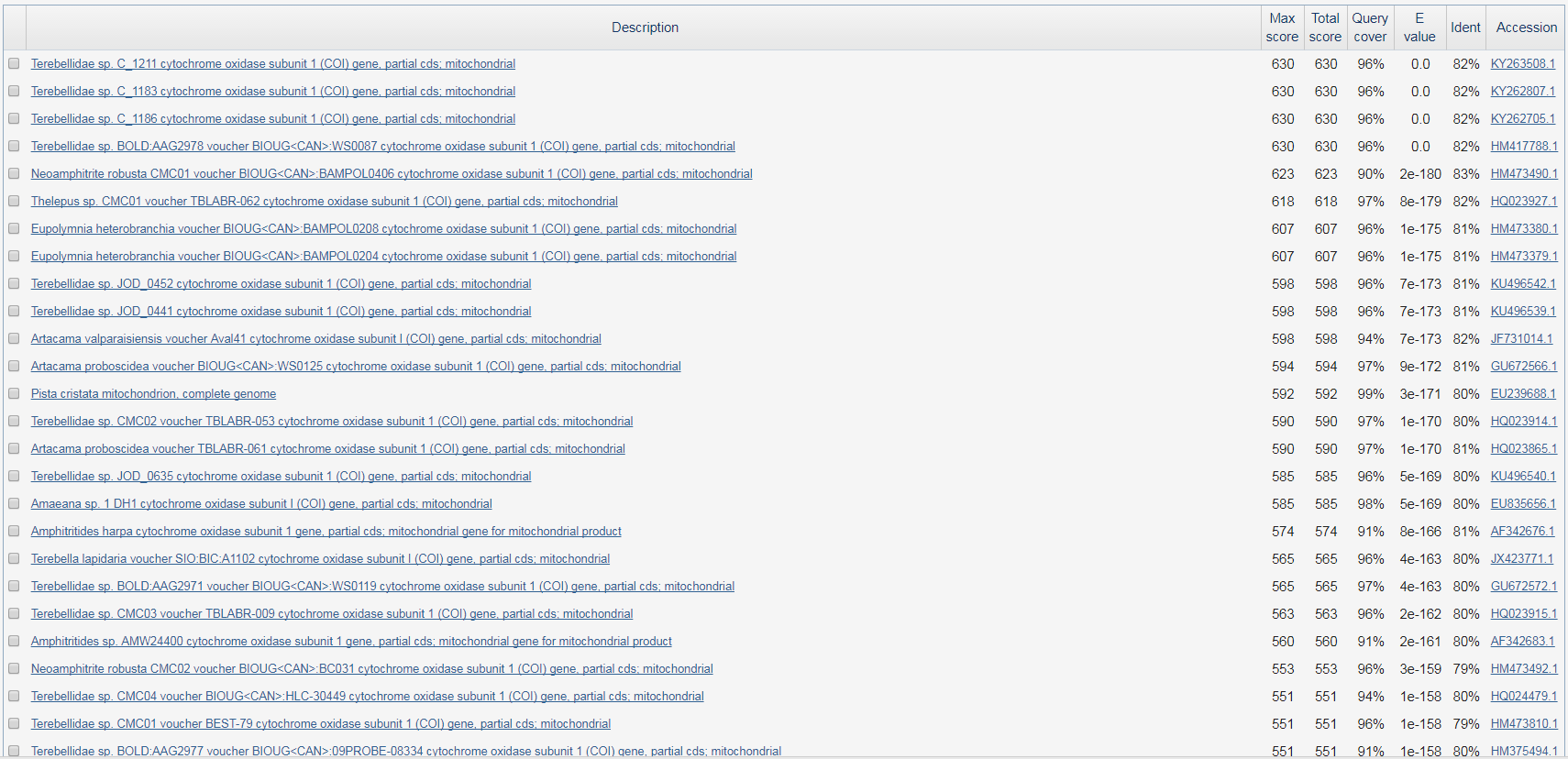
|
Результаты поиска megablast
Рисунок 6. Результаты поиска megablast
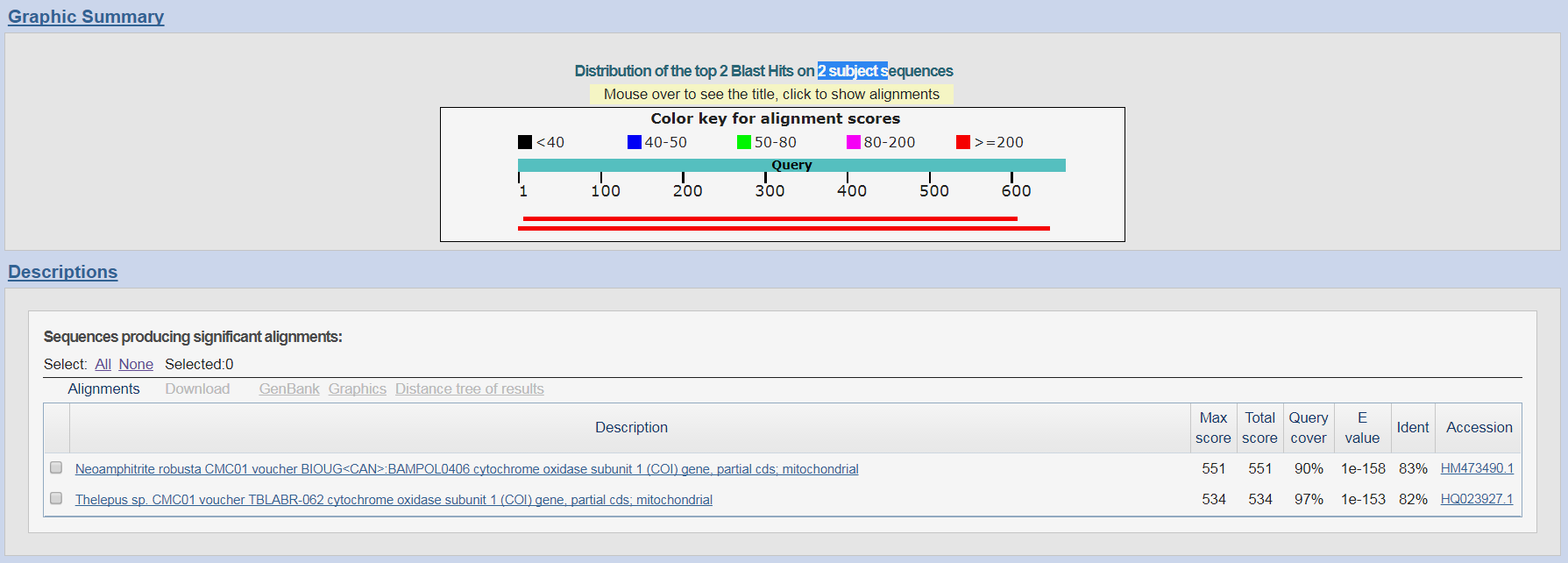
|
Таблица 2. Результаты поиска алгоритмов BLAST
| Алгоритм |
Длина слова |
Число находок |
| blastn |
11 |
39 |
| discontiguous megablast |
11 |
39 |
| megablast |
28 |
2 |
|
Поиск с использованием разных программ дал различные результаты. Программа blastn, как и discontiguous megablast, дала максимальное число находок (39), тогда как программа megablast
нашла всего лишь 2 последовательности. Данное различие можно объяснить неодинаковыми стратегиями поиска, как то: megablast ищет длинные, по 28 нуклеотидов в длинну, последовательности,
тогда как blastn и discontiguous megablast осуществляют поиск по подстрокам длины 11. Discontiguous megablast ищет сходство между последовательностями с низкой идентичностью и ожидалось,
что результат поиска данной программы будет самым разнообразным. Три программы нашли две одинаковые последовательности (HM473490.1, HQ023927.1). 37 последовательностей, найденных первыми алгоритами были пропущены треьим, так как,
скорее всего, они не содержали 28 консервативных нуклеотида подряд. Эти 37 последовательностей были скачаны в файл .
Проверка наличия гомологов трех белков в геноме одного организма
Для выполнения задания была взята сборка генома Amoeboaphelidium protococcarum - представителя таксона Афелд, родственного грибам из группы опистоконт.
Афелды являются внутриклеточными паразитами или паразитоидами планктонных водорослей.
С помощью локальной версии blast - blast 2.2.31+ был произведён поиск против локально созданной из сборки базы данных. Для поиска была выбрана программа tblastn, обратно
транслирующая последовательность белка в последовательность нуклеотидов ДНК и производящая поиск схожих последовательностей по этим данным. Результаты поиска приведены в таблице 3.
Для выполнения задачи из банка данных были скачаны:
- HSP71_YEAST - шаперон HSP71, белок теплового шока;
- TERT_SCHPO - теломераза,восстанавливающая длину хромосомы при репликации; имеется у большинства (но не всех) эукариот
- PRPC_EMENI - митохондриальная цитратсинтаза
С полными результатами поиска можно ознакомиться, перейдя по ссылке или открыв таблицу Exсel. Жёлтым цветом выделены лучшие находки из
отфильтрованных по E-value.
Таблица 3. Результаты поиска локального BLAST +
| Идентификатор белка |
Идентификатор скэффолда |
Длинна выравнивания |
E-value |
Идентичность |
Заключение |
| sp|P10591.4|HSP71_YEAST |
scaffold-199 |
609 |
0 |
78,98 |
Гомолог |
| sp|O13339.1|TERT_SCHPO |
scaffold-17 |
491 |
1E-023 |
25,05 |
Не гомолог |
| sp|Q9TEM3.1|PRPC_EMENI |
scaffold-693 |
376 |
6E-121 |
56,38 |
Предположительно гомолог |
|
Из полученных результатов видно, что последовательность белка HSP71_YEAST, скорее всего, является
гомологичной белкам из генома Amoeboaphelidium protococcarum. Она имеет низкий E-value, высокую идентичность, длинну. Последовательности белков TERT_SCHPO и PRPC_EMENI нельзя
с той же уверенностью назвать гомологичными: первый имеет низкую идентичность при сравнительно повышенном E-value, а второй имеет средние показатели, что наравне с небольшим количеством
находок может говорить о гомологии доменов.
Поиск гена белка, закодированного в одном скэффолде Amoeboaphelidium protococcarum
Из сборки генома Amoeboaphelidium protococcarum был извлечён скэффолд scaffold-670 с длинной 50106 и GC составом 40.31%. С помощью программы blastx было произведено
несколько пробных поисков и выяснено, что наиболее богатым по доменному составу является область от 0 до 2000 нуклеотидов. Из рисунков 7 и 8 видно, что участок с 1 до 800 нуклеотид
является, по-видимому, последовательностью потенциальной гомологии. Лучшей находкой является транспозаза - фермент, связывающий одноцепочечную ДНК и встраивающий последнюю
в геномную ДНК.
Выбран именно этот ген, так как он является наиболее правдоподобным и имеет большую, пусть и недостаточную, поддержку:
относительно низкий E-value, покрытие ~50%. Видимо, данная последовательность из сборки не содержит функциональных последовательностей или содержит их части.
Рисунок 7. Результаты поиска blastx
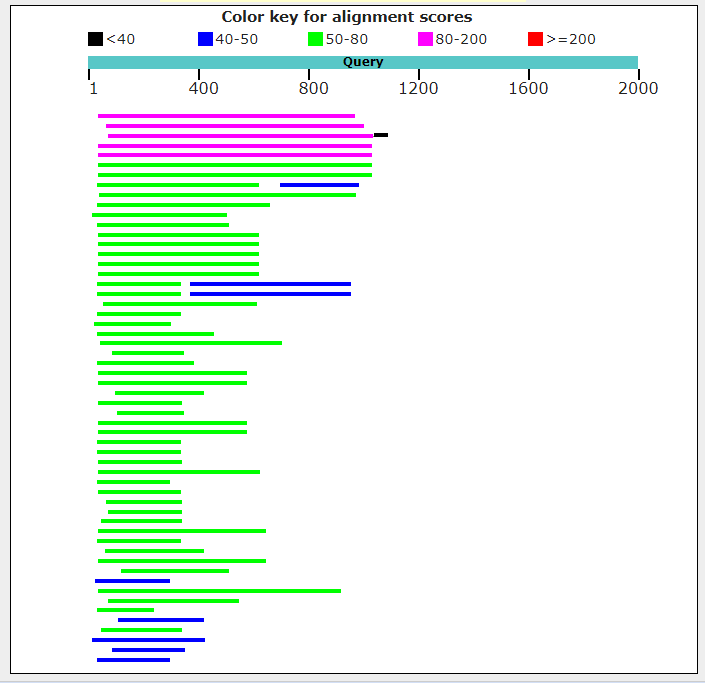
|
Рисунок 8. Результаты поиска blastx
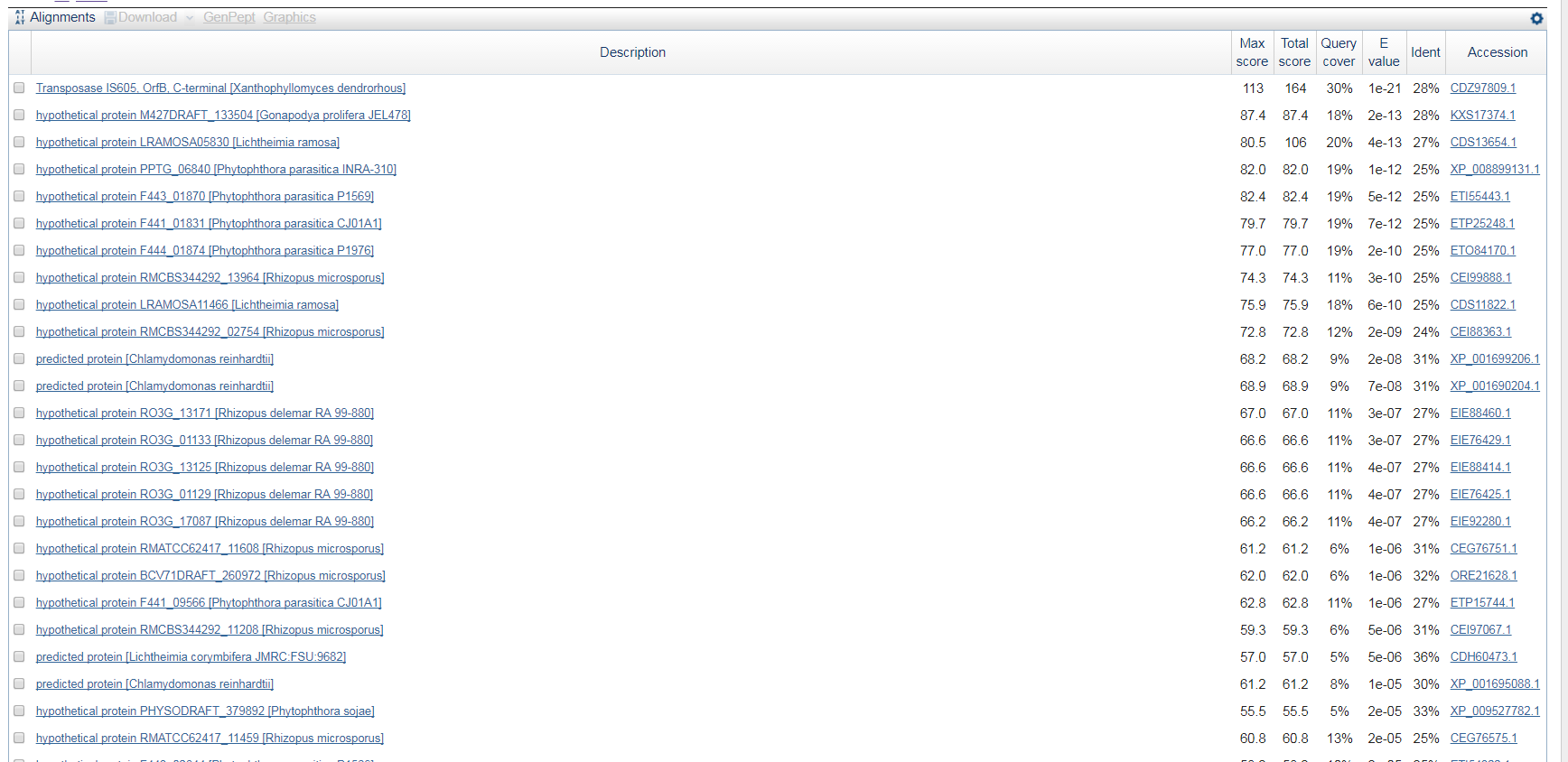
|
Классификация геномов родственных вирусов по сходству последовательностей
Была взята последовательность генома вируса, изучавшегося в первом семестре
Alternaria brassicicola endornavirus









