Реконструкция филогении. Дополнительное задание
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
Построение дерева выбранных организмов
В дополнительном задании мы сравнили работу программ пакета PHYLIP и программы Mega 7. Для анализа использованись последовательности, полученные в практикуме и сохранённые в файл RL1_aligned.fasta.
Для построения филогенетических деревьев был использован пакет PHYLIP.
Сначала для получения таблицы с рассчитанными расстояниями между последователностями белков была использована программа fprotdist. Для построения матрицы можно использовать разные методы, задающиеся параметром -method
Команда: fprotdist RL1_aligned.fasta
Выдача:
Protein distance algorithm Phylip distance matrix output file [rl1_aligned.fprotdist]: Computing distances: RHIEC RHOS4 . NEIMA .. HAEIN ... PROMH .... ECOLI ..... YERPE ...... Output written to file "rl1_aligned.fprotdist" Done.После с помощью программы fneighbor, использующей полученную матрицу расстояний,с помощью алгоритма Neighbor-Joining было построено филогенетическое дерево.
Программа принимает на вход разные типы входных матриц, достыпные по параметру -matrixtype {s -квадратная, u - нижняя треугольная и l - верхняя треугольная).
Также можно задать тип построения дерева (выбрать алгоритм) (-treetype [n (Neighbor-joining); u (UPGMA))]), рисовать построенное дерево в файл (-trout [name of file]).
Команда: fneighbor -datafile rl1_aligned.fprotdist
Выдача:
Phylogenies from distance matrix by N-J or UPGMA method Phylip neighbor program output file [rl1_aligned.fneighbor]: Cycle 4: species 1 ( 0.17484) joins species 2 ( 0.18669) Cycle 3: node 1 ( 0.27134) joins species 3 ( 0.16005) Cycle 2: node 1 ( 0.15360) joins species 4 ( 0.08000) Cycle 1: node 1 ( 0.04332) joins species 6 ( 0.03341) last cycle: node 1 ( 0.00878) joins species 5 ( 0.05377) joins species 7 ( 0.04688) Output written on file "rl1_aligned.fneighbor" Tree written on file "rl1_aligned.treefile" Done.
Получена скобочная формула
(RHOS4:0.18669,(NEIMA:0.16005,(HAEIN:0.08000,(ECOLI:0.03341, (PROMH:0.05377,YERPE:0.04688):0.00878):0.04332):0.15360):0.27134,RHIEC:0.17484);
Команда: fneighbor -datafile rl1_aligned.fprotdist -trout tree_out.txt
Выдача:
Neighbor-Joining/UPGMA method version 3.69.650 7 Populations Neighbor-joining method Negative branch lengths allowed +----------RHOS4 ! ! +---------NEIMA 1---------------2 ! ! +----HAEIN ! +--------3 ! ! +-ECOLI ! +--4 ! ! +--PROMH ! +-5 ! +-YERPE ! +---------RHIEC remember: this is an unrooted tree! Between And Length ------- --- ------ 1 RHOS4 0.18669 1 2 0.27134 2 NEIMA 0.16005 2 3 0.15360 3 HAEIN 0.08000 3 4 0.04332 4 ECOLI 0.03341 4 5 0.00878 5 PROMH 0.05377 5 YERPE 0.04688 1 RHIEC 0.17484
Следующим шагом является прорисовка изображения дерева с помощью программы fdrawtree. Программа получает на вход скобочную формулу дерева, а выдаёт изображение дерева в формате .ps
Команда: fdrawtree -intreefile rl1_aligned.treefile -plotfile rl1_aligned.ps
Выдача:
Plots an unrooted tree diagram DRAWTREE from PHYLIP version 3.69.650 Reading tree ... Tree has been read. Loading the font ... Font loaded. Writing plot file ... Plot written to file "rl1_aligned.ps" Done.Можно получить файл с рисунком дерева в формате .pdf, что может быть удобно для публиакации результата и создания автоматических отчётов.
Команда: ps2pdf rl1_aligned.ps
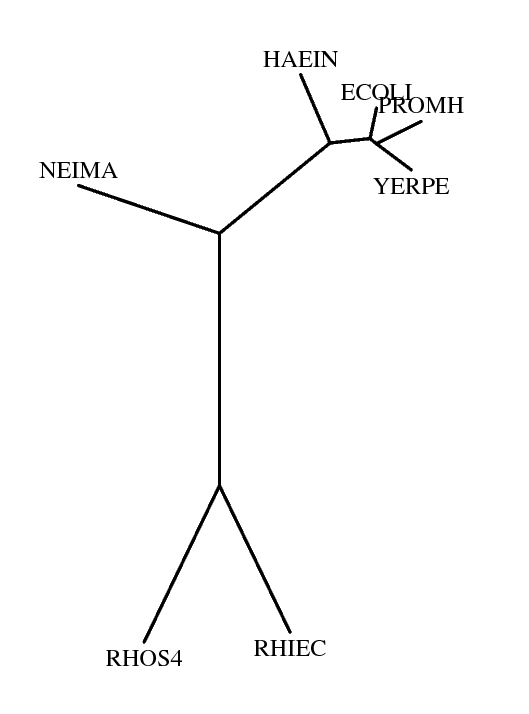 |
Для изображения дерева в виде дендрограммы можно использовать программу fdrawgram. Для избавления от надоедливого оконного режима используйте -previewer n.
Команда: fdrawgram -intreefile rl1_aligned.treefile -plotfile rl1_aligned.fdrawgram -auto -previewer n
Выдача:
DRAWGRAM from PHYLIP version 3.69.650 Reading tree ... Tree has been read. Loading the font .... Font loaded. Writing plot file ... Plot written to file "rl1_aligned.fdrawgram" Done.
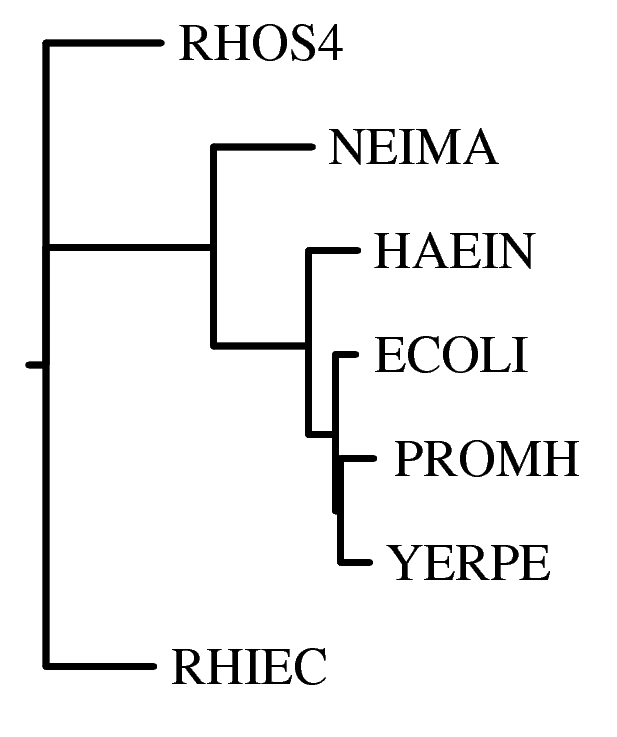 |
Сравнение работы программ пакета PHYLIP и программы Mega 7
Для сравнения результатов работы с помощью обоих программ были построены аналогичные деревья.
 |
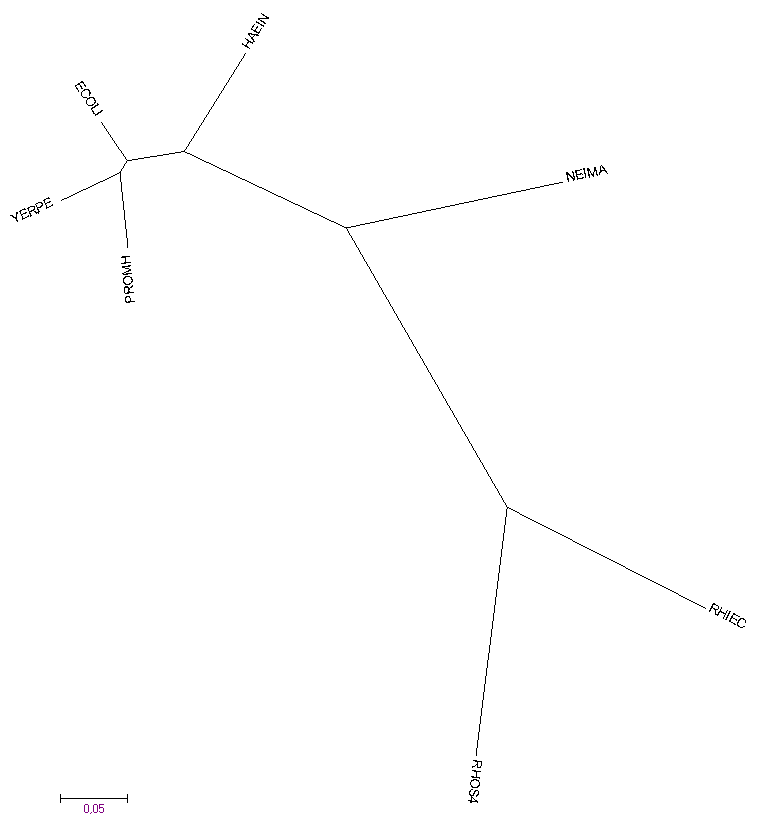 |
 |
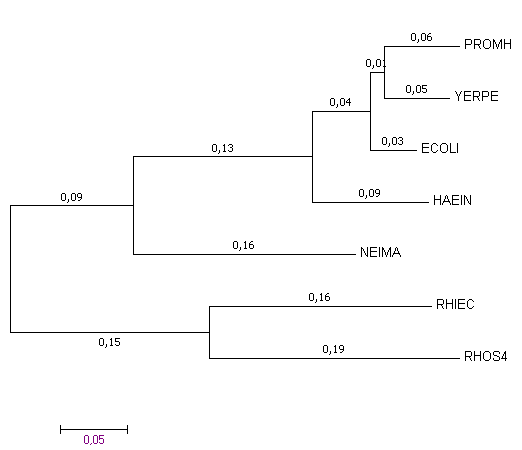 |
Из рисунков видно, что топологически деревья аналогичны. PHYLIP строит укоренённую дендрограмму приблизительно в центре. Можно попросить Mega укоренить дерево где-то в центре.
Также мне удалось освоить построение графиков в R, и расскраску исследуемых ветвей. Это достаточно увлекательно, и необходимо для моей научной работы. С некоторыми результатами можно ознакомиться ниже.
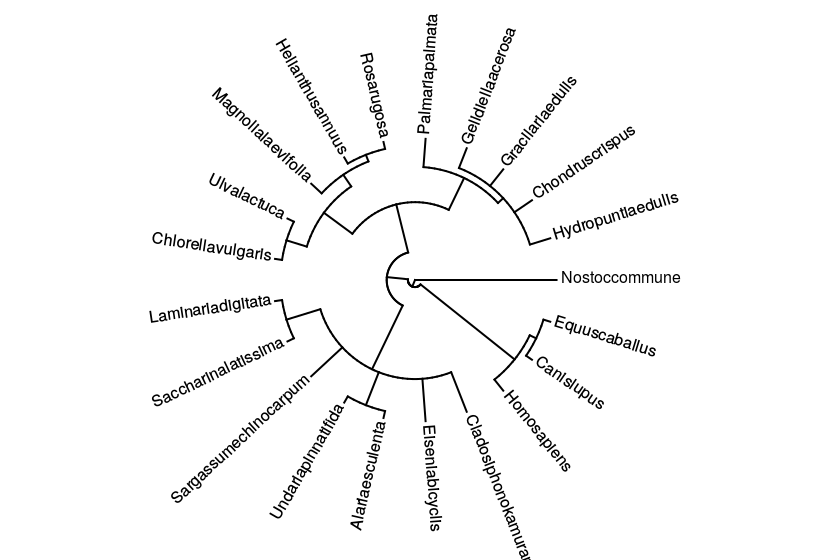 |
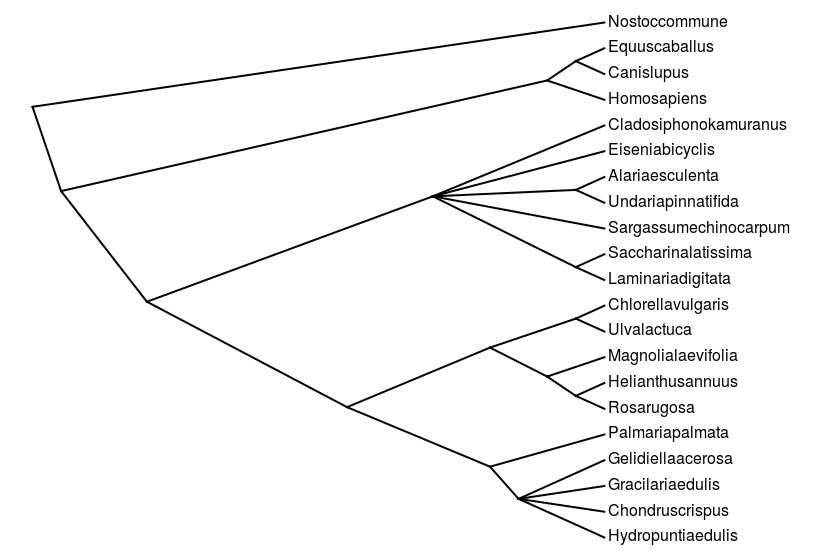 |
