Укоренение и бутстрэп
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
Построение и анализ дерева, содержащего паралоги
При выполнении практикума был проведён поиск гомологов белка CLPX_ECOLI - АТФ-зависимой Clp протеазы.
Родственные белки искались в протеомах бактерий, отобранных в предыдущем практикуме (Представлены в таблице 1 с указанием мнемоники).
|
Поиск гомологов осуществлялся с помощью локальной версии программы Blastp.
Для создания словаря использовалась команда:
makeblastdb -in PROTEOMES.fasta -dbtype prot
Для поиска по словам с E-value 0.001 использовалась команда:
blastp -query P0A6H1.fasta -db PROTEOMES.fasta -evalue 0.001 -outfmt 7
В результате анализа было найдено 31 неидентичных гомологичных последовательности. Результаты были записаны в файл out.txt
С помощью скрипта script.sh на bash последовательности были извлечены из файла PROTEOMES.fasta
Все последовательности были собраны в один файл командой
cat *_*.fasta >> alignment.fasta
Последовательности были выровнены с помощью программы emma из пакета EMBOSS.
Команда: emma alignment.fasta
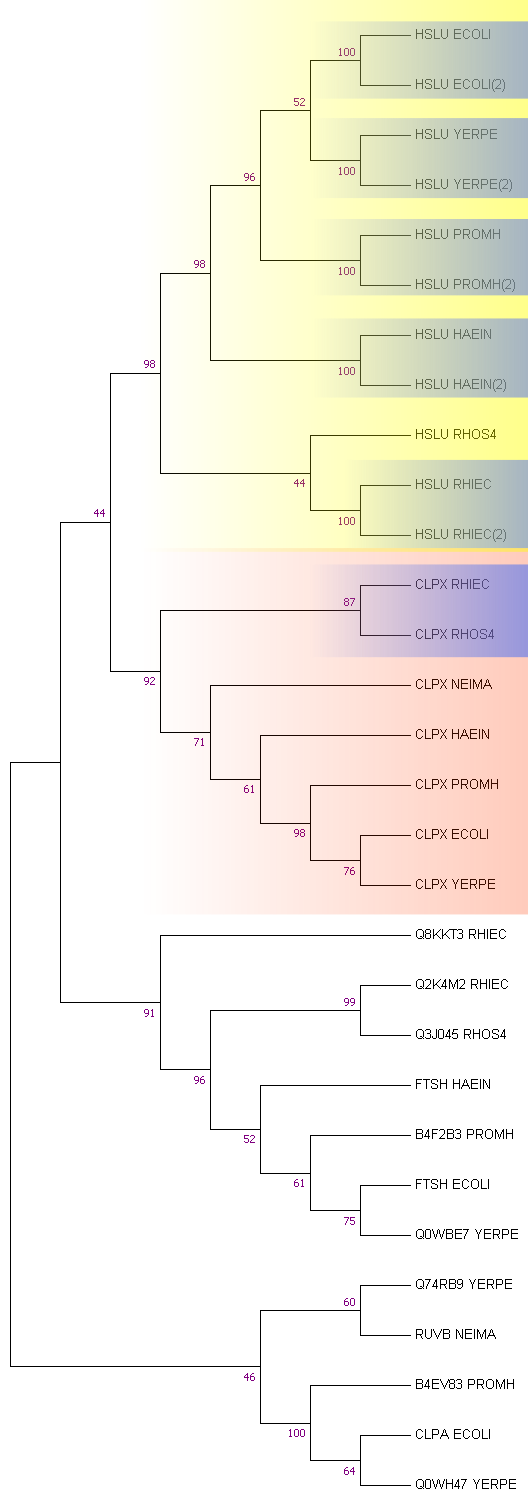
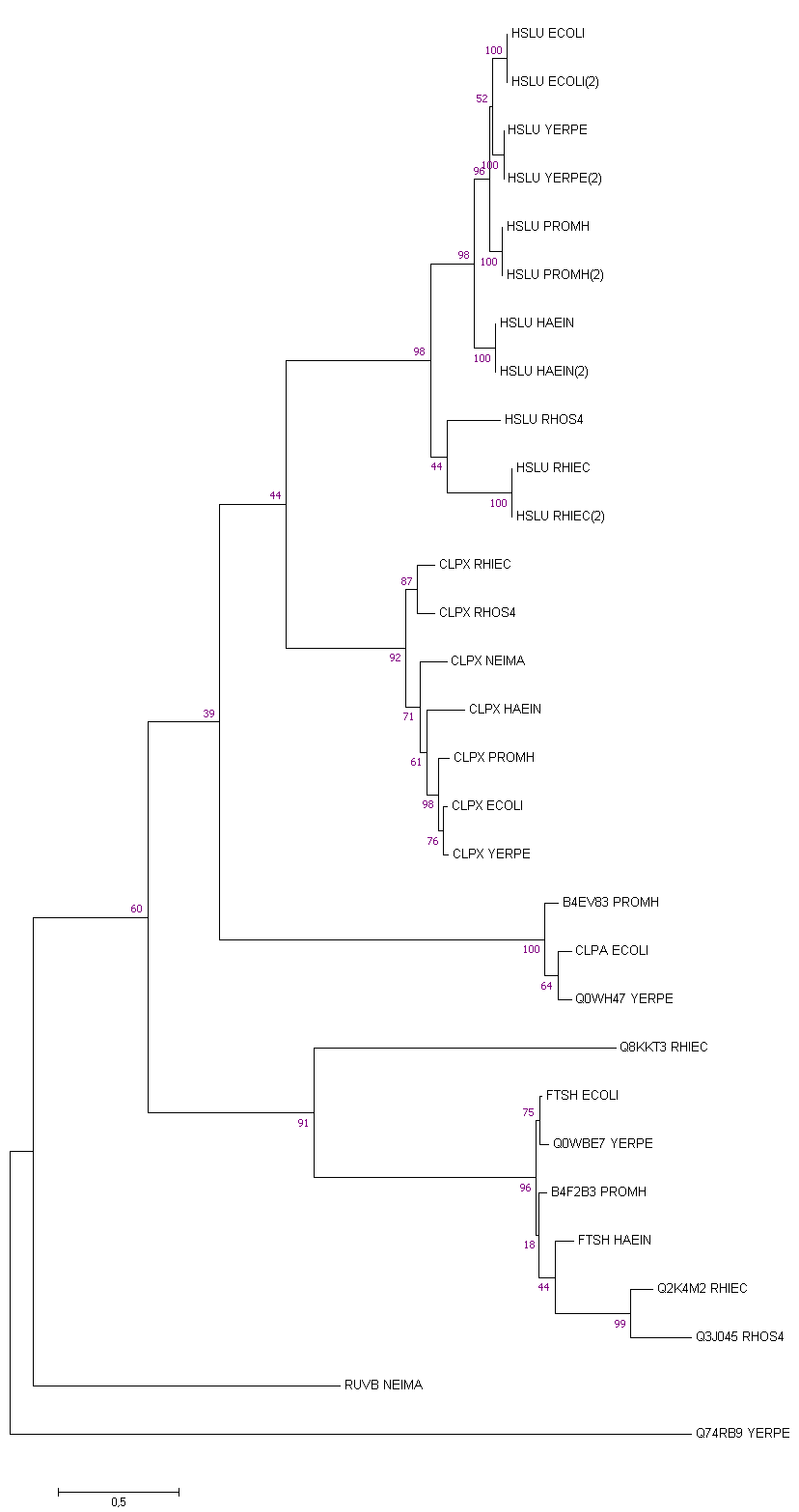
Дерево последовательностей было реконструировано в MEGA 7 с помощью метода Maximum Likelihood, а также был визуализирован вариант дерева, реконструируемого самой программой emma.
Команда: fdrawgram -intreefile den-emma.dnd -plotfile den_plot.fdrawtree -auto
Изображения, полученные разными программами совпадал. Было решено остановиться на Mega 7.
 | ||
|