Мотивы в белках. PSSM и паттерны. PSI-BLAST. Банк Prosite
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
PSI-BLAST
В данном практикуме для последовательности P17265 белка было составлено семейство гомологов. Данный белок (Ribosome hibernation promotion factor) активирует и стабилизирует димеризацию 70S рибосом с помощью RMF (ribosome modulation factor). Для выполнения практикума использовался PSI-BLAST
Таблица 1 содержит в себе информацию о проведённых итерациях в PSI-BLAST. Видно, что на пятой итеррации идентификаторы худшей находки выше порога и лучшей находи ниже порога перестают меняться. Здесь можно было бы прекрвтить итерации, но мне было интересно, через сколько запусков значения E-value перестанут меняться. Оказалось, что данное состояние наступает на 8 итерации. Разница между E-value худшей находки выше порга и лучшей ниже может быть оценена примерно в 20 порядков, что говорит о, возможно, очень высокой гомологии найденных белков.
|
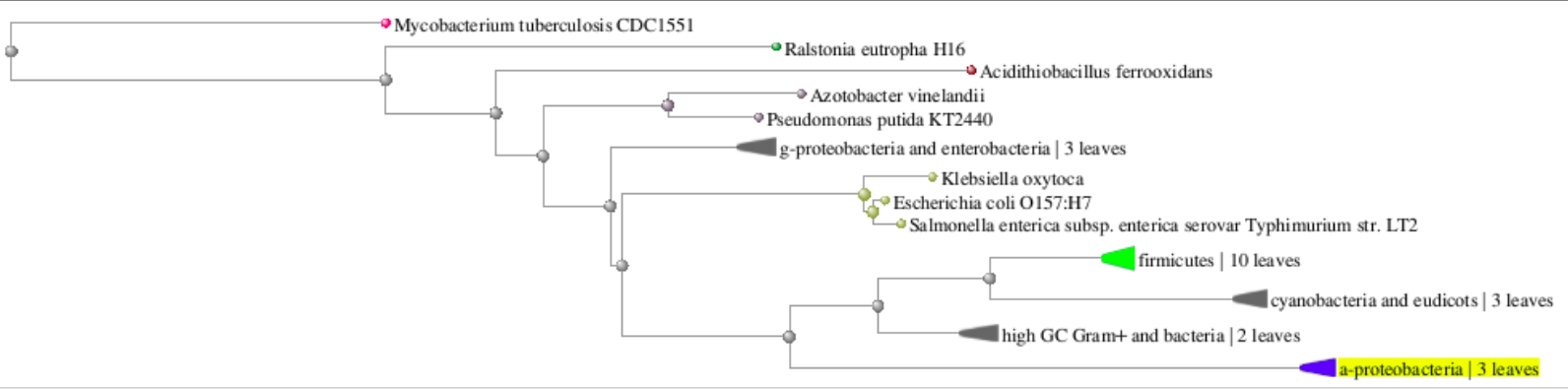
Чтобы оценить полученное семейство было построено дерево данных белков, а также разобрана сама выдача. Дерево, показанное на рисунке 1 отражает предпологаемое эволюционное положение последовательностей внутри группы. Мы видим, что как по названиям семейств белков выдачи, так и по дереву группы получается разбеление на две минорные ветви, одна из которых является HPF, а вторая - Hibernation factor HPF
 |
(**) Разберитесь, как запускать PSI-BLAST из командной строки
USAGE
psiblast [-h] [-help] [-import_search_strategy filename]
[-export_search_strategy filename] [-db database_name]
[-dbsize num_letters] [-gilist filename] [-seqidlist filename]
[-negative_gilist filename] [-entrez_query entrez_query]
[-subject subject_input_file] [-subject_loc range] [-query input_file]
[-out output_file] [-evalue evalue] [-word_size int_value]
[-gapopen open_penalty] [-gapextend extend_penalty]
[-qcov_hsp_perc float_value] [-max_hsps int_value]
[-xdrop_ungap float_value] [-xdrop_gap float_value]
[-xdrop_gap_final float_value] [-searchsp int_value]
[-sum_stats bool_value] [-seg SEG_options] [-soft_masking soft_masking]
[-matrix matrix_name] [-threshold float_value] [-culling_limit int_value]
[-best_hit_overhang float_value] [-best_hit_score_edge float_value]
[-window_size int_value] [-lcase_masking] [-query_loc range]
[-parse_deflines] [-outfmt format] [-show_gis]
[-num_descriptions int_value] [-num_alignments int_value]
[-line_length line_length] [-html] [-max_target_seqs num_sequences]
[-num_threads int_value] [-remote] [-comp_based_stats compo]
[-use_sw_tback] [-gap_trigger float_value] [-num_iterations int_value]
[-out_pssm checkpoint_file] [-out_ascii_pssm ascii_mtx_file]
[-in_msa align_restart] [-msa_master_idx index] [-ignore_msa_master]
[-in_pssm psi_chkpt_file] [-pseudocount pseudocount]
[-inclusion_ethresh ethresh] [-phi_pattern file] [-version]
DESCRIPTION
Position-Specific Initiated BLAST 2.2.31+
Есть ряд параметров, общих для всего семейства BLAST+. Данные параметры представлены в электронной таблице.
Prosite
В данной части работы был уточнён паттерн одного из семейств белков так, чтобы он описывал не все белки данного семейства, а только белки протеобактерий.
В качестве входных данных использовалось выравнивание белков протеобактерий из практикума 2 и паттерн из банка Prosite, описывающий данное семейство.
На заглавной странице Prosite в окошко поиска был внесён идентификатор белка
(RL1_ECOLI).
Был найден один домен Ribosomal protein L1 signature (PATTERN)
Координаты находки в белке:121 - 139
Идентификатор: PS01199
Семейство: RIBOSOMAL_L1
Мотив в белке RL1_ECOLI: MrvVgq.LGqvLGPRGlMPN
Консенсус: [IMGV]-x(2)-[LIVA]-x(2,3)-[LIVMY]-[GAS]-x(2)-[LMSF]-[GSNH]-[PTKR]-[KRAVG]-[GN]-x-[LIMF]-P-[DENSTKQPRAGVI]
 |
Видим, что домен получился какой-то сложный и неточный. Давайте улучшим его.
Из прошлого практикума были взяты и выровнены последовательности семи белков RL1 из разных бактерий. На рисунке 2 представлена визуализация мотив в блоке Jalview.
Возможные улучшения для консенсуса:
1). Можно уточнить имеющийся M-[RP]-[VIL]-V-G-[QR]-L-G-[QTK]-[VI]-L-G-P-R-[GN]-[LM]-M-P-N
2). А также дополнить его F-D-[VR]-[VC]-I-A-[ST]-P-D-[MA]-M-[RP]-[VIL]-V-G-[QR]-L-G-[QTK]-[VI]-L-G-P-R-[GN]-[LM]-M-P-N-P-K-V-G-T-V-T-[PM]-[ND]-V-[AK]
 |
С помощью поиска в базе данных Swees Prot было найдено 114 белков, имеющих улучшенный и дополненный домен.
Они доступны по ссылке на файл и по ссылке на выравнивание.
По запросу в Uniprot "mnemonic:rl1_* taxonomy:proteobacteria" было найдено 427 белков, которые были скачаны в файл
Средствами Excel было сравнено два списка: белков, найденных моим паттерном, и найденных белков из семейства протеобактерий.
Было рассчитано три числа, указанных в таблице 1: число истинных находок (True positives, TP), то есть размер пересечения списков, число ложных находок
(False positives, FP), то есть число тех белков, которые нашлись паттерном, но не входят в правильный список, и число ненайденных (False negatives, FN).
Полученные результаты могут говорить о том, что удивительным образом удалось улучшить консенсус так, чтобы по нему искались только белки семейства протеобактерий (но никакие другие из имеющихся в банке данных не искались).
|
|
