Реконструкция эволюции доменной архитектуры
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
Построение выравнивания представителей домена Pfam белков с разной доменной архитектурой
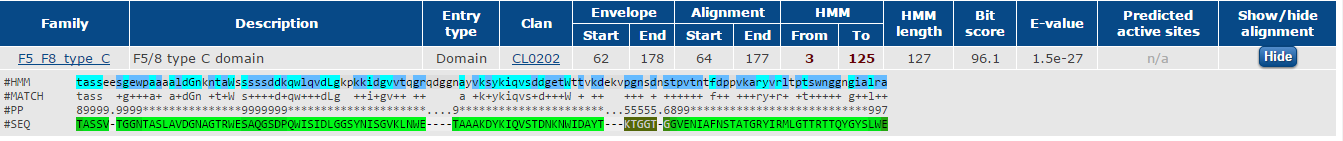
Для выполнения практикума использовался домен F5/8 type C моего белка (сиалидазы из первого и второго семестра). Домен представлен во многих факторах, обеспечивающих коагуляцию крови (5 и 8). Содержит два консервативных цистеина во многих белках, которые замыкают структуру посредством концевых дисульфидных связей. Сервис предлагает 1560 архитектур, содержащих данный домен. При сравнительном анализе видно, что, чаще всего, F5/8 type C домен представлен в двух экземплярах, располагающихся скраю последовательностей.

 |
1. Выбор семейства доменов
Выравнивание, белков из содержащих рассматриваемый домен было скачано в Jalview из Pfam и сохранено в файл . Также было визуализировано две структуры, для которых имелись записи в PDB (Визуализация 1, 2). Работа сохранена в виде проекта. Если вы столкнулись с нехваткой памяти, то следует добавить две следующие строки в конец файла Jalview.lax lax.nl.java.option.java.heap.size.max=1000m lax.nl.java.option.java.heap.size.initial=500m
 |
 |
2. Выбор архитектур
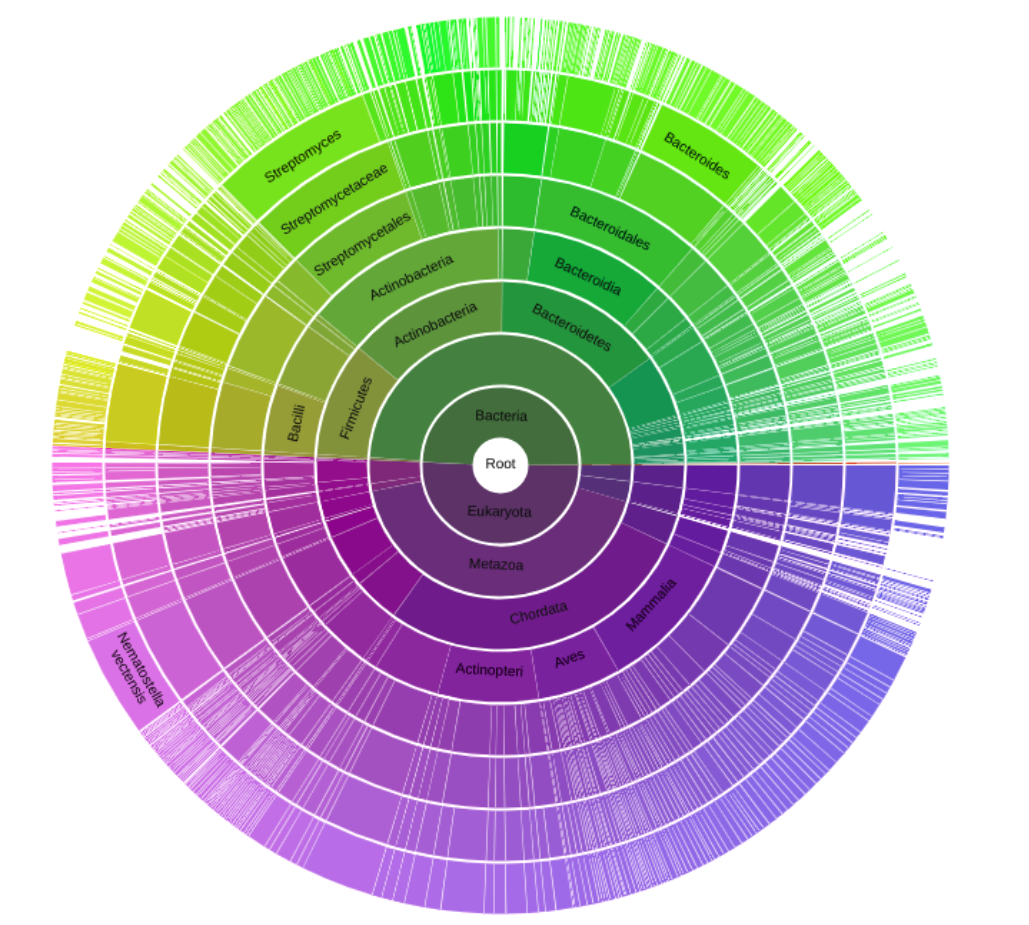
Была получена информация об архитектуре всех последовательностей, входящих в семейство F5_F8_type_C (PF00754). С помощью скрипта swisspfam-to-xls.py. информация об архитектуре была записана в файл PF03067.xls. Команда: python swisspfam-to-xls.py -i /srv/databases/pfam/swisspfam.gz -z -p PF03067 -o PF03067.xls На новом листе была получена сводная таблица, в которой строки – AC последовательностей, столбцы – домены Pfam. Далее для каждого AC в базе данных Uniprot с помощью скрипта uniprot_to_taxonomy.py. была скачана таксономия, и записана в файл taxonomy.xls. Команда: python uniprot-to-taxonomy.py -i uniprot_out.txt -o taxonomy.xls Таблица была дополнена информацией о таксономии доменов, также был добавлен столбец длин доменов. Получившаяся таблица доступна в виде Excel таблицы PF00754_tables.xlsx
Опишем некоторые архитектуры, характерные для данного домена. Для дальнейшего анализа было выбрано две архитектуры
 |
 |
3. Выбор таксона и подтаксонов
Для дальнейшего анализа был взят таксон cellular organisms, который был разделён на подтаксоны Bacteria, Eukaryota. Введём обозначения для данных подтаксонов: Bacteria - B, Eukaryota - E.4. Выбор представителей архитектур
Был произведён выбор представителей из электронной таблицы с целью дальнейшего парсинга с момощью скрипта filter-alignment.pyКоманда: python filter-alignment.py -i alignment.mfa -m in_AC.txt -o align_AC.mfa -a "_"
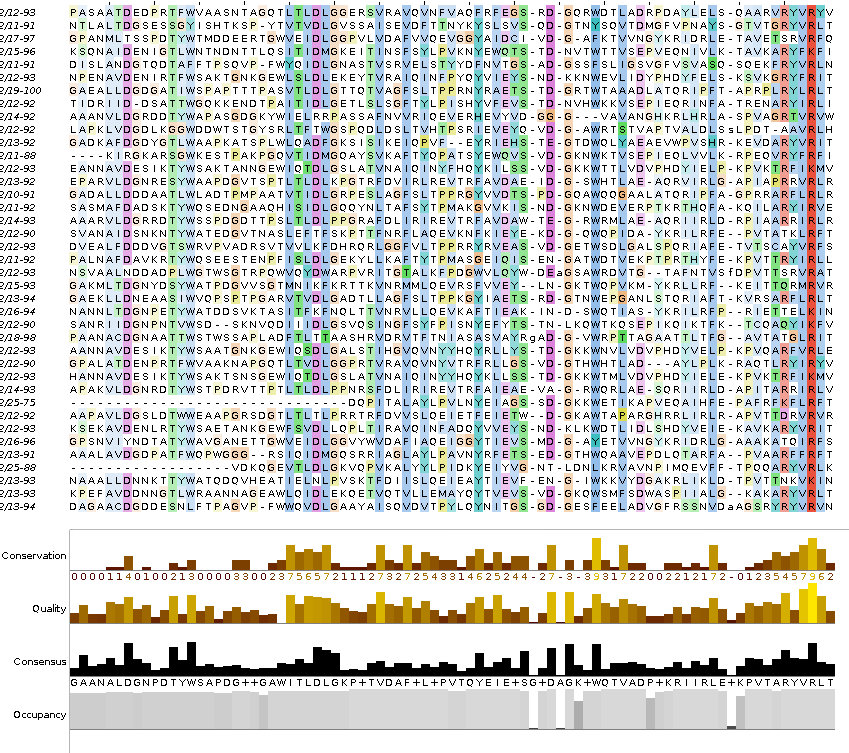
В программе Jalview были обработаны полученные выравнивания. Были удалены С и N концы, содержащие мало информации. Были удалены неинформативные столбцы. Последовательности были покрашены по ClustalX с порогом консервативности равным 15.
 |
Построение филогенетического дерева домена
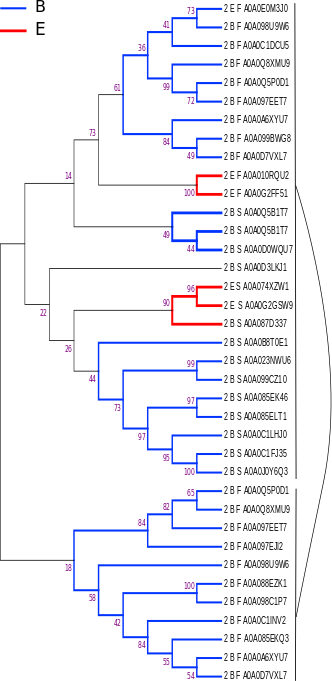
в программе Mega по данному выравниванию бвло построено дерево, изображённое на рисунке 7. Для построения дерева использовался метод Neighbor-Joining с bootstrap 100.
 |
Обсуждение
Из дерева видно, что, в целом, две архитектуры нечётко кластеризуются на дереве. При укоренении в другую точку можно добиться получения двух клад, включающих в себя раздельно практически все последовательности двух подтаксонов. К сожалению, представленность обеих архитектур в группе Eukaryota оказалась очень низкой. Если представить дерево в другом виде (Рисунок 8), и покарасить по принадлежности к архитектуре, то более наглядно видно, что, по-видимому, данные белки обладают общей эволюционной историей. Вероятно, произошёл горизонтальный перенос генов между группами бактерий, что не удивительно.
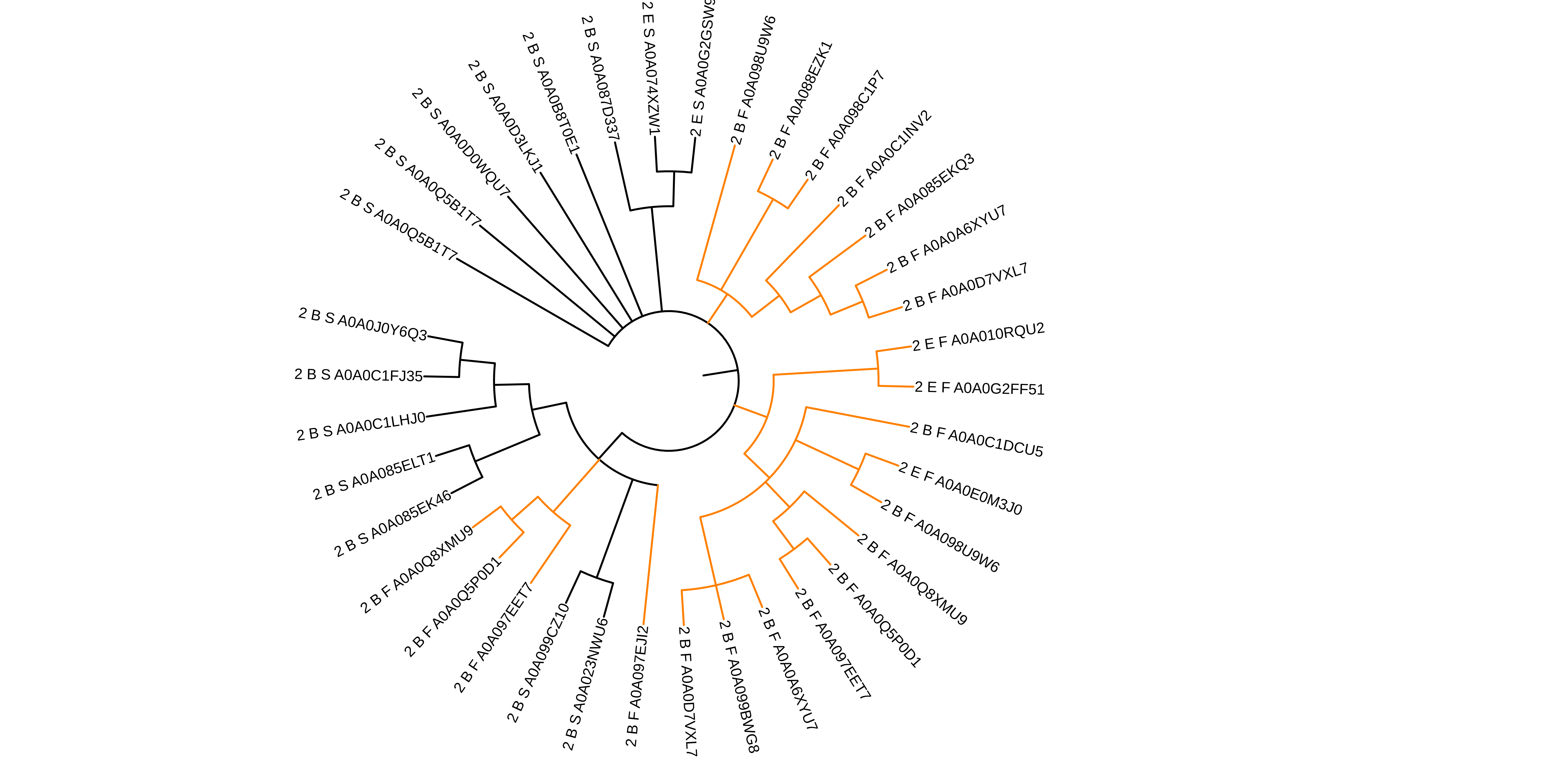 |
