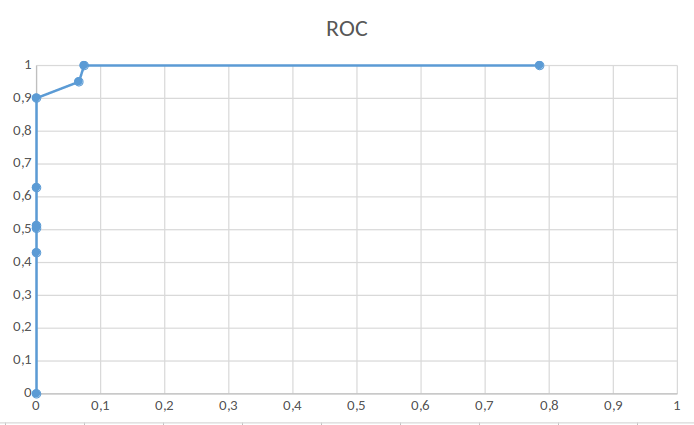
ROC-кривая
Следите за обновлениями и дополнениями
Если Вы заметили опечатки, или ссылка испортилась, пожалуйста, напишите мне
Составление списка белков целевого семейства из `SwissProt`
Так как были получены сложно интерпретируемые результаты, было решено использовать семейство семейство Telomerase_RBD (PF12009).
Теломераза добавляет особые повторяющиеся последовательности ДНК к
3'-концу цепи ДНК на участках теломер, располагающихся на концах хромосом в эукариотических клетках.
При каждом делении клетки её теломерные участки укорачиваются, что создаёт необходимость в постоянной компенсации данного процесса.
Сервис Pfam предлагает 16 архитектур, принадлежащих к семейству.
При сравнительном анализе видно, что, чаще всего, домены семейства представлены в одном экземпляре, располагающемся ближе к центру последовательностей. Почти всегда домены семейства
соседствуют с Reverse transcriptase (RNA-dependent DNA polymerase).
Оказалось, что обратная транскриптаза представлена шире на дереве жизни, что логично. Поэтому было решено сначала рассмотреть данное семейство, а после,
при возможной неудаче, вернуться к Telomerase_RBD.
После всей этой путаницы было выбрано семейство RVT_1 (PF00078).
Данное семейство белков ответственно за создание комплементарной цепи ДНК с РНК матрицы в процессе обратной транскрипции. В обольшинстве случаев данное семейство ассоциировано с ретровирусами.
Используя advanсed search в Uniprot, был произведён поиск вида:
database:(type:* id:PF00078) AND reviewed:yes
позволивший установить все проверенные последовательности, которых оказалось 174.
Последовательности и нужная информация о них была скачана и отфильтрована в таблице Excel по таксономическому положению.
Были выбраны архитектуры, найденные в Retroviridae. Таких архитектур оказалось 121.
С помощью команды:
database:(type:* id:PF00078) taxonomy:"Retroviridae [11632]" AND reviewed:yes
Они были найдены и скачаны из базы данных Uniprot.
Выравнивание доступно по ссылке или в проекте Jalview
Кстати, у Jalview приостановлена работа web сервисов для некоторых сборок java. Я так и не разобрался, какая рабочая. Пришлось делать в Mega7.
Выравнивание Jalview
Построение и калибровка профиля семейства Retroviridae
Попробуем построить профиль для поиска белков из семейства обратных транскриптаз, относящихся к семейству Retroviridae.
Профиль был построен с помощью команды
hmm2build profile_two.out alignment_two.mfa
И отколиброван с помощью команды
hmm2calibrate profile_two.out
Профиль доступен по ссылке
Поиск по профилю по `SwissProt`
Был произведён поиск по базе данных SwissProt с помощью команды
hmm2search profile_two.out /srv/databases/emboss/data/uniprot/uniprot_sprot.fasta > found_two.out
Дополнительные параметры использовать не было необходимости, так как выдача содержала примерно поровну находок выше и ниже порога.
Результат доступен по ссылке
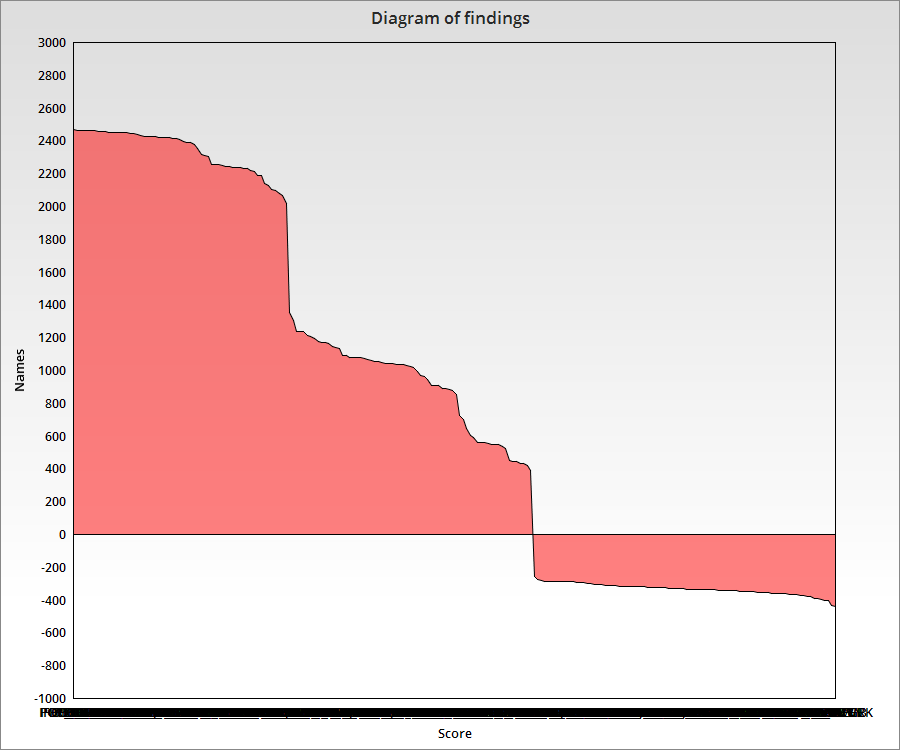
Была построена гистограмма весов хороших и плохих находок. Был выбран предполагаемый порог >1000.
 |
 |
Для построения графиков использовался скрипт, доступный по ссылке. (проблемы с русскоязычными комментариями)
Ссылка на таблицу Excel. Пороговое значение было выбрано со значением 800.
При данном скоре мы всё ещё отделяем нужные последовательности согласно правилу, но не
берём лишние ложноположительные находки.
TP (True Positive) — верно классифицированные последовательности, принадлежащие семейству. Это количество последовательностей, расположенных выше порога.
TN (True Negative) — верно классифицированные последовательности, не принадлежащие семейству, то есть, не содержащие искомую последовательность.
Это количество последовательностей, расположенных ниже порога.
FP (False Positive) — ложно положительные находки.
FN (False Negative) — ложно отрицательные находки.
Результаты. При выполенеии практикума удалось произвести все необходимые действия и манипуляции с файлами. Были произведены вычисления и вызуализированы данные. На основе полученных результатов можно сказать, что профиль, построенный по белкам рассматриемого семейства, способен точно находить последовательности в базе данных Uniprot. Но построенный профиль одновременно с истинными находками охватывает ещё примерно 80% ложноположительных последовательностей. Полученный профиль можно использовать для поиска последовательностей теломеразных белков из семейства Retroviridae. Ссылки |
