Определение вторичной структуры
Выбор объекта и разметка структуры
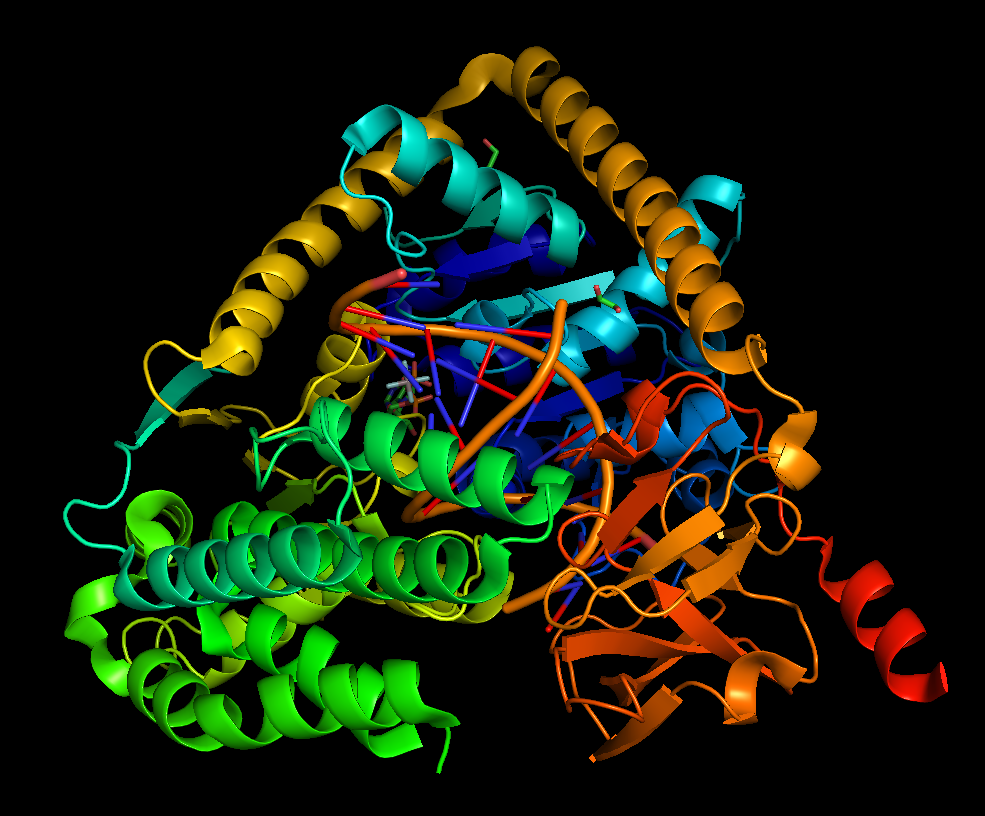
В данном практикуме предлагалось определить вторичную структуру белка. Выбранный белок LGP2 с PDB ID: 5JAJ был найден у Gallus gallus (петух) и является РНК-зависимой АТФ-азой, играющей ключевую роль в индукции иммунного ответа против РНК вирусов, таких как Influenza A virus, Flavivirus, Sendai virus [1]. На рисунке 1 представлен общий вид белка LGP2.
 |
Сравнение выдачи программ
Далее была получена информация о вторичной структуре 5JAJ с помощью двух программ, доступных на сервере kodomo:
DSSP mkdssp -i 5jaj.pdb -o 5jaj_dssp.txt
Stride stride -o 5jaj.pdb > 5jaj_stride.txt (были игнорированы X и Y цепи как содержащие менее 5 остатков)
На вход подавался PDB файл 5jaj.pdb
На выход получили файлы 5jaj_dssp.txt и 5jaj_stride.txt
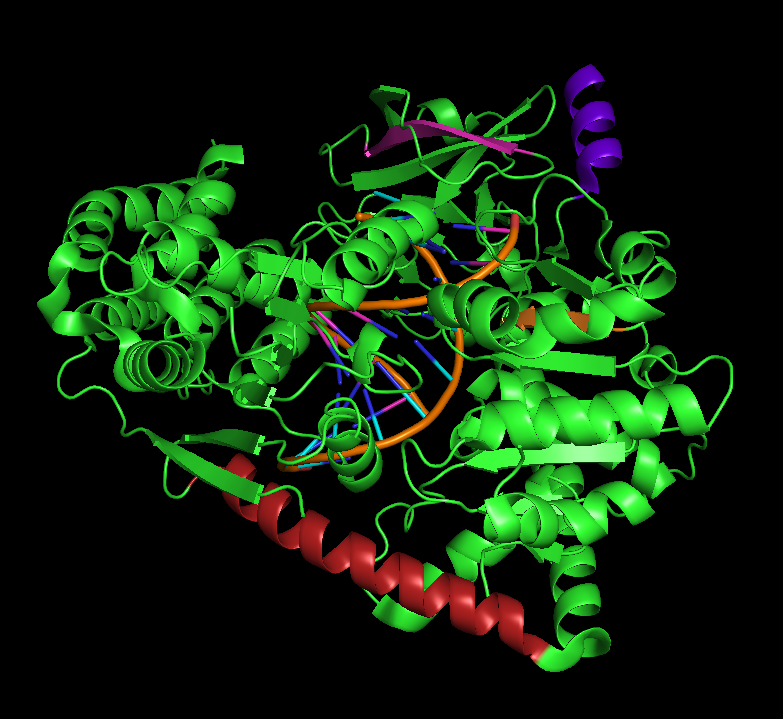
Для сравнения выдачи программ были выбраны две α-спирали и два β-листа.
Объекты специально выбирались максимально различные.
Я взял одну большую и одну маленькую α-спираль. Малиновая спираль располагается в центре цепи, а синяя - на конце цепи.
Складки также были выбраны исходя из их размера и окружения. Оранжевая складка маленькая и находится в окружении спиралей, а розовая - большая и окружена другими
складками.
 |
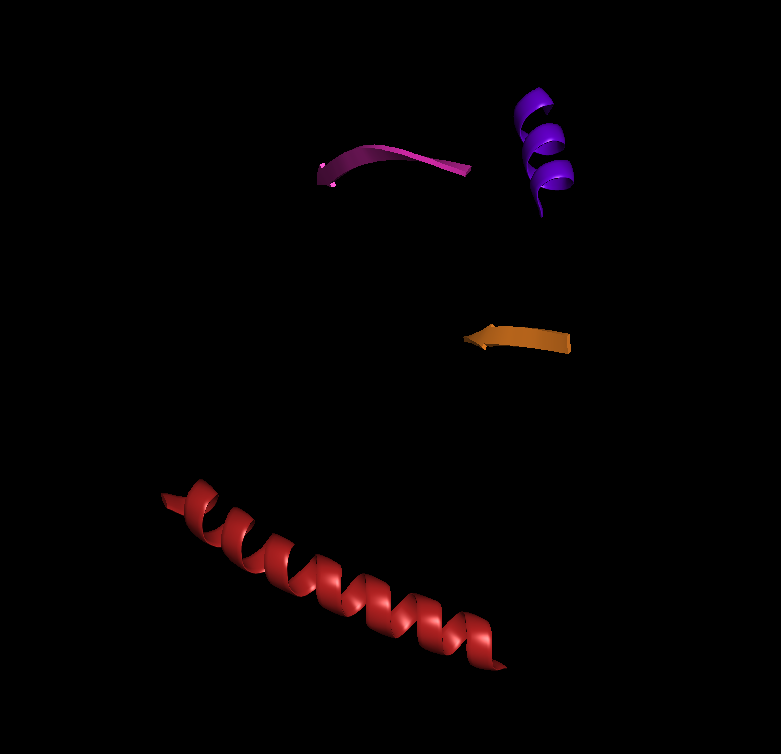 |
Таблица 1. Сравнение разных алгоритмов разметки вторичной структцры.
| Элемент | PDB | DSSP | Stride |
| α-спираль 1 | SER A 485 - MET A 511 | ARG 486 A - MET A 511 | ARG 486 A - ALA 510 A |
| α-спираль 2 | SER A 661 - ASP A 671 | TYR 662 A - GLN 670 A | TYR 662 A - GLN 670 A |
| β-лист 1 | TRP A 602 - CYS A 610 | TRP A 602 - CYS A 610 | TRP 602 A - CYS 610 A |
| β-лист 2 | VAL A 99 - THR A 103 | VAL A 99 - THR A 103 | VAL 99 A - THR 103 A |
 |
Интересные особенности
Из таблицы видно, что в некоторых случаях, в сравнении с разметкой PDB, DSSР и Stride укорачивают α-спирали на несколько аминокислот. Вероятно, это связано с особенностями алгоритмов: для разметки особенностей вторичной структуры они используют водородные связи, которые а) могут быть неверно определены из-за погрешностей РСА, б) могут пере (что чаще) или недооцениватья алгоритмами. Возможно также, что именно в этом белке значения торсионных углов были расхожи с зашитыми в Stride, что привело к укорочению аннотации.Разметка β-листов для обеих программ согласуется с разметкой в PDB файле.
Плюсом разметки с помощью Stride я считаю возможность увидеть не только α-спирали, но и 3-10 спирали, π-спирали.
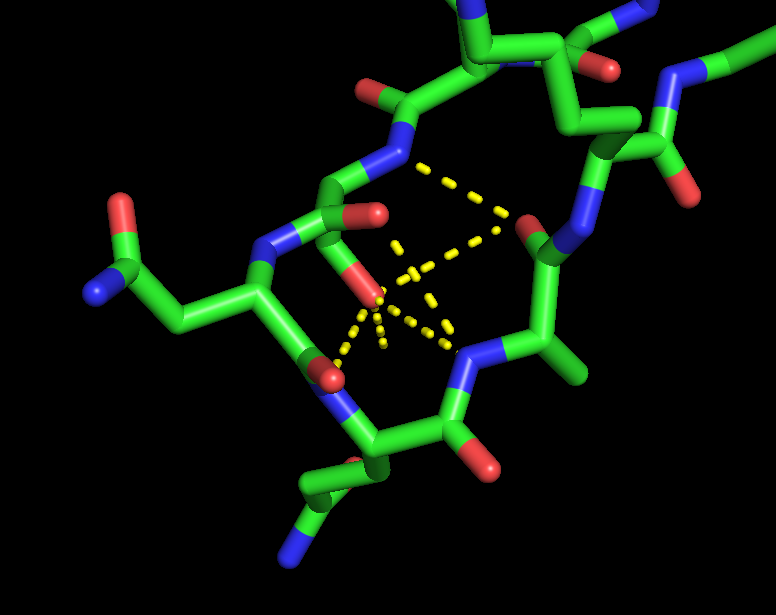
Также Stride нашел 2 изолированных β-моста (на рис. 3 показаны желтыми стрелками, на рис. 4 визуализирован первый из них).
 |
