Практикум 10.
Задание 1.
Для выполнения данного задания я выбрал геномы Phytobacter diazotrophicus (по этой бактерии я делал миниобзор ы первом семестре) и Cronobacter sakazakii (относится к тому же семейству, что и Phytobacter; выбор пал именно на этот организм из-за названия). Запуск алгоритмов BLAST происходил по средством использования "кодов идентификации" (accession number). Их я нашел через базу данных Nucleotide на сайте NCBI. Выравнивание последовательностей происхдило с использованием алоритмов blastn с длиной слова 11 и megablast с длиной слова 16 и 28.
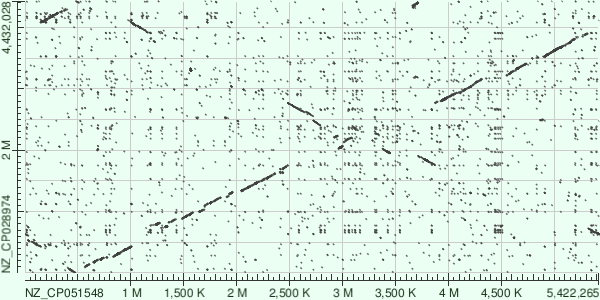
Анализ Dot Plot.
Анализ проводился по изображению Fig. 2.. По оси Ox расположен геном Phytobacter, по оси Oy - геном Cronobacter.
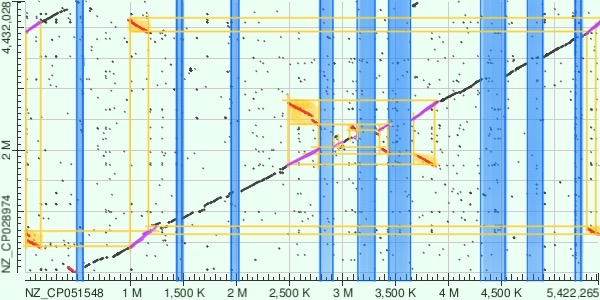
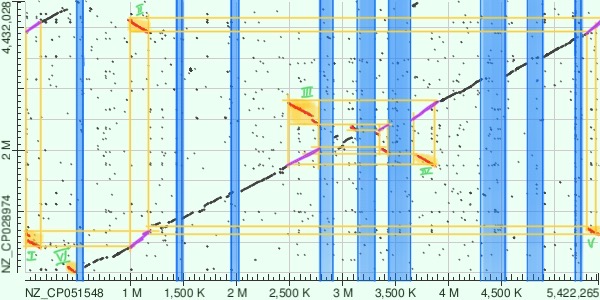
Линия выравнивания является восходящей, что свидетельствует об одинаковой ориентации цепей.Синие столбцы соответсвуют участкам, где либо произошли делеции, либо инсерции. Красными линиями я выделил участки, претерпевшие инверсию. Фиолетовым цветом показаны недостающие учаскти линии. Желтыми цветом - возможное соответствие участков, подвергшимся релокации, недостающим участкам линии выравнивания. Начало линии по оси Ox только с 400K нуклеотида можно объяснить тем, что разрезали геномы бактерий в разных местах.
На графике можно выделить шесть крупных участков, сответсвующих глобальным перестройкам:
- Длирна составляет около 150 тыс. нуклеотидов; претерпел траслокацию и инверсию; небольшой разрыв по оси Oy мог образоваться в результате инсерции.
- Длина примерно равна 220 тыс. нуклеотидов; подвергся транслокации и инверсии.
- Длина 300 тыс. нуклеотидов; изменения аналогичные перестройке на участке 1.
- Длина около 150 тыс. нуклеотидов; был транслоцирован и инвертирован.
- Длина около 100 тыс. нуклеотидов; был транслоцирован и инвертирован.
- Длина около 100 тыс. нуклеотидов; претерпел инверсию.