Практикум 15.
1. Подготовка чтений программой trimmomatic.
Сначала я создал файл со всеми адаптерами для Illumina:
cat /mnt/scratch/NGS/adapters/* >> /mnt/scratch/NGS/petros/pr15/adapters.fasta
Чтения для данного практикума я скачал командой (вес файла - 167 мегабайт):
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/009/SRR4240379/SRR4240379.fastq.gz
Для последующей работы требовалось удалить адаптеры:
TrimmomaticSE SRR4240379.fastq.gz trimm1.fq.gz ILLUMINACLIP:adapters.fasta:2:7:7
В результате выяснилось, что 130303 (1.76%) чтений являются остатками адаптеров. Также была проведена фильтрация по качеству нуклеотидов (нуклеотиды справа качеством не ниже 20) и по длине (>_32 нукл.):
TrimmomaticSE trimm1.fq.gz trimm2.fq.gz ILLUMINACLIP:adapters.fasta:2:7:7 TRAILING:20 MINLEN:32
После выполнения команды из файла было удалено 425888 (5.76%) чтений, а вес его уменьшился с 167 мегабайтов до 156.
2. Работа с программой velveth.
Я использовал программу velveth, чтобы подготовить k-меры длины 31 в директории ./velveth_dir:
velveth velveth_dir 31 -short -fastq.gz trimm2.fq.gz
3. Работа с программой velvetg.
Далее была произведена сборка на основе k-меров:
velvetg ./velveth_dir
Программа выдала на STDOUT, что N50=25646. Потом я определил длины трёх самых длинных контигов, их ID и покрытие (во второй команде значениям опции -e соответствуют длины, полученные предыдущей командой):
cut -f2 stats.txt | tail -n+2 | sort -h -r | head -n 3
grep -e 49912 -e 49262 -e 33085 stats.txt > longcontigs.txt
В файле longcontigs.txt первый cтолбец соотвесвует ID контига, второй - длине, а шестой - покрытию.
Для анализа покрытия я сначала определил значение "типичного" (оно, на мой взгляд, изменяется от 30 до 38, хотя самыми частыми являются значения от 1 до 12):
cut -f6 stats.txt | tail -n+2 | sort -h -r | cut -d . -f1 | uniq -c
Из полученных данных можно выделить значения аномально большого покрытия: 474299, 2694, 610, и аномально малым покртием: 1, 2, 3, 4, 5.
4. Анализ.
Для анализа выравнивания с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253) были выбраны контиги из следующих файлов:
- node_5_length_33085_cov_36.259029.fasta
- node_6_length_49912_cov_35.907238.fasta
- node_9_length_49262_cov_34.772179.fasta
Контиг Node 5.
На Рис. 1 видно, что линии являются восходящими, из чего следует одинаковая ориентация контига и цепи генома. Первая линия сответствует участку 1 из таблицы 1. Далее следует разрыв, который мог образоваться в результате инсерции или делеции, а потом еще одна линия, соответствующая трем остальным участками из таблицы 1, "разрывы" между которыми тоже могли появиться из-за инсерций или делеций.
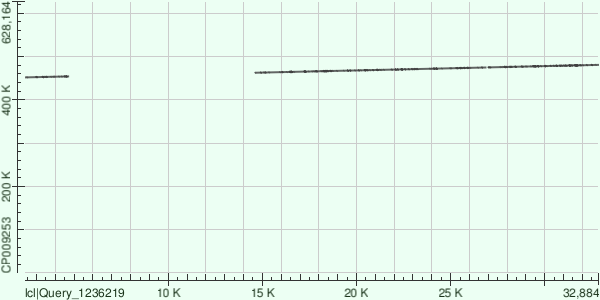
| номер участка | координаты участка контига | координаты участка хромосомы | SNP | Gap | Идентичность |
|---|---|---|---|---|---|
| 1 | 2390-4733 | 451729-454069 | 488 | 55 | 77% |
| 2 | 14624-19565 | 462496-467421 | 992 | 162 | 77% |
| 3 | 19595-26906 | 467412-474667 | 1489 | 208 | 77% |
| 4 | 27009-32884 | 474844-480660 | 1288 | 255 | 74% |
Контиг Node 6.
Как и в случае с node 5, контиг одинаково ориентирован с цепью генома. Из заметного на рис. 2 можно отметить две крупные инсерции/делеции между 1 и 2, а также 2 и 3 участками из таблицы 2.
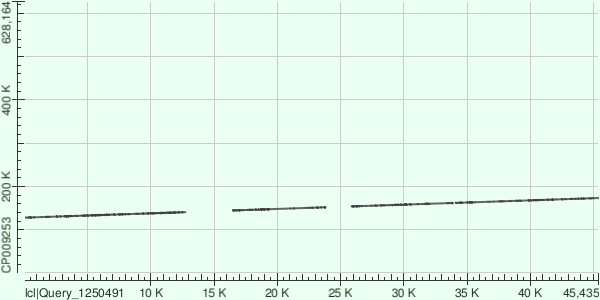
| номер участка | координаты участка контига | координаты участка хромосомы | SNP | Gap | Идентичность |
|---|---|---|---|---|---|
| 1 | 53-12793 | 127825-140555 | 2723 | 544 | 75% |
| 2 | 16432-23831 | 144368-151796 | 1430 | 243 | 78% |
| 3 | 25811-33895 | 153752-161738 | 1556 | 266 | 78% |
| 4 | 34101-38961 | 161898-166752 | 894 | 108 | 80% |
| 5 | 38992-45435 | 166750-173180 | 1393 | 159 | 76% |
Контиг Node 9.
Основываясь на том же признаке, что был указан для node 5 и node 6, можно сделать вывод об одинаковом направлении контига и цепи генома . У данного контига больше всего участков с крупными делециями/инсерциями, что обусловлено его большим относительно предыдущих двух контигов размером.
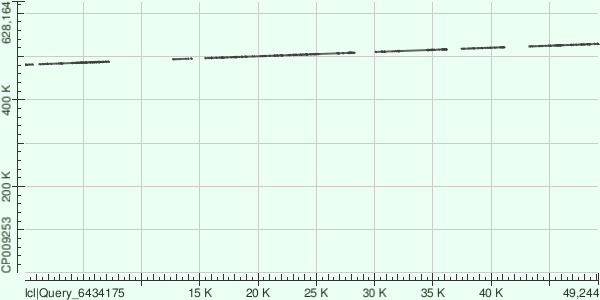
| номер участка | координаты участка контига | координаты участка хромосомы | SNP | Gap | Идентичность |
|---|---|---|---|---|---|
| 1 | 4-683 | 480874-481545 | 102 | 20 | 82% |
| 2 | 1157-7214 | 481997-488106 | 1309 | 308 | 74% |
| 3 | 12687-14063 | 493487-494864 | 262 | 13 | 80% |
| 4 | 14197-14315 | 495033-495148 | 7 | 5 | 90% |
| 5 | 15481-19759 | 496111-500325 | 915 | 154 | 75% |
| 6 | 19875-28320 | 500370-508806 | 1750 | 351 | 76% |
| 7 | 30011-36189 | 510438-516539 | 1150 | 187 | 79% |
| 8 | 37450-41179 | 517766-521500 | 760 | 101 | 77% |
| 9 | 43182-48769 | 523105-528679 | 1109 | 207 | 77% |
| 10 | 48839-49244 | 52879-529211 | 42 | 26 | 84% |