Практикум 1.
Отобранные бактерии
| Название | Мнемоника |
|---|---|
| Bartonella henselae | BARHE |
| Burkholderia mallei | BURMA |
| Escherichia coli | ECOLI |
| Haemophilus influenzae | HAEIN |
| Paracoccus denitrificans | PARDP |
| Pasteurella multocida | PASMU |
| Polynucleobacter asymbioticus | POLAQ |
Скобочная формула дерева
((PARDP, BARHE), ((POLAQ, BURMA), ((PASMU, HAEIN), ECOLI)))
Отображение дерева
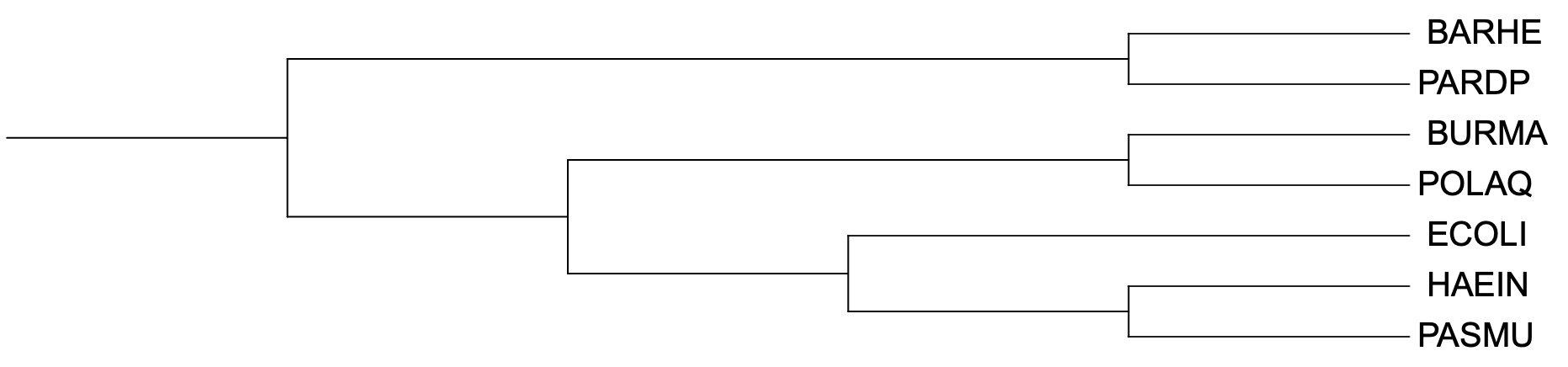
Ветви дерева
Дерево содержит следующие ветви:
- {PARDP, BARHE} против {POLAQ,BURMA,PASMU,HAEIN,ECOLI}
- {BURMA, POLAQ} против {PARDP,BARHE,PASMU,HAEIN,ECOLI}
- {PASMU, HAEIN} против {PARDP,BARHE,POLAQ,BURMA,ECOLI}
- {PASMU,HAEIN,ECOLI} против {PARDP,BARHE,POLAQ,BURMA}
Практикум 2.
Задание 1.
В списке, представленном ниже, содержится информация о таксонах бактерий, по которым строилось филогенетическое дерево из практикума 1 (рис. 1):
- Bartonella henselae: cellular organisms; Bacteria; Pseudomonadota; Alphaproteobacteria; Hyphomicrobiales; Bartonellaceae; Bartonella
- Burkholderia mallei: cellular organisms; Bacteria; Pseudomonadota; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Burkholderia; pseudomallei group
- Escherichia coli: cellular organisms; Bacteria; Pseudomonadota; Gammaproteobacteria; Enterobacterales; Enterobacteriaceae; Escherichia
- Haemophilus influenzae: cellular organisms; Bacteria; Pseudomonadota; Gammaproteobacteria; Pasteurellales; Pasteurellaceae; Haemophilus
- Paracoccus denitrificans: cellular organisms; Bacteria; Pseudomonadota; Alphaproteobacteria; Rhodobacterales; Paracoccaceae; Paracoccus
- Pasteurella multocida: cellular organisms; Bacteria; Pseudomonadota; Gammaproteobacteria; Pasteurellales; Pasteurellaceae; Pasteurella
- Polynucleobacter asymbioticus: cellular organisms; Bacteria; Pseudomonadota; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Polynucleobacter
Соответсвие таксонов и нетривиальных веток, которые выделяют их занесено в таблицу 2.
| Нетривиальная ветка | таксон |
|---|---|
| {PARDP, BARHE} | Alphaproteobacteria |
| {BURMA, POLAQ} | Burkholderiaceae |
| {PASMU, HAEIN} | Pasteurellaceae |
| {PASMU,HAEIN,ECOLI} | Gammaproteobacteria |
Задание 2.
Для выполнения данного задания мною был выбран белок с мнемоникой IF2, который является фактором трансляции 2 и характерен для бактерий. Данный белок стабилизирует комплекс из формилметиониновой тРНК и 50S (большой субъединицы рибосомы) за счет энергии, полученной при катализируемым им гидролизе ГТФ. Ниже представлены три раздела, посвященные построению филогенетических деревьев с помощью разных алгоритмов.
Fastme.
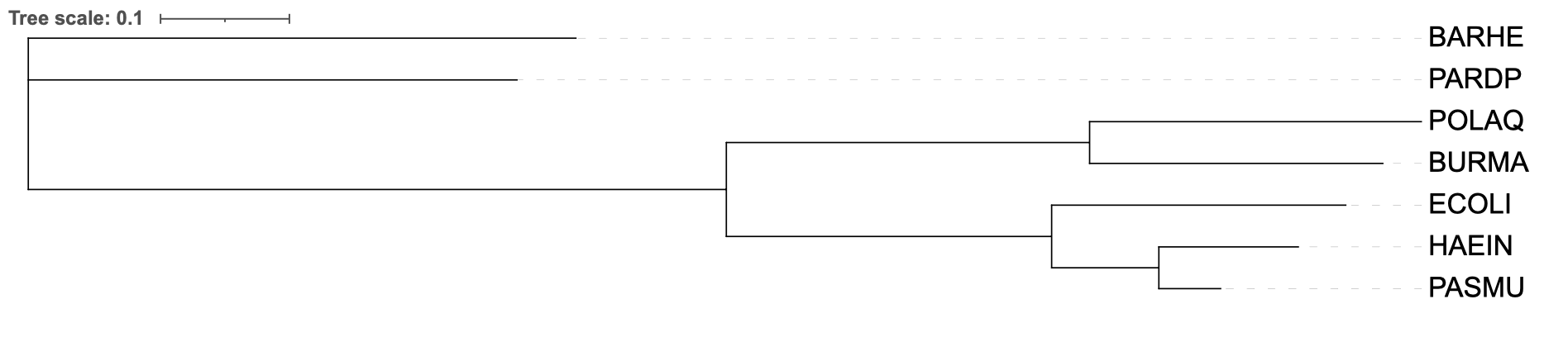
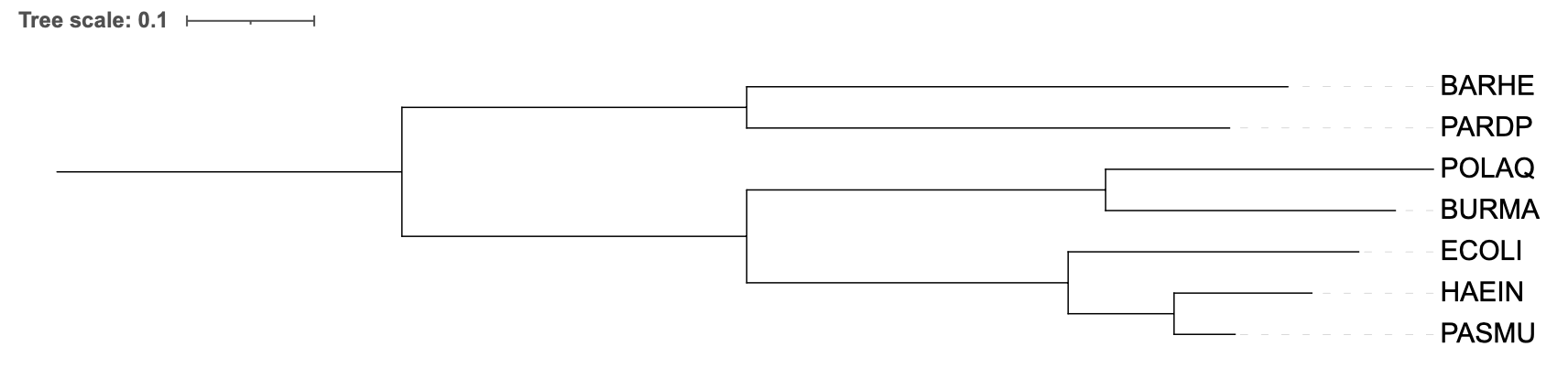
TNT.
PhyML.
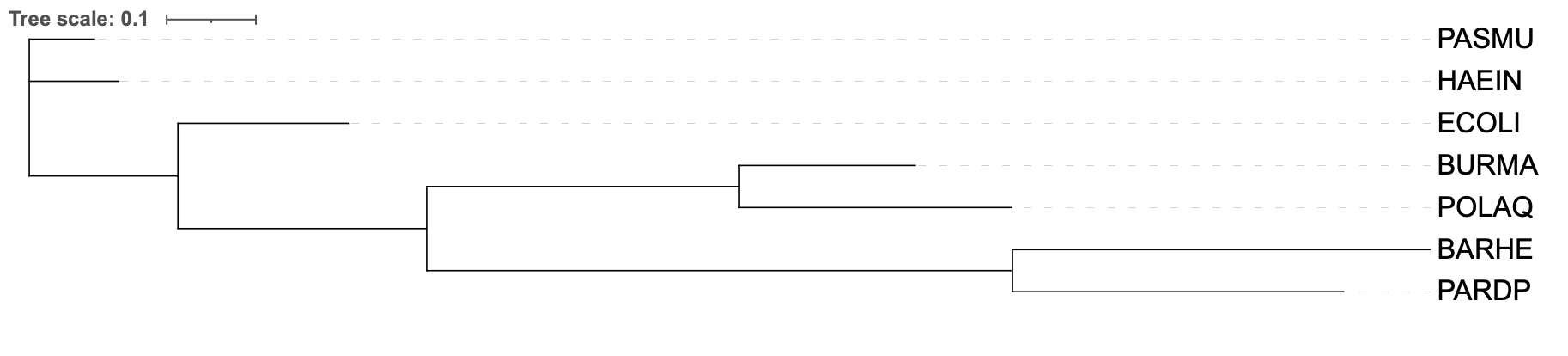
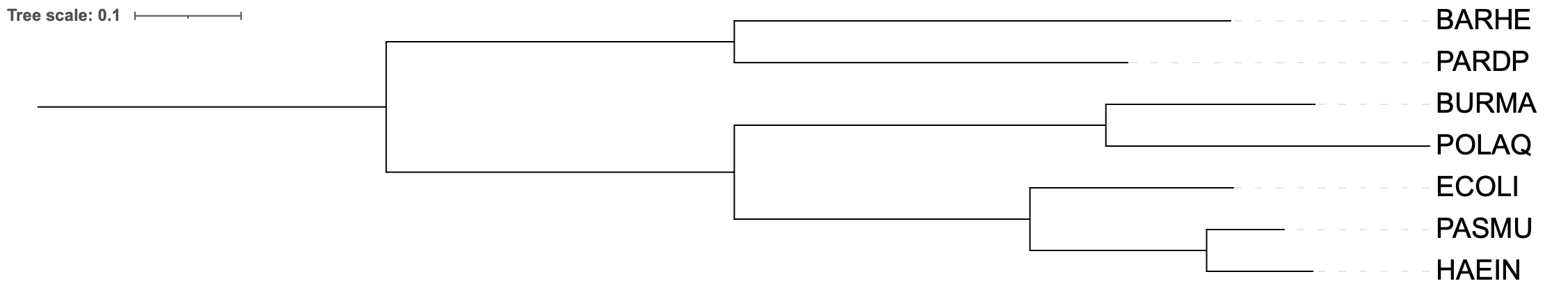
Практикум 3.
Задание 1.
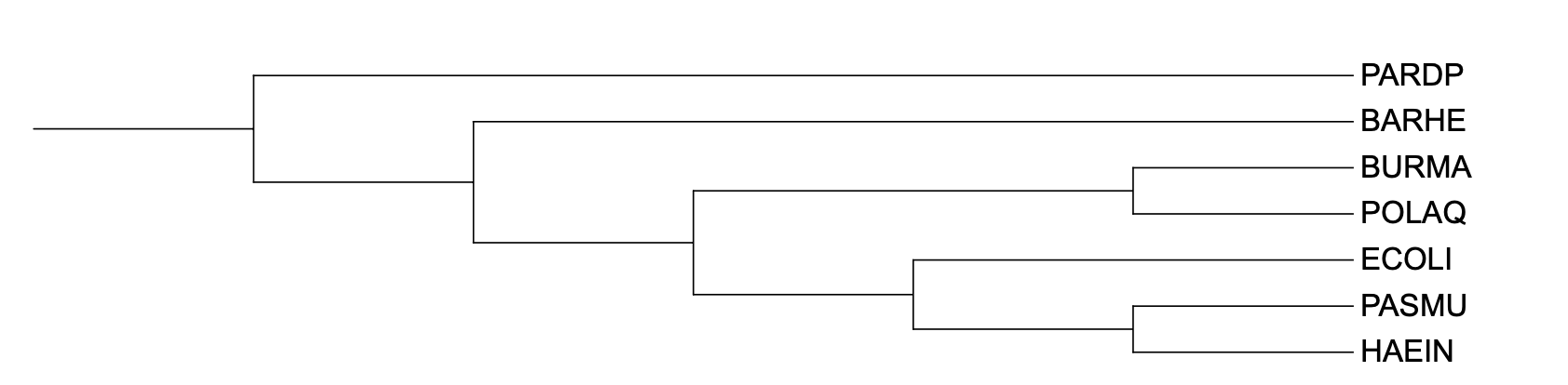
В ходе выполнения данного задания было построено филогенетическое дерево с укоренением посредством внешней группы. Для этого был выбран белок IF2 сенной палочки (Bacillus subtilis). Дерево создано на основе результата работы алгоритма FastME.
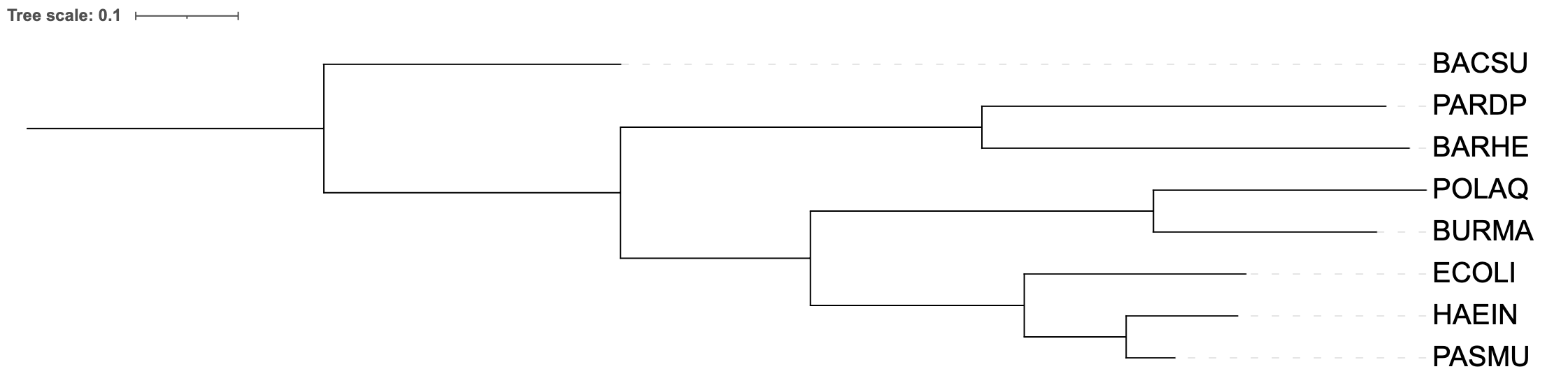
Задание 2. Bootstrap
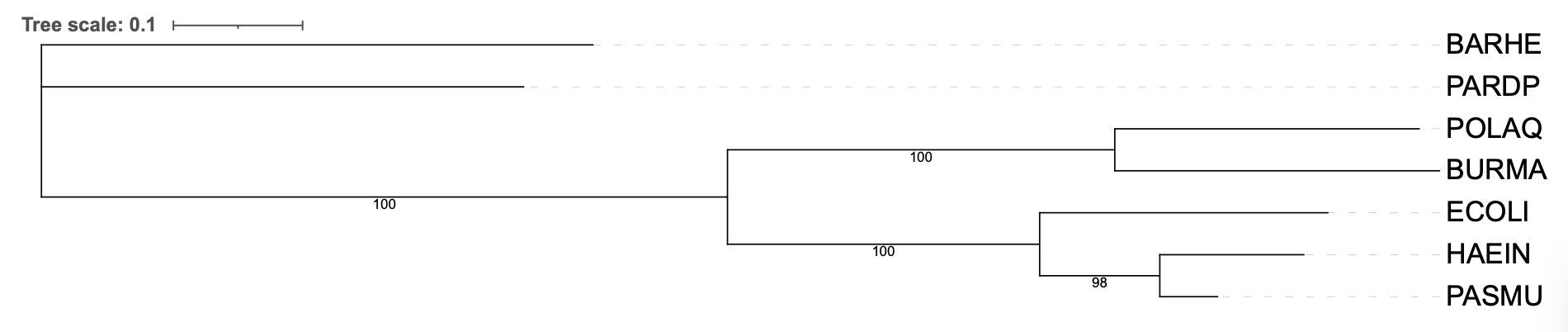
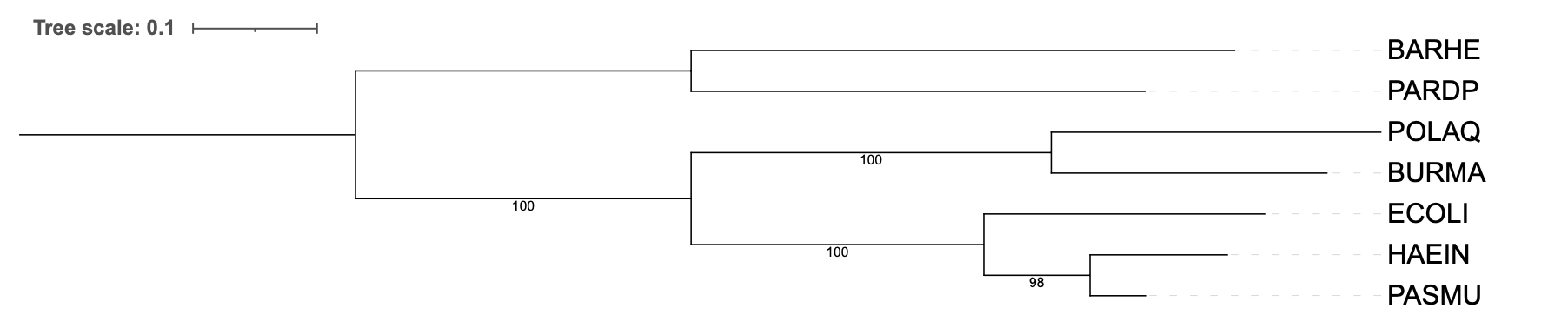
Задание 3. Построение дерева по нуклеотидным последовательностям 16S РНК.
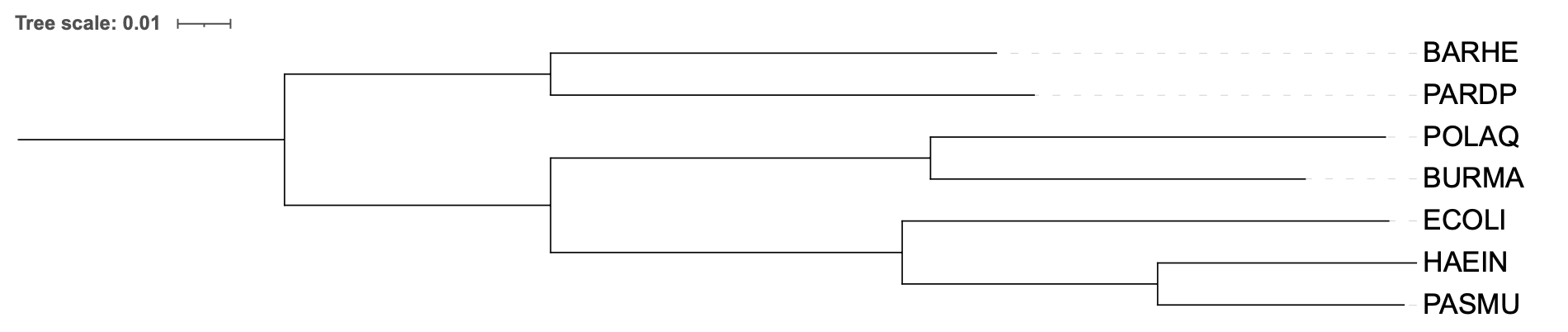
Формула дерева: (((ECOLI:0.092435,(PASMU:0.046805,HAEIN:0.049154):0.048529):0.06679,(PARDP:0.091853,BARHE:0.084671):0.101133):0.072163,BURMA:0.071179,POLAQ:0.086421)