Практикум 4.
Составление списка гомологичных последовательностей.
Для выполнения данного пункта я собрал все нужные мне протеомы в один файл с помощью команды cat. Далее я создал базу данных по этому файлу командой makeblastdb (с параметром -dbtype prot). Поиск гомологов был осущетвлен с помощью blastp с порогом e-value 0.001 (результат работы алгоритма).
- список находок из выдачи BLAST
- Последовательности, скаченные из Uniprot, с отредактированными названиями
Построение дерева
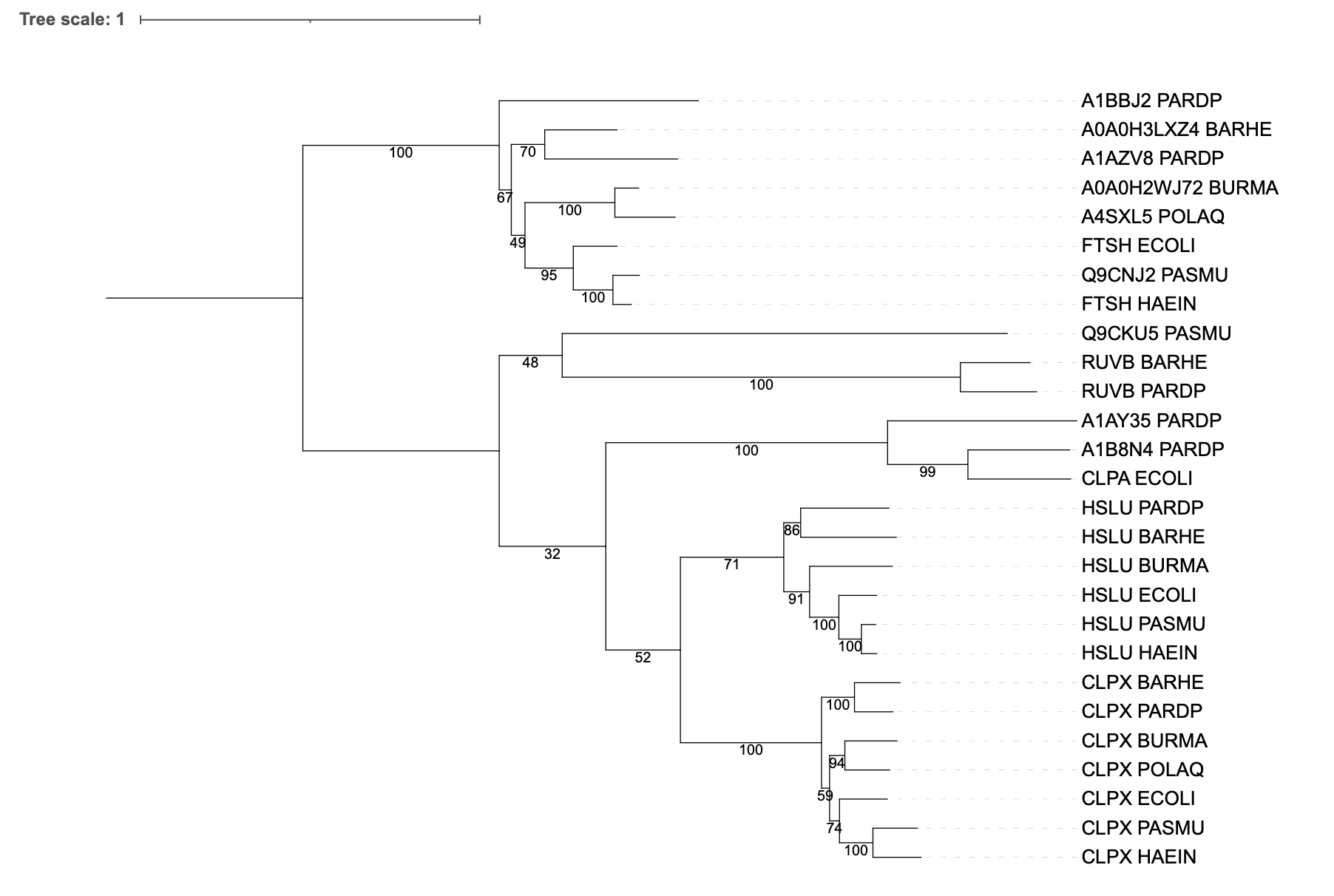
Результат работы алгортма (формула дерева).
Паралоги:
- RUVB PARDP, A1BBJ2 PARDP
- A1AY35 PARDP, A1B8N4 PARDP
- RUVB BARHE, A0A0H3LXZ4
Ортологи:
- CLPX PASMU, CLPX HAEIN
- HSLU PARDP, HSLU BARHE
- RUVB PARDP, RUVB BARHE
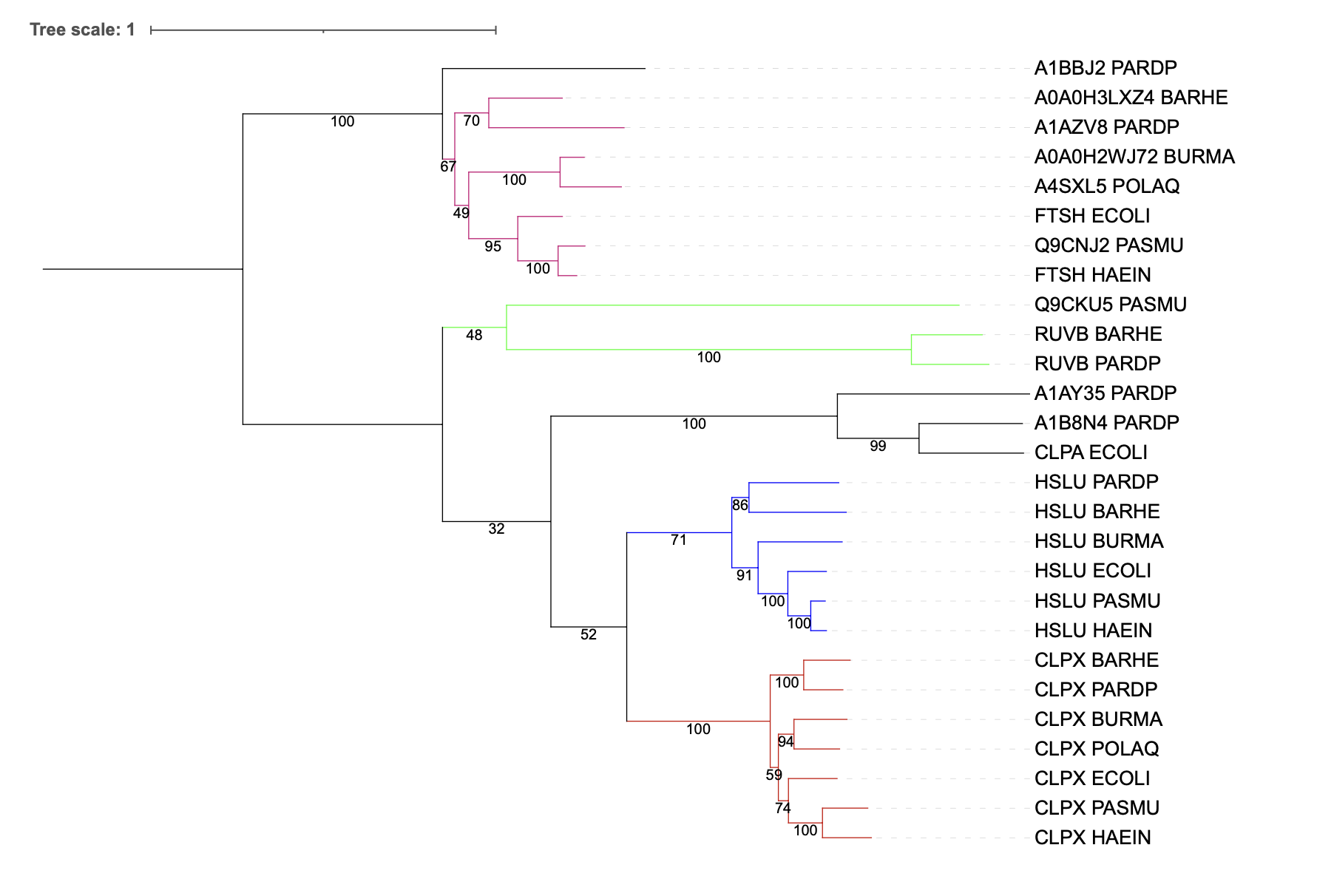
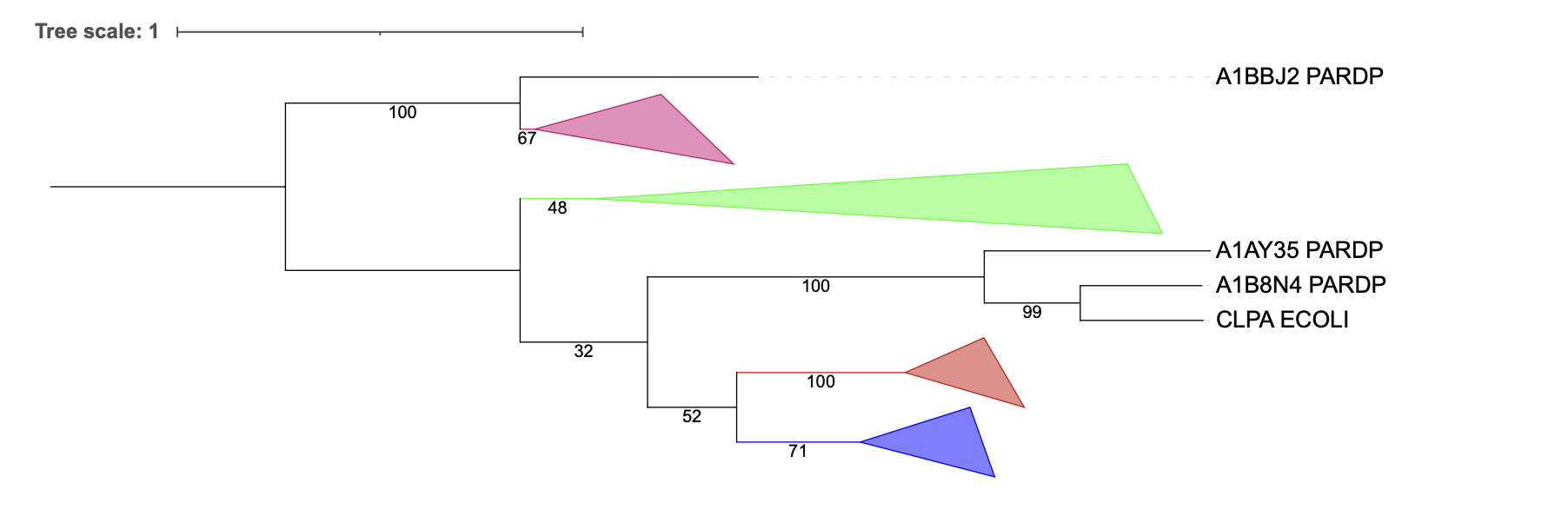
Обсуждение результатов
В красную группу попали все бактерии, и филогения данного белка соответствует филогении бактерий (см. практикум 1, Рис. 1). Аналогичная ситуация для розовой группы. В синей группе нет белка из POLAQ, но ветки не противоречат филогении бактерий. Зеленая группа состоит только из трех белков (из PASMU, BARHE, PARDP); противоречий в ветках с бактериальными деревом нет.