Практикум 6.
Задание 1.
Краткая информация по выбранному белку:
- Название на русском: Кислотный сахаро-специфичный порин NanC
- Название на английском: Acidic sugar-specific porin NanC
- Идентификатор PDB: 2wjr
- Идентификатор UniProt: NANC_ECOLI
- Из какого организма белок: Escherichia coli
- В какой мембране располагается: внешняя мембрана грамотрицательной бактерии
- Функция: канал во внешней мембране, пропускающий внутрь бактерии н-ацетилнейраминовую кислоту (самую распространенную сиаловую кислоту на поверхности клеток хозяина)
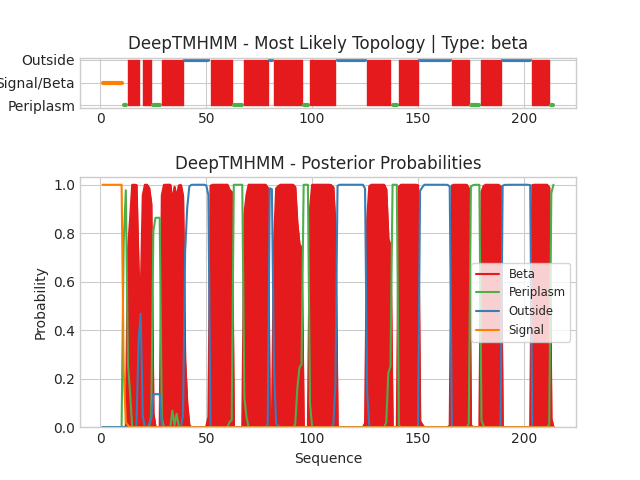
Для предсказания трансмембранных участков методом DeepTMHMM я использовал последовательности своего белка, взятую из PDB (ссылка на последовательность). Хотя количество предсказанных бета-листов совпадало и большинство из них отличались друг от друга только на одну, две аминокислоты, некоторые участки плохо перекрывались (первые два). Это возможно связано с длиной первого бета-листа, который довольно короткий относительно других 11, образующих бета-цилиндр.
Текстовая выдача метода DeepTMHMM.
| Номера участков | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| OPM | 3-9 | 17-26 | 30-38 | 54-63 | 69-79 | 85-94 | 100-109 | 129-136 | 142-150 | 168-177 | 181-189 | 206-213 |
| DeepTMHMM | 13-18 | 20-24 | 29-39 | 52-62 | 68-79 | 82-95 | 99-111 | 126-137 | 141-150 | 166-174 | 180-189 | 204-212 |

Задание 2.
Краткая информация по выданному белку:
- Название на русском: неохарактеризованный белок BURPS1710b_3216 главного суперсемейства белков, осуществляющих мембранный транспорт
- Название на английском: Uncharacterized MFS-type transporter BURPS1710b_3216
- Идентификатор UniProt: Y3216_BURP1
- Из какого организма белок: Burkholderia pseudomallei (strain 1710b)
- В какой мембране располагается: внутренняя мембрана грамотрицательной бактерии
- Функция: мембранный транспорт
Последовательность белка была взята из Uniprot (файл с последовательностью).
Текстовая выдача метода DeepTMHMM.

| Номера участков | 1 | 1.5 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 8.5 | 9 | 10 | 11 | 12 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| DeepTMHMM | 22-43 | 56-72 | 88-109 | 119-139 | 156-176 | 183-203 | 224-245 | 256-275 | 286-304 | 309-329 | 345-367 | 375-394 | ||
| AlphaFold | 15-40 | 46-50 | 55-82 | 85-108 | 113-146 | 151-179 | 183-202 | 216-250 | 256-274 | 277-281 | 283-302 | 307-334 | 341-370 | 372-406 |
Так как PPM 3 не работал (или делал это слишком долго), участки для AlphaFold я взял, открыв файл, который нужно было загрузить на PPM 3, в Jalview в разделе Secondary Structure. В AlphaFold на две альфа-спирали больше, но эти участки довольно короткие, поэтому мне кажется, что они не соответсвуют действительности. Остальные участки совпадают, хотя их границы не такие точные как у предсказания бета-листов.