Практикум 7.
Подготовительный этап.
Подготовительный этап.
Для анализа были выбраны Reactome (по эстетическому критерию) и Human Protein Atlas (потому что очень красивый). Так как я ничего не знал про свои белки, то решил установить следующие:
- их функции (можно ли сформировать функциональные группы или нет?)
- представленность митохондриальных белков в списке
- источник списка (вдруг удастся установить, из какой ткани они выделены)
Перед анализом через базы данных своего списка белков я отсортировал его по названиям, чтобы определить наличие похожих (так как имена генов часто являются сокращениями от названия белков). Мне показалось, что можно выделить примерно 15 групп:
- ABAT
- ACACA, ACACB, ACADS, ACAT1, ACAT2
- ACOX1, ACOX3
- ACSS1, ACSS2, ACSS3
- ALDH6A1
- BCKDHA, BCKDHB
- DBT, DLD
- ECHDC1, ECHS1, EHHADH
- HADHA, HIBCH
- LDHA, LDHAL6A, LDHAL6B, LDHB, LDHC
- MCEE, MLYCD, MMUT
- PCCA, PCCB
- SUCLA2, SUCLG1, SUCLG2
Reactome
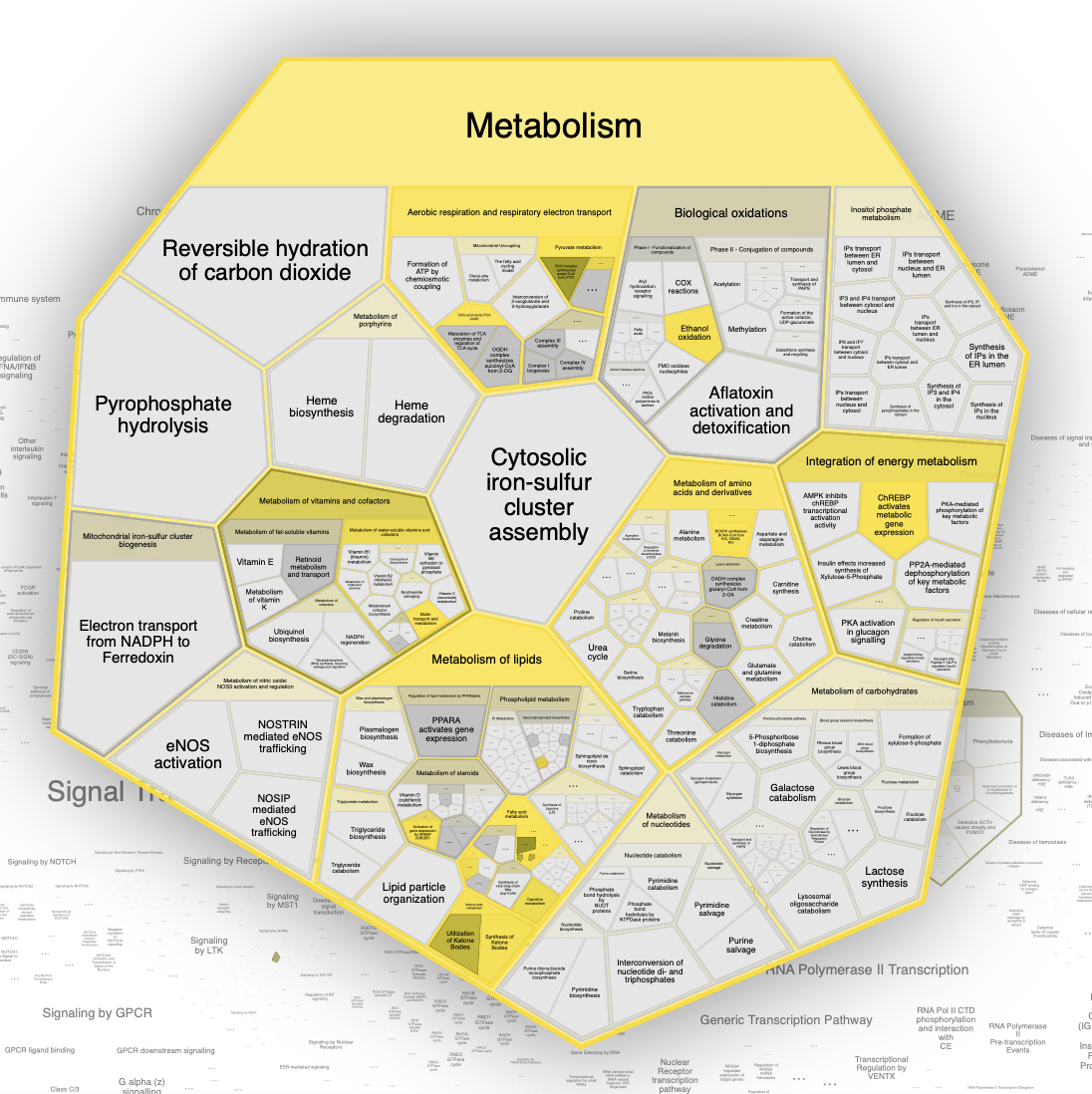
После того как я запустил анализа списка белков на «карте Reactome» выделилось 5 ячеек: Metabolism, Protein localization, Diseases of metabolism, Mitochondrial protein degradation, Plasma lipoprotein clearance.
Начнем анализ с блока Metabolism, так как он самый большой. В нем выделилось множество под блоков. Окислением этанола занимаются два белка: ACSS1, ACSS2. Следующим подблоком было кислородное дыхание. В группе, связанной с пируватом, были LDHA, LDHB, LDHC, DLD, LDHAL6A, LDHAL6B; с циклом Кребса — ACAT1, SUCLA2, SUCLG1, DLD. В подгруппу энергетического метаболизма попали гены ACACA, ACACB, которые играют роль в углеводном метаболизме, так как их экспрессия связана с белком ChREBP. Далее я изучил кластер белков, связанных с аминокислотами: ACAT1, ECHS1, DBT, BCKDHB, BCKDHA, ALDH6A1, DLD, HIBCH. Самая большая группа относилась к липидам.
Связанные с жирными кислотами: ACACA, ACACB, ACADS, ACOX1, ACOX3, ECHS1, EHHADH, HADHA, MCEE, MLYCD, MMUT, PCCA, PCCB
- Бета-окисление митохондриальных жирных кислот: ACADS, ECHS1, HADHA, MCEE, MMUT, PCCA, PCCB
- Метаболизм липидов пероксисом (тоже бета-окисление): ACOX1, ACOX3, EHHADH, MLYCD
- Метаболизм карнитина: ACACA, ACACB
Участвующие в синтезе и утилизации кетоновых тел: ACAT1, ACSS3
Активация экспрессии генов из-за SREBP (регулирует синтез холестерина; относится к стероидам): ACACA, ACACB
Также некоторые белки из моего списка (ACACA, ACACB, PCCA, PCCB) связаны с транспортом и метаболизмом биотина.
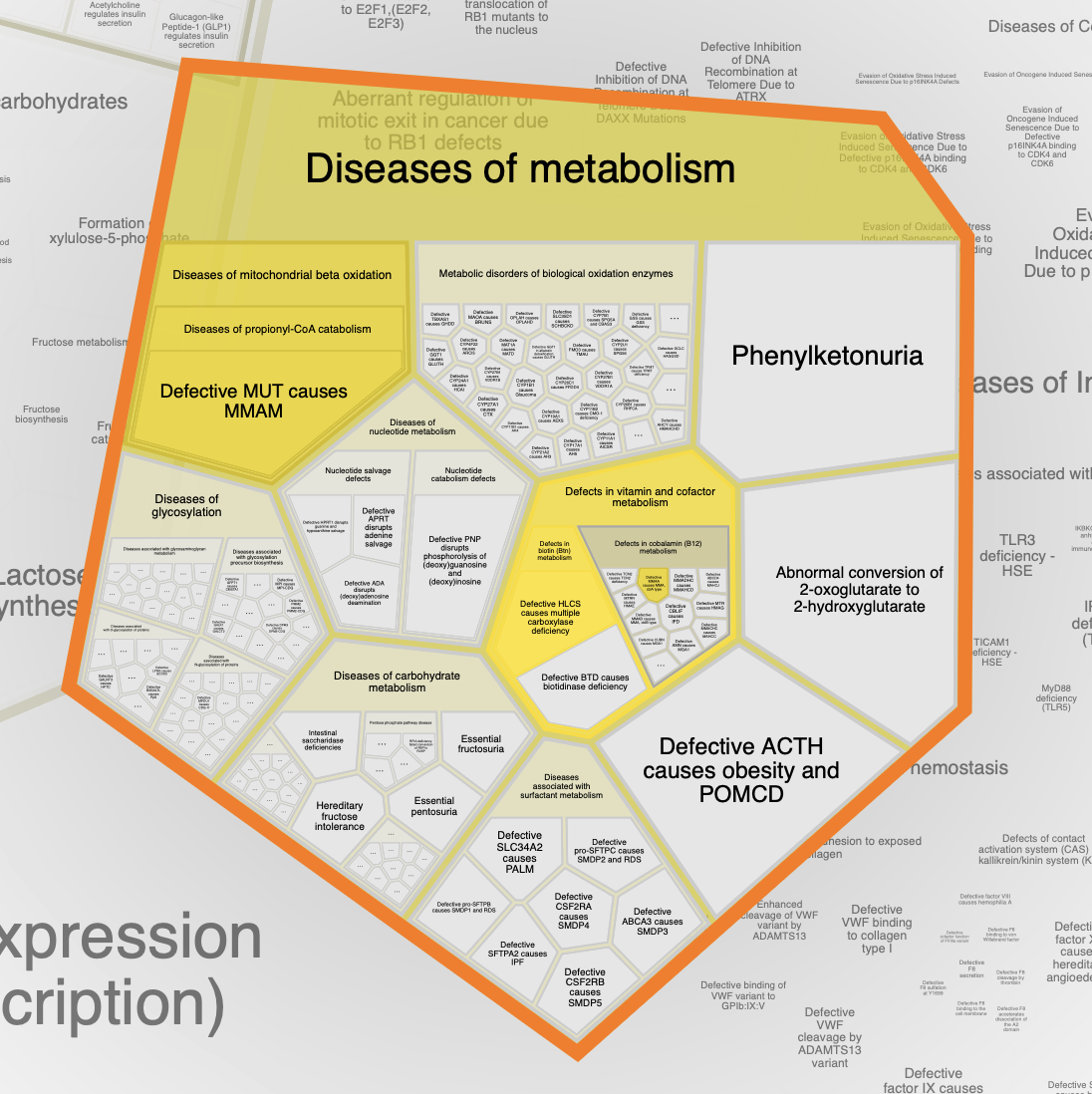
Далее я обратил свой взор к блоку «Disease», а точнее к его подблоку, о нарушениях связанных с метаболизмом. К нарушениям в митохондриях, а именно в бета-окислению, относится всего один белок MMUT. В группу дефектов, связанных с метаболизмом, попали ACACA, PCCA, PCCB (нарушение в метаболизме биотина), MMUT (последний связан с MMAA — белок, вовлеченный в транспорт кобалимина (кофермент B12) внутрь митохондрии).
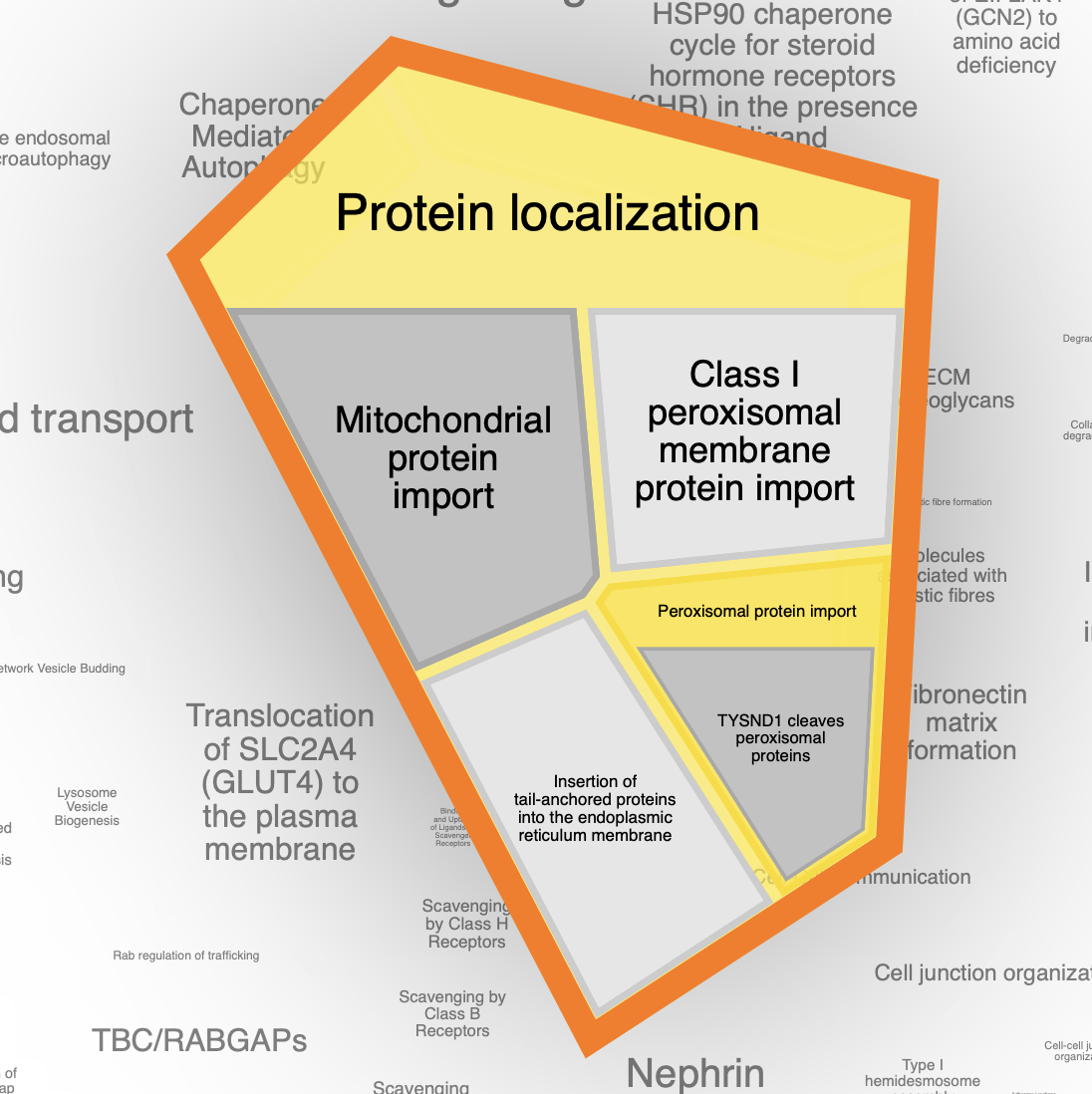
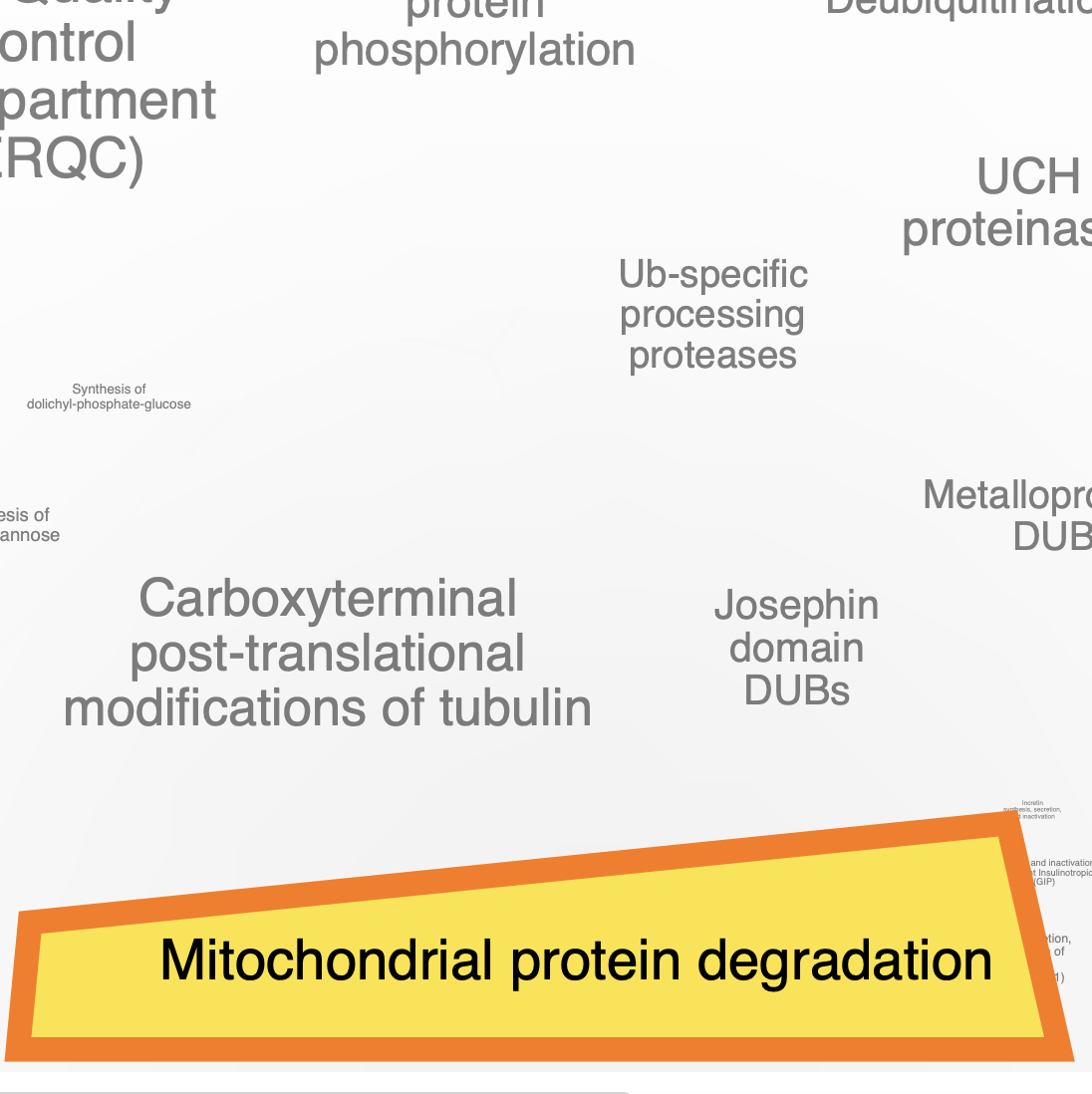
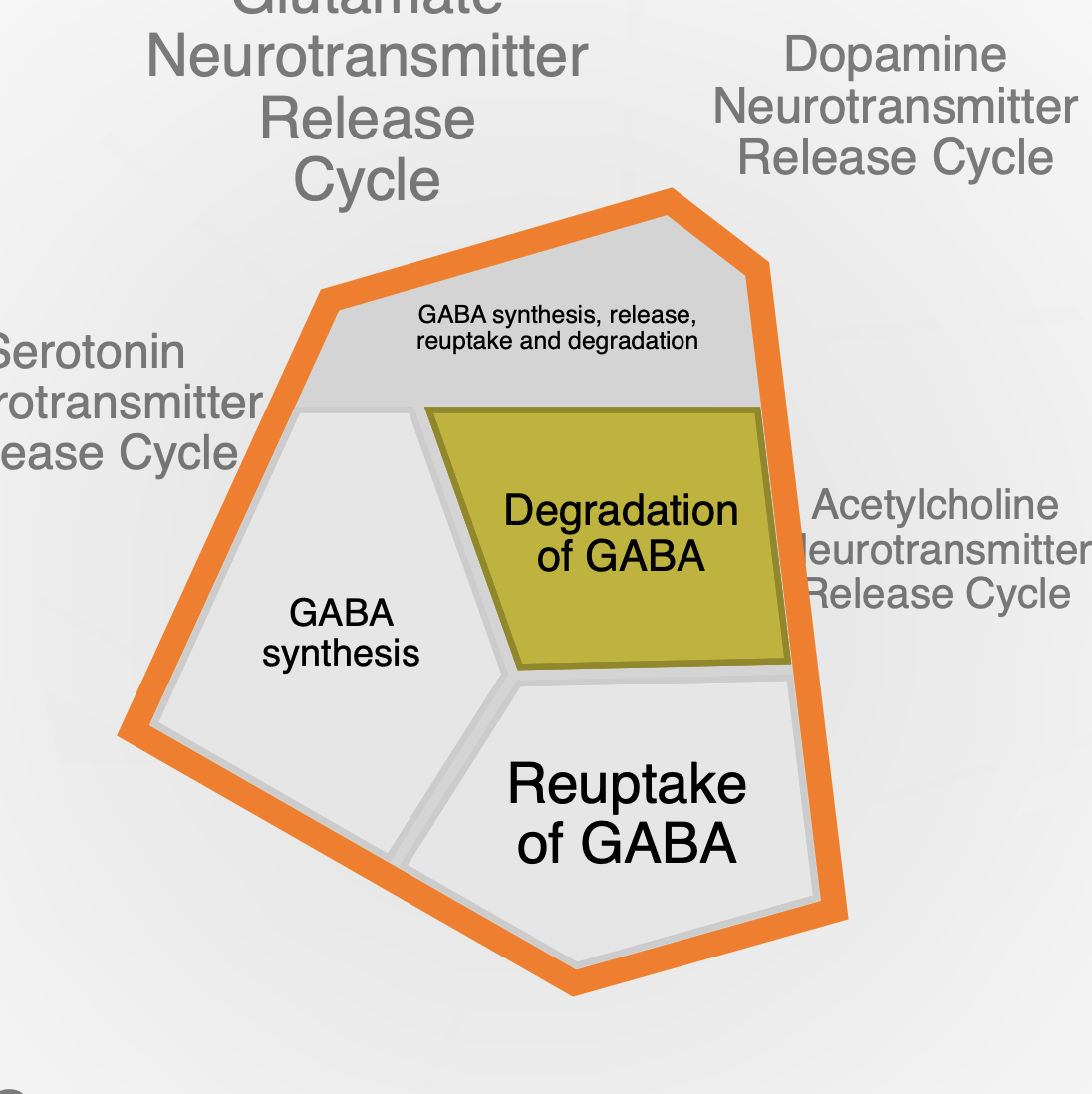
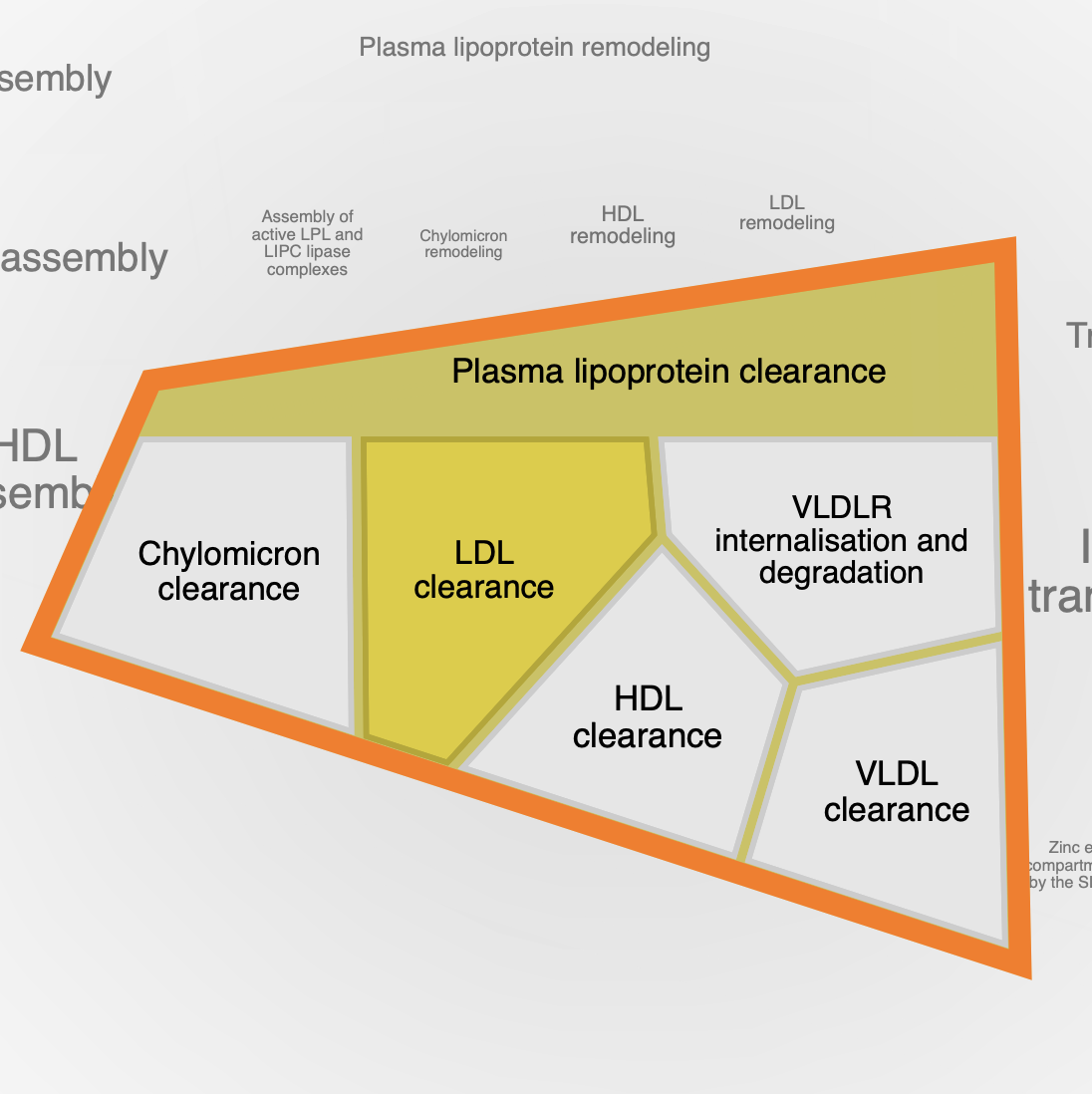
Про локализацию известно, что ACOX1 связан с пероксисомами. К деградацией митохондриальных белков относится довольно большая группа: ACAT1, PCCB, SUCLG2, DLD, DBT. Нашелся даже один белок (ABAT), который участвует в деградации GABA. Последний блок «Plasma lipoprotein clearance», относящийся к транспорту малых молекул, содержал в себе ACAT1 и ACAT2.
Выводы по анализу Reactome.
- В моем списке определено присутствуют белки, связанные с митохондриями: ACADS, LDHA, LDHB, LDHAL6A, LDHAL6B, LDHC, DLD, DBT, ACAT1, SUCLA2, SUCLG1, SUCLG2, HADHA, MCEE, MMUT, PCCB, и их довольно много (на мой скромный взгляд)
- Можно выделить несколько групп белков: митохондриальная, пероксисомальная (ACOX1, ACOX3, EHHADH, MLYCD), аминокислотная (ACAT1, ECHS1, DBT, BCKDHB, BCKDHA, ALDH6A1, DLD, HIBCH), номер 3 (ACACA, ACACB; эти белки всегда появлялись вместе, но делали это в разных процессах, поэтому я пока затрудняюсь дать функциональное название этой группе), окисление этанола (ACSS1, ACSS2), метаболизм кетоновых тел (ACAT1, ACSS3), метаболизм биотина (ACACA, ACACB, PCCA, PCCB), очистка липопротеинов в плазме (ACAT1, ACAT2) деградация GABA (ABAT). Белок ECHDC1 не был найден.
- Про локализацию этих белков (откуда они были выделены) или их общие черты (например, с каким процессом или заболеванием они связаны) сложно сказать что-нибудь конкретное. В списке много митохондриальных белков; есть белок расщепляющий GABA, но он всего один; большая группа пероксисомальных и аминокислотных. Поэтому логично предположить, что, если белки относятся к одной ткани, то там требуется большое количество энергии, нехватка которой приводит к каким-то заболеваниям.
Human Protein Atlas
ABAT
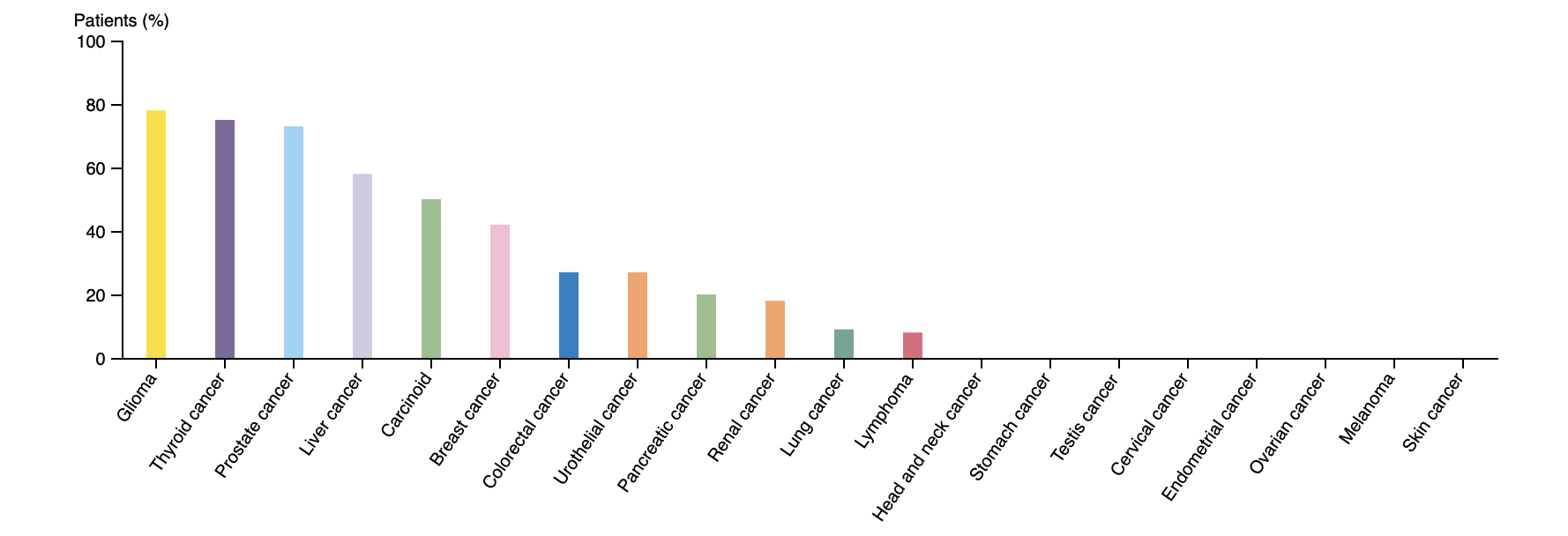

Первым белком, который я решил, проверить был ABAT, так как он единственный из 33, кто был связан с GABA. Его экспрессия замечена не только во многих отделах мозга, но и в других тканях и органах: почки, печень, поджелудочная железа и т.д. (В мозгу и первых трех органах из этого списка наблюдается повышенная экспрессия данного гена). Из приятного: он локализован в митохондриях, что позволяет занести его в группу связанную с этой органеллой. Есть даже информация о связанных с ним заболеваниях: при образовании глиомы, раке щитовидной железы, печени и предстательной железы наблюдается умеренная или высокая экспрессия мРНК этого белка.
ECHDC1
Так как Reactome не сумел его найти, то мне показалось логичным глянуть на него в атласе. Занимается он декарбоксилированием этилмалонил-КоА (токсичный метаболит). Данный белок не является тканеспецифичным, а локализован он в везикулах. Про заболевание я не заметил ничего интересного.
LDHAL6A и LDHAL6B
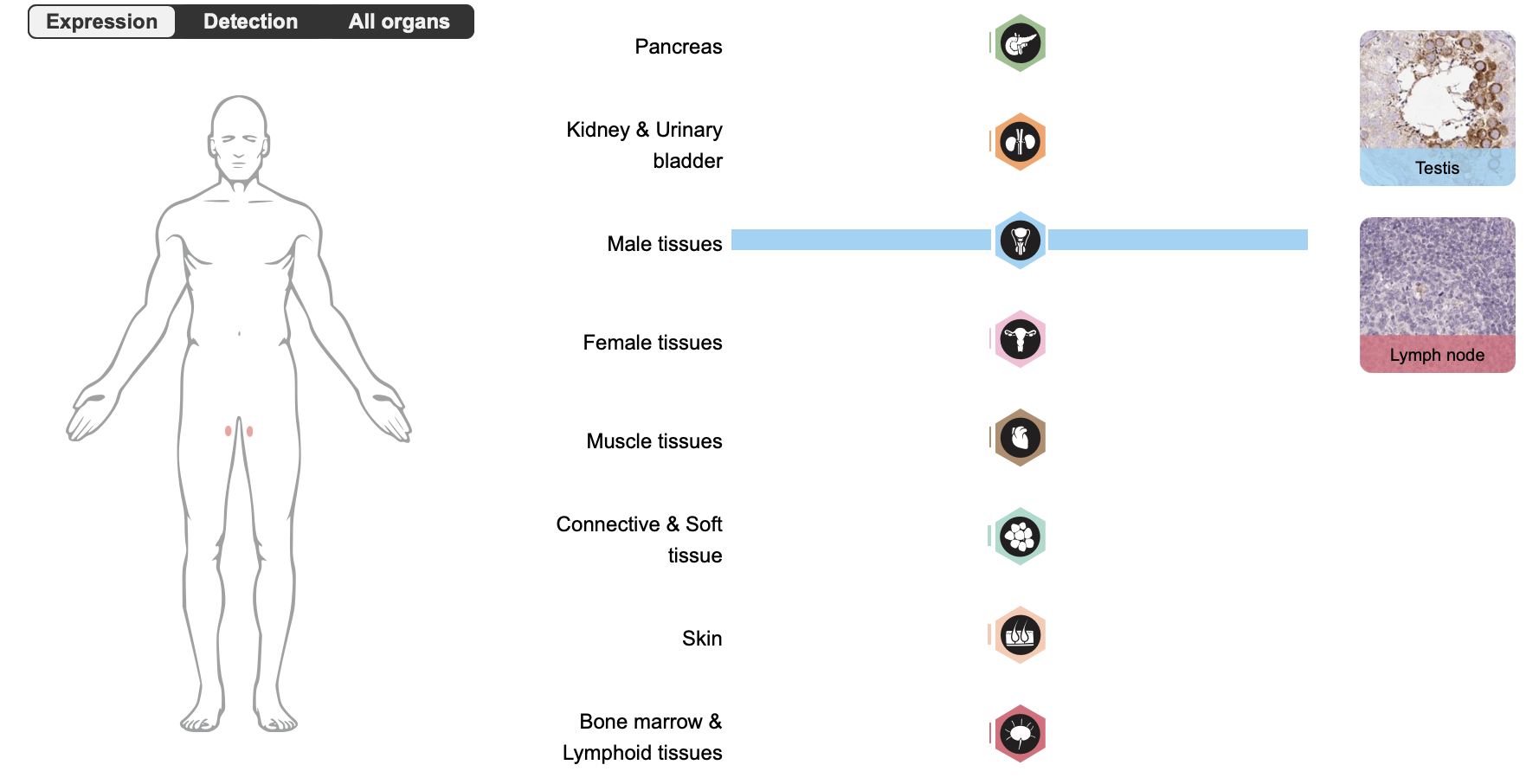
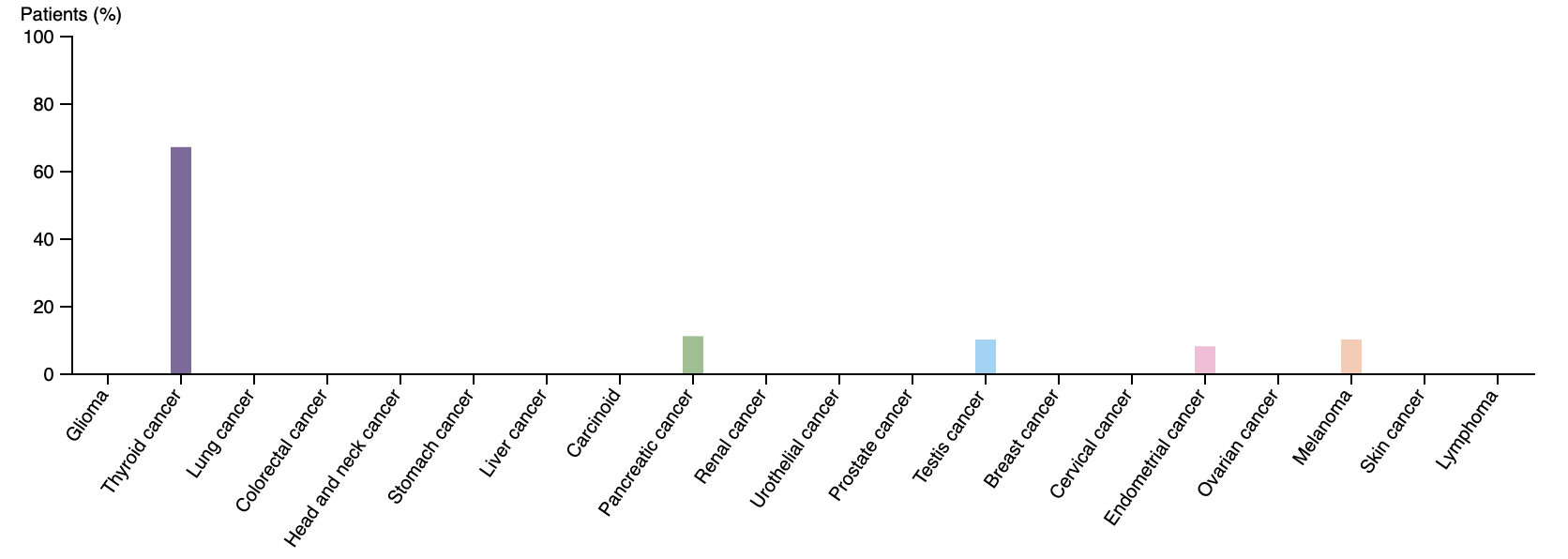
Выбрал я их почти случайно (игрался со String и заметил, что они почему-то не объединяются в один кластер). Они оба являются лактатдегидрогеназами А (превращают лактат в пируват) и локализованы только в семенниках. Это все, что известно о LDHAL6A. LDHAL6B описан лучше. Известно, что он локализован в цитоплазме и зафискированы случаи высокой или средней экспрессии данного белка при раке и семенника, щитовидной и поджелудочной желез (но это были единичные случаи в выборках малого объема).
ACACA и ACACB
Так как определить функциональное название для данной группы по информации из Reactome я не смог, то решил изучить их более детально. Они оба не являются ткани специфичными и встречаются в семенниках. Оба являются ацетил-КоА-карбоксилазами (синтезируют малонил-КоА, который нужен для регуляции бета-окисления жирных кислот в митохондриях; также для синтеза малонил-КоА требуется биотин). Про ACACA известна его локализация в клетке: в цитоплазме, ядрышках и на актиновых филаментах. Экспрессия ACACA была низкой только при раке семенников или глиоме; у ACACB высокая экспрессия наблюдалась только у одного пациента из 10 при раке печени.
Аминокислотная группа
Некоторые гены из этой групп по данным Reactome попадают в митохондриальную. Поэтому я решил проверить в Human Protein Atlas и выяснил, что для всех них показана локализация только в митохондриях для клеточной линии мыши. Таким образом мне показалось логичным объединить две эти группы, выделив аминокислотную кавычками.
Выводы по Human Protein Atlas и списку белков.
- Мой список белков связан с метаболизмом жирных кислот
- Данные белки могли быть из семенников (видимо их выделили для изучения нарушения в метаболизме жирных кислот)
- Можно выделить следующие группы: митохондриальная (бета-оксиление жирных кислот(ABAT, ACADS, LDHA, LDHB, LDHAL6A, LDHAL6B, LDHC, DLD, DBT, ACAT1, SUCLA2, SUCLG1, SUCLG2, HADHA, MCEE, MMUT, PCCB) + аминокислотная (ACAT1, ECHS1, BCKDHB, BCKDHA, ALDH6A1, HIBCH, DLD, DBT) + окисление этанола (ACSS1, ACSS2) + метаболизм кетоновых тел (ACAT1, ACSS3)), пероксисомальная (ACOX1, ACOX3, EHHADH, MLYCD), синтез малонил-КоА (ACACA, ACACB, PCCA, PCCB), очистка липопротеинов в плазме (ACAT1, ACAT2)