МИНИОБЗОР
Обзор генома/протеома Rhodococcus erythropolis R138
Петрова Анастасия
Факультет биоинженерии и биоинформатики, Московский государственный университет имени М. В. Ломоносова, Ленинские горы д. 1 стр. 73, 119234, Москва, Россия
РЕЗЮМЕ
Собраны и проанализированы стандартные сведения о геноме Rhodococcus erythropolis R138, получены и проанализированы кумулятивные GC-skew графики для хромосомы и плазмид, получена гистограмма длин белков и проверена гипотеза о случайности распределения генов по двум цепочкам ДНК: на 2 половине хромосомы преобладают гены, расположенные на "-" цепи, на всей кольцевой плазмиде - гены, расположенные на "+" цепи, получены и проанализированы статистические данные о генах РНК Rhodococcus erythropolis R138.
Сопроводительные материалы: Google Sheets, гистограмма, отдельные последовательности, genomic.fna, feature_table.txt.
ВВЕДЕНИЕ
Rhodococcus erythropolis R138 это штамм актинобактерий из порядка Mycobacteriales семейства Нокардий рода Rhodococcus. Это облигатно аэробные грамположительные бактерии, в геноме которых высоко содержание G-C пар оснований (60-62%) [1,2]. Их геном состоит из одной кольцевой хромосомы и 2 плазмид: линейной и кольцевой. Род Rhodococcus известен своим разнообразием метаболических путей, которое позволяет им расщеплять множество труднометаболизируемых соединений, таких как производные фенола, катехола, стирола. Такое разнообразие может быть вызвано вариативностью линейных плазмид, в разных штаммах R. erythropolis [3], а также кодированием в плазмидах и хромосоме большого количества гомологов различных ферментов, задействованных в схожих или идентичных метаболических путях [4]. В частности, катаболический путь γ-лактона позволяет Rhodococcus erythropolis R138 расщеплять N-ацилгомосерин лактоны - сигналы “чувства кворума” [5,6] у грамотрицательных бактерий мягкой гнили таких как Dickeya и Pectobacterium [7,8,2]. Это позволяет использовать их для биоконтроля в сельском хозяйстве и пищевой промышленности. Миниобзор включает в себя рассмотрение и анализ базовых особенностей генома и протеома бактерии с помощью методов биоинформатики. Исследуются кодирующие белок последовательности и составляющие их нуклеотиды, особенности распределения последовательностей по хромосоме и плазмидам бактерии и распределение белков
МАТЕРИАЛЫ И МЕТОДЫ
Данные о бактерии, такие как feature table, assembly stats, геном в fasta-формате были взяты из базы Национального Центра Биотехнологической информации (NCBI). Для анализа данных в работе использовался сервис Google Sheets. Для вычисления кумулятивного графика GC-skew использовался сервис Webskew. Использовались также команды биоинформатического пакета EMBOSS установленного на kodomo и скрипты на языке программирования Python. Данные, используемые в работе, таблицы Google Sheets и доступны по ссылкам и приложены в разделе Сопроводительные материалы.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЯ
В ходе работы были проведены:
- Сбор и анализ стандартных сведений о геноме Rhodococcus erythropolis R138.
- Исследование показателей GC-skew.
- Сбор и анализ статистических данных о белках протеома Rhodococcus erythropolis R138.
- Сбор и анализ статистических данных о генах РНК Rhodococcus erythropolis R138.
3.1 Стандартные сведения о геноме
Мы разбили командой seqret -ossingle2 файл genomic.fna, на 3 части: nz_cp007255.1 (хромосома), nz_cp007256.1 (кольцевая плазмида pCRE138), nz_cp007257.1 (линейная плазмида pLRE138). Далее с помощью команд wordcount -wordsize1 и geecee мы собрали стандартные сведения о геноме. Результаты представлены в таблицах.
| Таблица 1. Длина кольцевой хромосомы и плазмид Rhodococcus erythropolis R138 | ||||
| Хромосома | Плазмида pCRE138 | Плазмида pLRE138 | Всего | |
|---|---|---|---|---|
| Длина (в парах оснований) | 6236862 | 91729 | 477915 | 6806506 |
Геном Rhodococcus erythropolis R138 состоит из одной кольцевой хромосомы длиной 6236862 пары оснований, и 2 плазмид: линейной (pLRE138) и кольцевой (pCRE138) длиной по 477915 и 91729 пар оснований соответственно.
| Таблица 2. Нуклеотидный состав генома Rhodococcus erythropolis R138 | ||||
| Хромосома | Плазмида pCRE138 | Плазмида pLRE138 | ||
|---|---|---|---|---|
| A | 1 168 101 (18.7% ) | 17 712 (19.3%) | 93 617 (19.6%) | |
| C | 1 945 666 (31.2%) | 27 592 (30.1%) | 142 103 (29.7%) | |
| G | 1 950 420 (31.3%) | 28 089 (30.6%) | 146 465 (30.6%) | |
| T | 1 172 675 (18.8%) | 18 336 (20.0%) | 95 730 (20.0%) | |
| G-C пары | 62.47% | 60.70% | 60.38% | |
Как видно из таблицы, второе правило Чаргаффа выполняется для всех трех последовательностей: содержание гуанина приблизительно равно содержанию цитозина, а содержание аденина - содержанию тимина. Высокое содержание G-C пар свидетельствует повышенной устойчивостью к денатурации и является отличительной особенностью Актинобактерий.
| Таблица 3. Стоп-кодон состав Rhodococcus erythropolis R138 | |||
| Chromosome | pCRE138 | pLRE138 | |
|---|---|---|---|
| TGA | 3990 | 62 | 295 |
| TAA | 537 | 8 | 48 |
| TAG | 1153 | 20 | 70 |
Как видно из таблицы, для всех 3 последовательностей преобладает кодон TGA, реже всего встречается TAA, что скорее всего объясняется высоким показателем содержания GC для данной бактерии
3.2 Исследование показателей GC-skew
С помощью сервиса WebSkew мы получили кумулятивные GC-skew графики для хромосомы и плазмид.
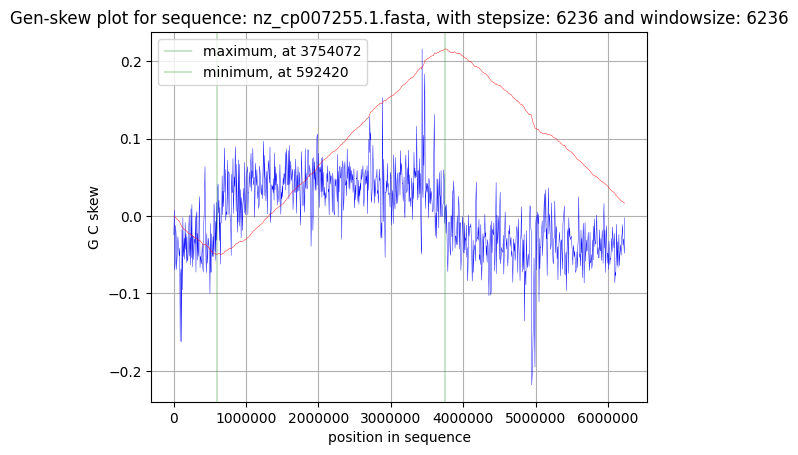
Если предположение о том, что точка начала репликации соответствует точке минимума GC-skew, а точке максимума - точка конца, верно, что oric хромосомы находится около 59240 позиции, а ter - около 3754072 позиции.
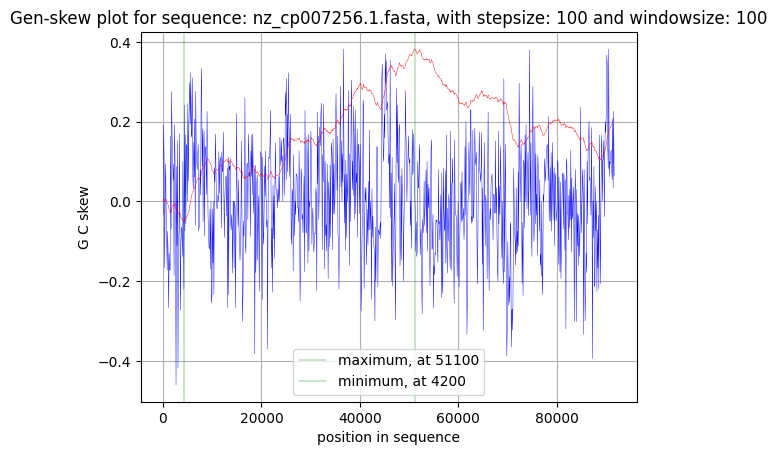
Если предположение о том, что точка начала репликации соответствует точке минимума GC-skew, а точке максимума - точка конца, верно, что oric плазмиды находится около 4200 позиции, а ter - около 51100 позиции.
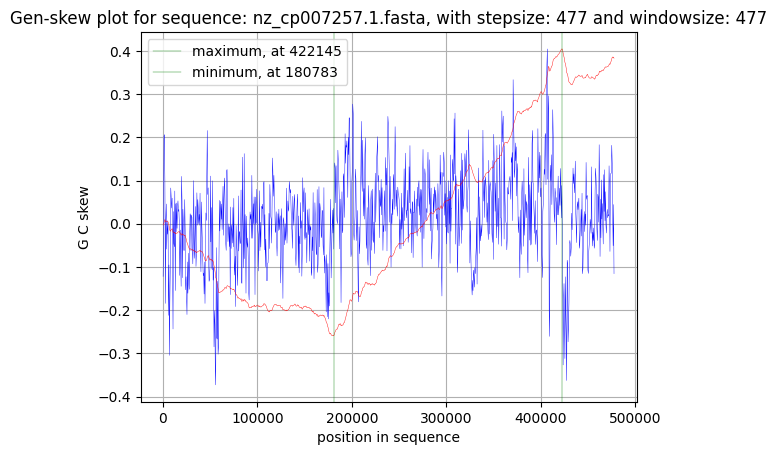
Если предположение о том, что точка начала репликации соответствует точке минимума GC-skew, а точке максимума - точка конца, верно, что oric плазмиды находится около 180783 позиции, а ter - около 422145 позиции.
3.3 Статистические данные о белках протеома
С помощью сервиса Google Sheets и файла feature_table.txt мы проанализировали протеом бактерии
3.3.1 Исследование распределения генов белков в протеоме
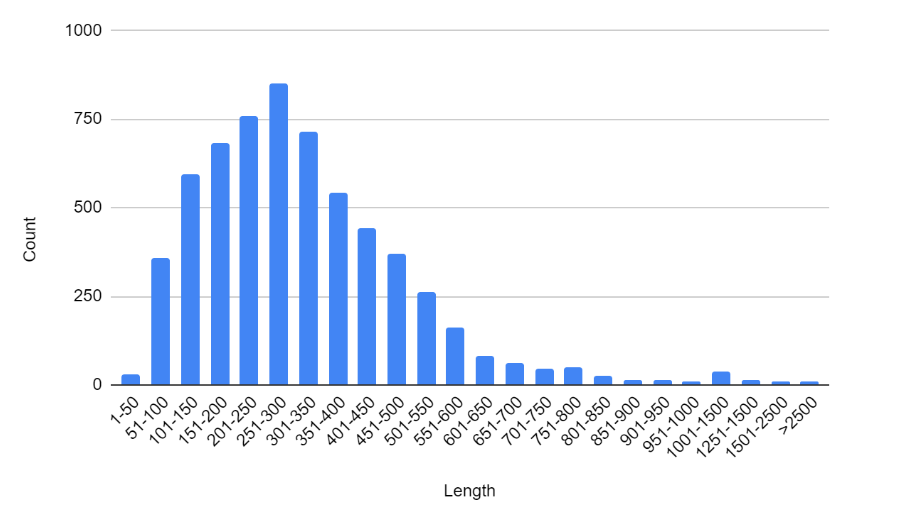
Средняя длина белков находится в диапазоне между 250 и 300 аминокислот. Таких белков 849. Группой самых длинных белков оказались синтетазы нерибособальных пептидов. Таких белков 8, они находятся на хромосоме и имеют длину от 4619 до 10611 аминокислот. Самый короткий белок - гипотетический белок на линейной плазмиде длиной 28 аминокислот. Самый короткий известный белок - прекурсор микофактоцина на хромосоме длиной 31 аминокислоту.
| Таблица 4. Распределение белков в геноме Rhodococcus erythropolis R138. | ||||
| Тип белков | Chromosome | pCRE138 | pLRE138 | Всего |
|---|---|---|---|---|
| Всего | 5661 | 89 | 391 | 6141 |
| Рибосомальные | 69 | 0 | 0 | 69 |
| Гипотетические | 628 (11,09%) | 47 (52,81%) | 106 (27,11%) | 781 (12,72%) |
| Транспортные | 555 (9,80%) | 0 (0,00%) | 31 (7,93%) | 586 (9,54%) |
Примечательно, что все рибосомальные белки находятся на хромосоме. При этом на кольцевой плазмиде нет ни рибосомальных, ни транспортных белков, но высока доля гипотетических.
3.3.2 Проверка гипотезы о случайности распределения генов по двум цепочкам ДНК.
С помощью критерия знаков проверим гипотезу о случайности распределения генов по двум цепочкам ДНК. За значимое отклонение принимается показатель меньше 0,05.
| Таблица 5. Распределение генов белков по хромосоме. | |||
| Генов белков | Chromosome | ||
|---|---|---|---|
| "+" цепь | "-" цепь | Критерий знаков | |
| Всего | 2762 | 2899 | 0,07066586456 |
| 1 половина | 1491 | 1389 | 0,05981397543 |
| 2 половина | 1271 | 1510 | 0,000006309547634 |
В то время как показатель на всей хромосоме находится в пределах нормы для случайного распределения, рассмотрев её 2 половины, становится очевидным, что на 2 половине распределение случайным считать нельзя, преобладают гены, расположенные на "-" цепи.
| Таблица 6. Распределение генов белков по плазмиде pCRE138. | |||
| Генов белков | pCRE138 | ||
|---|---|---|---|
| "+" цепь | "-" цепь | Критерий знаков | |
| Всего | 56 | 33 | 0,01918660866 |
| 1 половина | 31 | 16 | 0,03998605683 |
| 2 половина | 25 | 17 | 0,2799562385 |
Для всей кольцевой плазмиды распределение генов нельзя считать случайным, ввиду низкого показателя критерия знаков. Из таблицы видим явное преобладание генов на "+" цепи.
| Таблица 7. Распределение генов белков по плазмиде pLRE138. | |||
| Генов белков | pLRE138 | ||
|---|---|---|---|
| "+" цепь | "-" цепь | Критерий знаков | |
| Всего | 177 | 214 | 0,06853117694 |
| 1 половина | 91 | 100 | 0,5627984858 |
| 2 половина | 86 | 114 | 0,05596574049 |
3.4 Статистические данные о генах РНК
С помощью сервиса Google Sheets и файла feature_table.txt мы собрали данные о генах РНК. Все РНК находятся на хромосоме, поэтому таблица не разделяется по последовательностям.
| Таблица 8. Распределение генов РНК в геноме Rhodococcus erythropolis R138. | |
| Гены белков | 6141 |
|---|---|
| Гены РНК | 71 |
| рРНК | 15 |
| тРНК | 53 |
Помимо указанных типов РНК, в геноме Rhodococcus erythropolis R138 также присутствуют 2 некодирующие РНК (РНК, входящая в частицу узнавания сигнала и компонент класса A РНКазы P) и одна транспортно-матричная РНК
ЗАКЛЮЧЕНИЕ
Собраны и проанализированы стандартные сведения о геноме Rhodococcus erythropolis R138, получены и проанализированы кумулятивные GC-skew графики для хромосомы и плазмид, получена гистограмма длин белков и проверена гипотеза о случайности распределения генов по двум цепочкам ДНК: на 2 половине хромосомы преобладают гены, расположенные на "-" цепи, на всей кольцевой плазмиде - гены, расположенные на "+" цепи, получены и проанализированы статистические данные о генах РНК Rhodococcus erythropolis R138.
СПИСОК ЛИТЕРАТУРЫ
- de Carvalho, C.C.; da Fonseca, M.M. The remarkable Rhodococcus erythropolis. Appl. Microbiol. Biotechnol. 2005, 67, 715–726. [Google Scholar] [CrossRef]
- Kwasiborski A, Mondy S, Beury-Cirou A, Faure D. 2014. Genome sequence of the quorum-quenching Rhodococcus erythropolis strain R138. Genome Announc. 2014 Mar 27;2(2):null - PubMed
- Kwasiborski A, Mondy S, Chong T-M, Chan K-G, Beury-Cirou A, Faure D. Core genome and plasmidome of the quorum-quenching bacterium Rhodococcus erythropolis. Genetica. 2015;143(2):253–261. doi: 10.1007/s10709-015-9827-4. - DOI - PubMed
- van der Geize, R.; Dijkhuizen, L. Harnessing the catabolic diversity of rhodococci for environmental and biotechnological applications. Curr. Opin. Microbiol. 2004, 7, 255–261. [Google Scholar] [CrossRef]
- d’Angelo-Picard C, Faure D, Penot I, Dessaux Y. 2005. Diversity of N-acyl homoserine lactone-producing and -degrading bacteria in soil and tobacco rhizosphere. Environ. Microbiol. 7:1796–1808. 10.1111/j.1462-2920.2005.00886.x [PubMed] [CrossRef] [Google Scholar] [Ref list]
- Uroz S, D’Angelo-Picard C, Carlier A, Elasri M, Sicot C, Petit A, Oger P, Faure D, Dessaux Y. 2003. Novel bacteria degrading N-acylhomoserine lactones and their use as quenchers of quorum-sensing-regulated functions of plant-pathogenic bacteria. Microbiology 149:1981–1989. 10.1099/mic.0.26375-0 [PubMed] [CrossRef] [Google Scholar] [Ref list]
- Kwasiborski A, Mondy S, Chong T-M, Barbey C, Chan K-G, BeuryCirou A, Latour X, Faure D (2015) Transcriptome of the quorum-sensing signal-degrading Rhodococcus erythropolis responds differentially to virulent and avirulent Pectobacterium atrosepticum. Heredity. doi:10.1038/hdy.2014.121