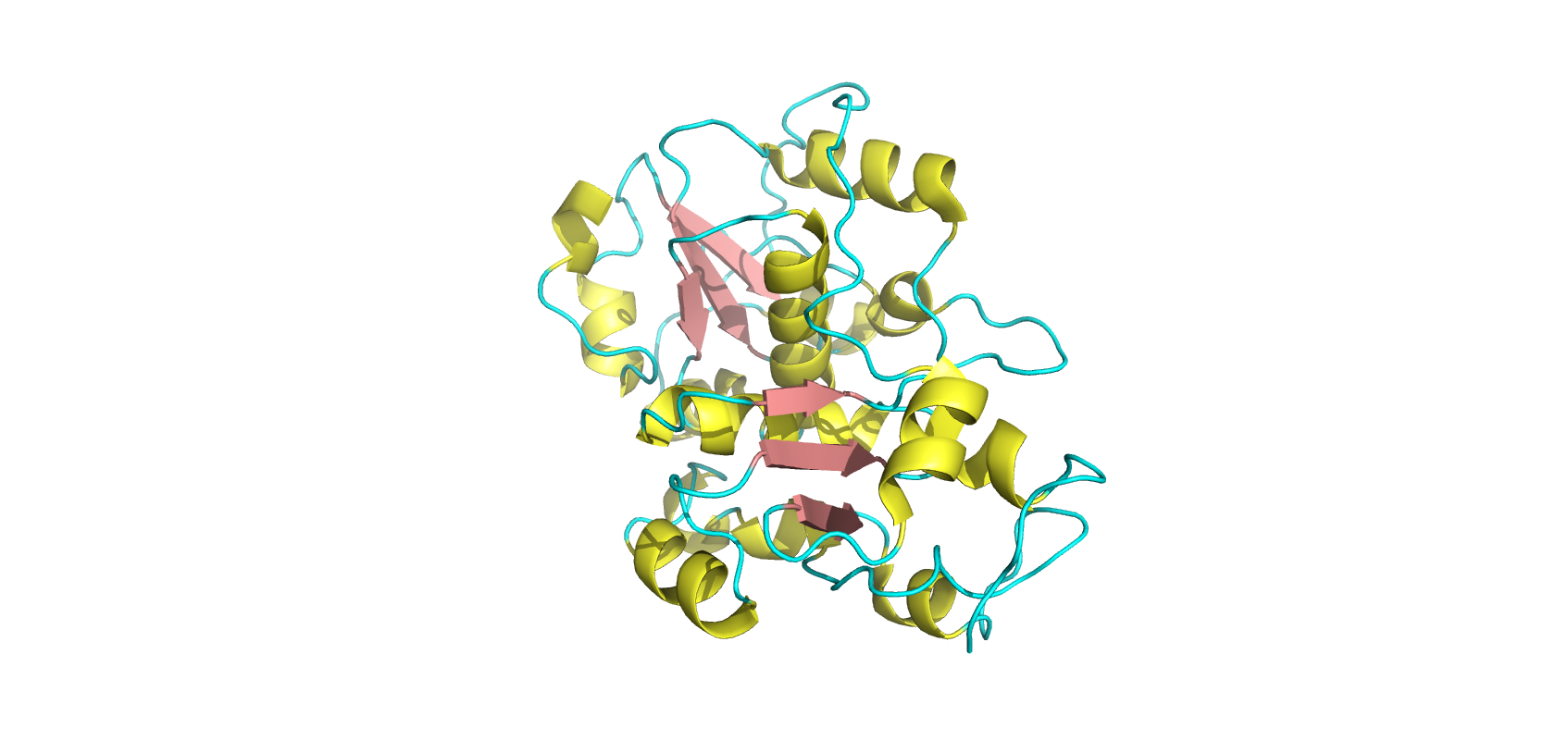Представленный тип макромолекул: белок, фермент -- тиосульфат-сульфотрансфераза (Thiosulfate sulfurtransferase).
В белке представлена одна полимерная цепь (А). В ее структуре больше представлены альфа-спирали.
В биологическую единицу (мономер) входит одна цепь (А):
"REMARK 350 BIOMOLECULE: 1
REMARK 350 AUTHOR DETERMINED BIOLOGICAL UNIT: MONOMERIC
REMARK 350 SOFTWARE DETERMINED QUATERNARY STRUCTURE: MONOMERIC
REMARK 350 SOFTWARE USED: PQS
REMARK 350 APPLY THE FOLLOWING TO CHAINS: A"
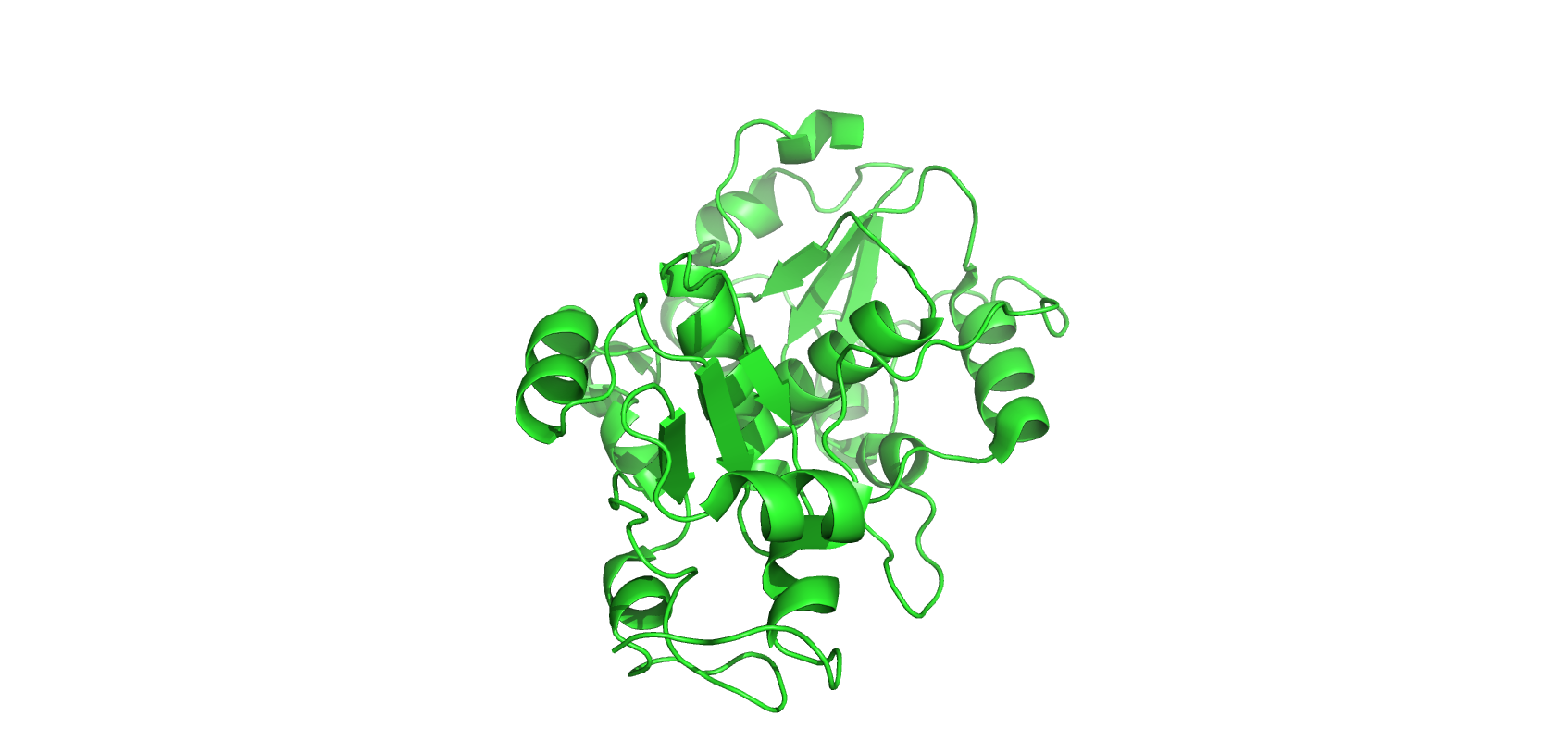
Рис.1 Структура тиосульфат-сульфотрансферазы
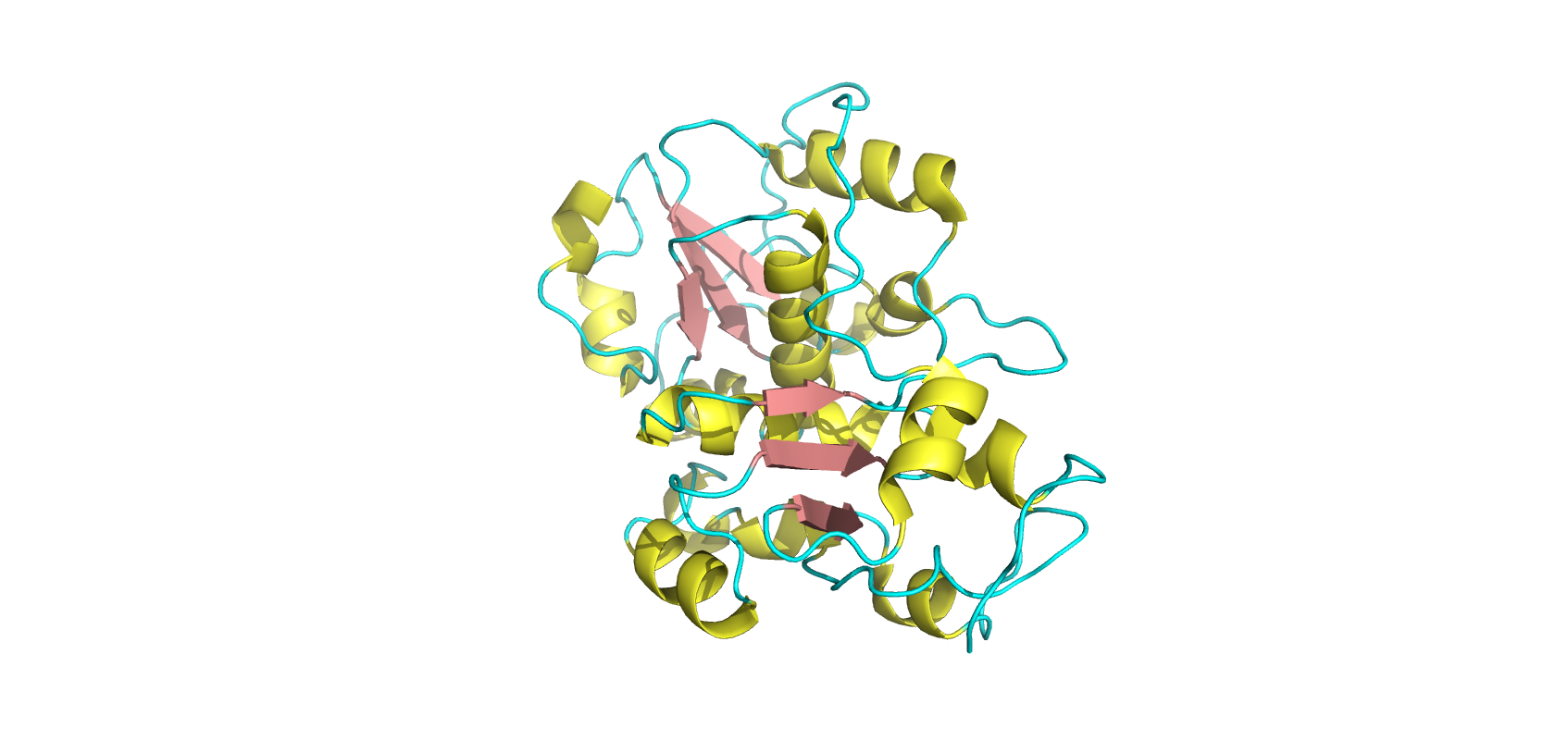
Рис.2 Структура тиосульфат-сульфотрансферазы. Покраска по элементам вторичной структуры.
Данная макромолекула относится к Azotobacter vinelandii.
В моём белке представлена только одна цепь А. Так что будем работать с ней.
Uniprot id: P52197.
Функция данной цепи: катализ реакции thiosulfate + hydrogen cyanide = thiocyanate + sulfite + 2 H+.
Мутаций в последовательности относительно референсного из Uniprot нет.
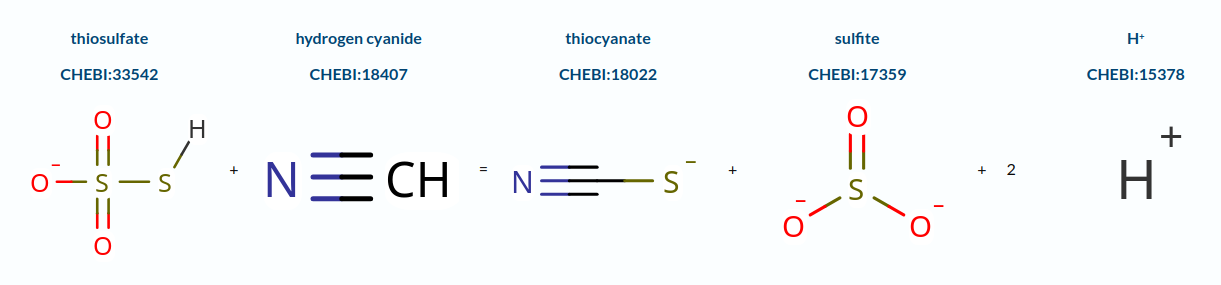
Рис.3 Реакция, катализируемая данным ферментом
В составе цепи есть модифицированные аминокислотные остатки. В позиции 230 цистеин преобразован в s-меркаптоцистеин:
"MODRES 1E0C CSS A 230 CYS S-MERCAPTOCYSTEINE"
Вырезка из файла PDB про модифицированные аминокислоты
Малые молекулы в записи:
SO4 (SULFATE ION);
EDO (1,2-ETHANEDIOL or ETHYLENE GLYCOL);
MG (MAGNESIUM ION);
Вырезка из файла PDB про малые молекулы
Описанные частицы, возможно, имеют отношение к функции белка (так как в его структуре в активных центрах есть сайты связывания для каждой из частиц), а также были необходимы в процессе получения его структуры. Тем не менее, сайты были предсказаны программно и практически очевидно из функций белка, что EDO - артефакт кристаллизации, а SO4 - лиганд, представленный в активном центре (см Рис. 4)
"Prior to
data collection crystals were transferred into a stabilizing
solution containing 1.8 M MgSO4, 50 mM MES (pH 6.0),
and 5 % (v/v) ethanediol." (doi:10.1006/jmbi.2000.3651)
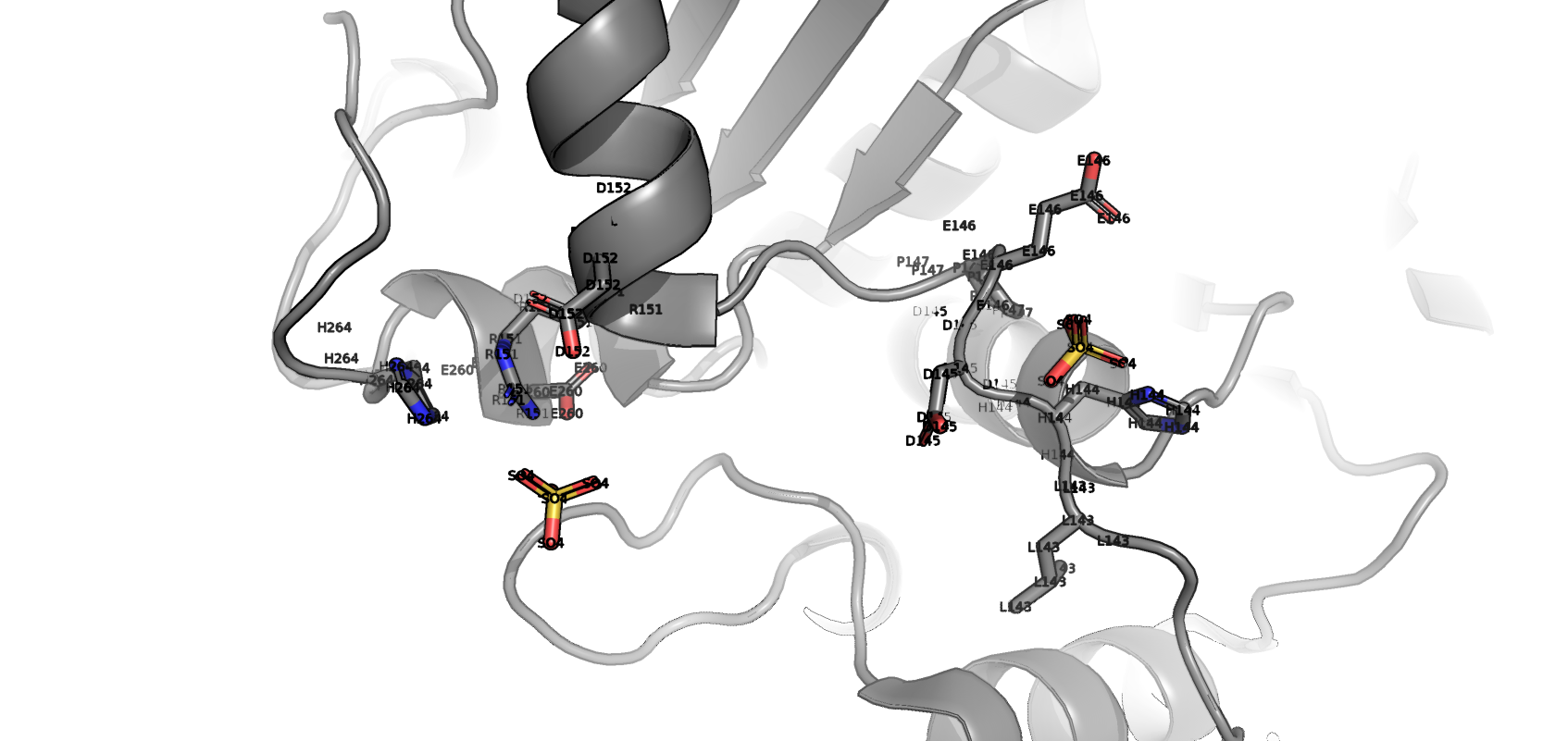
Рис.4 Активный центр белка с лигандом SO4