2. Знакомство с поисковыми системами ENA и NCBI
Выбранный белок: insulin
Поиск в NCBI Nucleotide:
Запрос: insulin[Title]
| Тип поиска |
Количество записей |
| Всего записей |
45020 |
| Гены с таким словом в названии |
4,129 |
| мРНК белков с таким словом в названии |
36,529 |
| Записей генов в GenBank |
4,109 |
| Записей генов в RefSeq |
20 |
| Записей мРНК в RefSeq |
31,341 |
| Записей мРНК в GenBank |
5,185 |
Только для последовательностей человека:
insulin[Title] AND Human[Organism]
| Тип последовательности |
База данных |
Количество записей |
| genomic DNA/RNA |
RefSeq |
19 |
| genomic DNA/RNA |
GenBank |
1,453 |
| mRNA |
RefSeq |
218 |
| mRNA |
GenBank |
3,875 |
| Всего |
5,565 |
Комментарий: Не все эти записи действительно содержат последовательности, кодирующие белки с соответствующей функцией. Например, встречается Insulin-like growth factor.
Поиск в ENA:
Запросы:
description="insulin" AND mol_type="mrna"
tax_tree(9606) AND description="insulin"
| Тип записи |
Количество записей |
| мРНК |
12756 |
| Гены белков человека с insulin в названии |
9341 |
Комментарий по DDBJ: Сайт DDBJ не прогружается на моем компьютере. А ENA УЖАСНО неудобен. В дальнейшем я буду использовать удобный и быстрый NCBI.
3. Органеллы в выбранной сборке генома
В таблице Chromosomes для Refseq sequences обнаружена хромосома MT (митохондриальный геном).
| Параметр |
Значение |
| Идентификатор записи (ACCESSION) |
NC_029855 |
| Тип органеллы |
мт |
| Число кодирующих последовательностей (CDS) |
35 |
| Число генов рРНК |
3 |
| Число генов тРНК |
15 |
| Число псевдогенов |
2 |
4. Поиск дельта-субъединицы АТФ-синтазы
С помощью текстового поиска в файле последовательности белков каннабиса найден белок с идентификатором XP_030491925.1, который является дельта-субъединицей АТФ-синтазы.
Идентификатор нуклеотидной записи, к которой относится ген, кодирующий данный белок: NC_083601
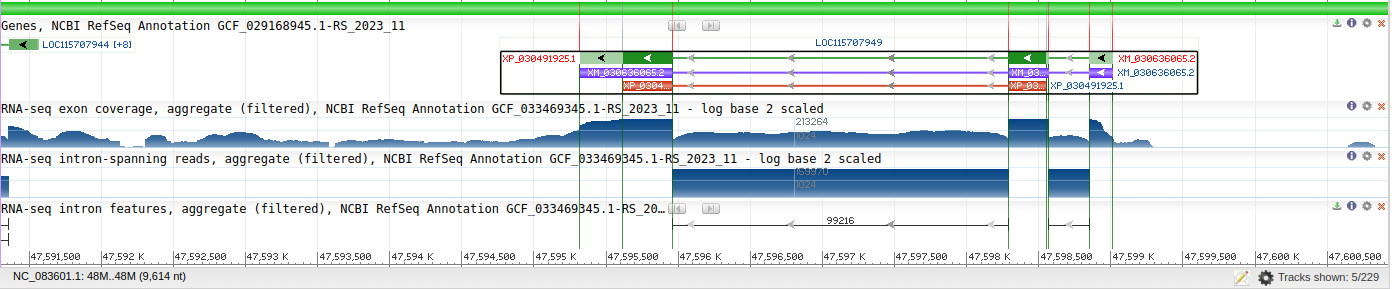
Рис 1. Изображение окрестности участка (47595317 – 47599013), содержащего кодирующую белок часть гена δ-субъединицы АТФ-синтазы, из геномного браузера.
5. Поиск BLAST в таксоне Apoidea
Выбранный таксон для поиска: Apoidea
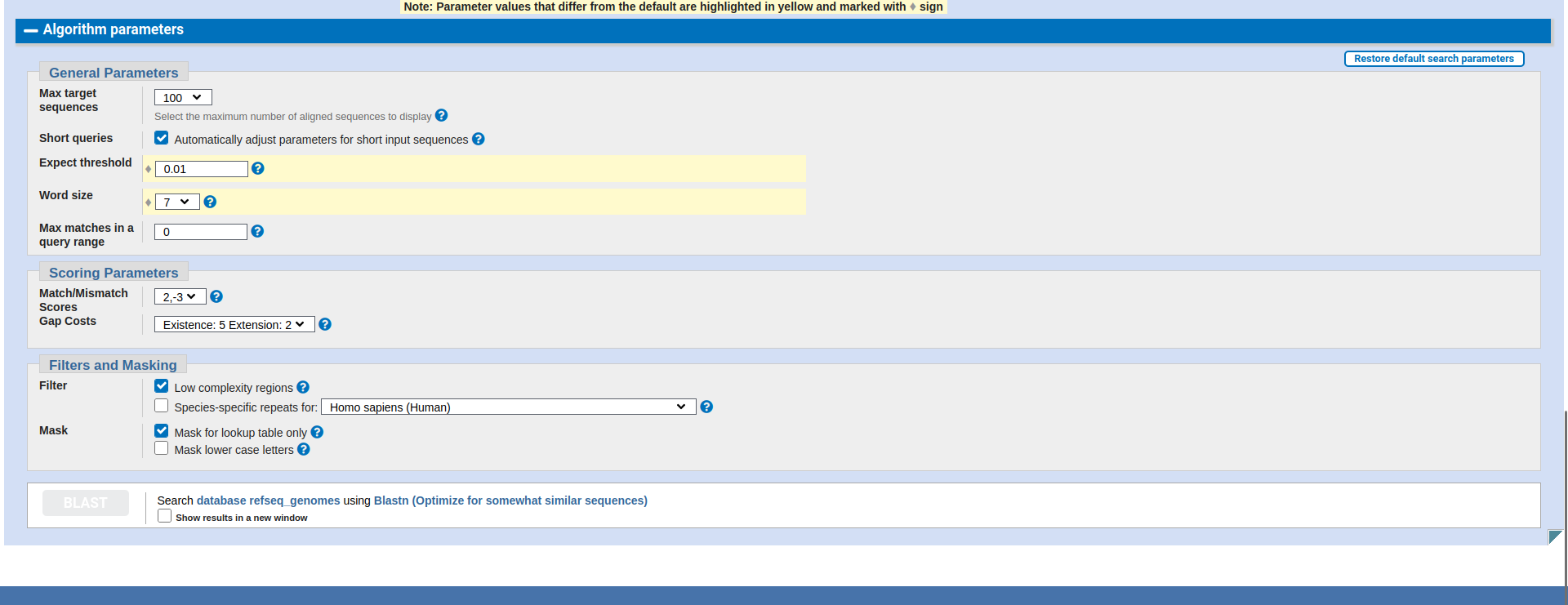
Рис. 2. Параметры поиска для алгоритма blastN. Использовалась база данных refseq_genomes. Число сборок в ней, входящих в выбранный таксон – 38.

Рис. 3. Результаты выдачи алгоритма blastn для последовательности субъединицы АТФ-синтазы каннабиса. Поиск производился по таксону Apoidea. Найдено 100 соответствий длиной 50-80 нуклеотидов, абсолютное большинство из них выровнялось на конец последовательности. Это объясняется удаленностью таксонов.

Рис. 4. Параметры поиска для алгоритма tblastN. Использовалась база данных refseq_genomes. Число сборок в ней, входящих в выбранный таксон – 38.

Рис. 5. Результаты выдачи алгоритма tblastn для последовательности субъединицы АТФ-синтазы каннабиса. Поиск производился по таксону Apoidea. Найдено 38 соответствий, большинство из них длиной 40-80 аминокислот. Значит, что в последовательности данной субъединицы есть консервативные участки даже для столь далёких таксонов.
6. Локальный поиск BLAST рРНК Escherichia coli
Индексация последовательностей генома каннабиса:
makeblastdb -in ./GCF_029168945.1_ASM2916894v1_genomic.fna -dbtype nucl
Локальный поиск BLAST для рРНК:
blastn -task blastn -query ./16s.fa -db ./GCF_029168945.1_ASM2916894v1_genomic.fna -outfmt 7 -evalue 0.005 -word_size 7 -out 16sblast
blastn -task blastn -query ./23s.fa -db ./GCF_029168945.1_ASM2916894v1_genomic.fna -outfmt 7 -evalue 0.005 -word_size 7 -out 23sblast
Ссылка на выдачу BLAST для 16s рРНК
Ссылка на выдачу BLAST для 23s рРНК
В случае 16s получилось 80 находок, в случае 23s - 82. Это много, однако учитывая, что выравнивал я на геном растения, выдача кажется адекватной.
Далее, т.к. для такого числа находок посчитать число гомологов будет крайне сложно, я решил сделать это для одного "хромосомы" NC_029855.1 - митохондрии Cannabis sativa
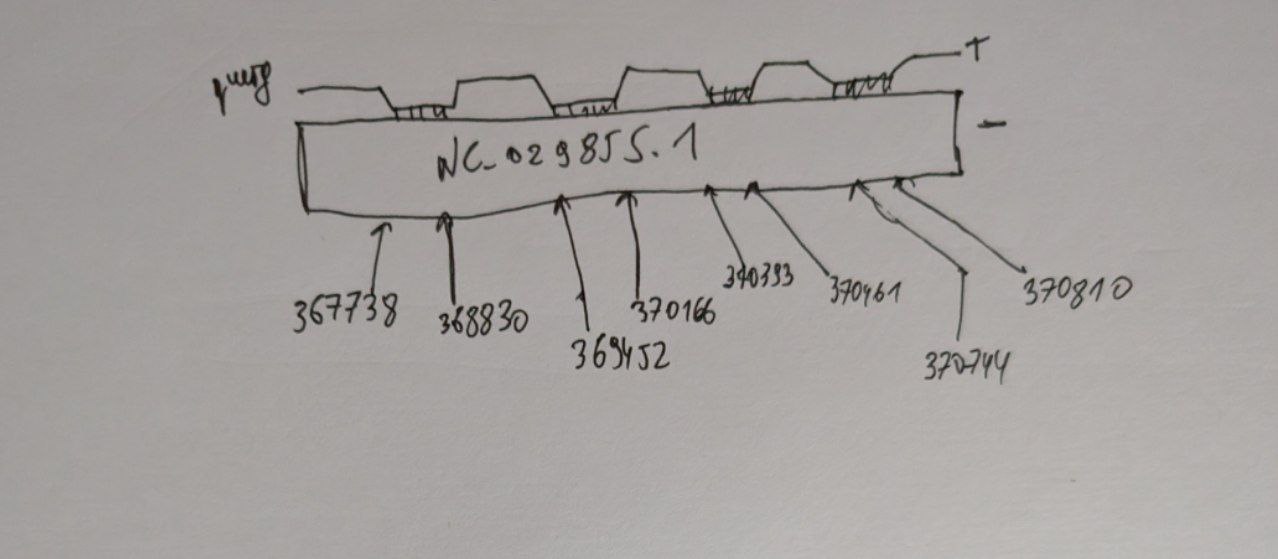
Рис. 6 Пример гомологов последовательности 23S рРНК E. coli, найденного на NC_029855.1 - митохондрия Cannabis sativa
На координатах 369249..370983, судя по таблице для митохондрии, находится ген rrnL, кодирующий 23s рРНК. Получается, трём выровненым находкам для query соответствует один гомолог.
Для находки, выровненной на 368830..367738, нет генов в таблице, включающих данные координаты в принципе. Однако e-value у неё 0. Взлянем на выравнивание:
>NC_029855.1 Cannabis sativa mitochondrion, complete genome
Length=415602
Score = 710 bits (787), Expect = 0.0
Identities = 848/1141 (74%), Gaps = 60/1141 (5%)
Strand=Plus/Minus
Результаты почти идеальные. Можно сказать, что данной находке соответствует гомолог.
Итак, на митохондрии Cannabis sativa найдено два гомолога
Также я решил провести исследование для NC_083607.1 - хромосомы 7 Cannabis sativa.
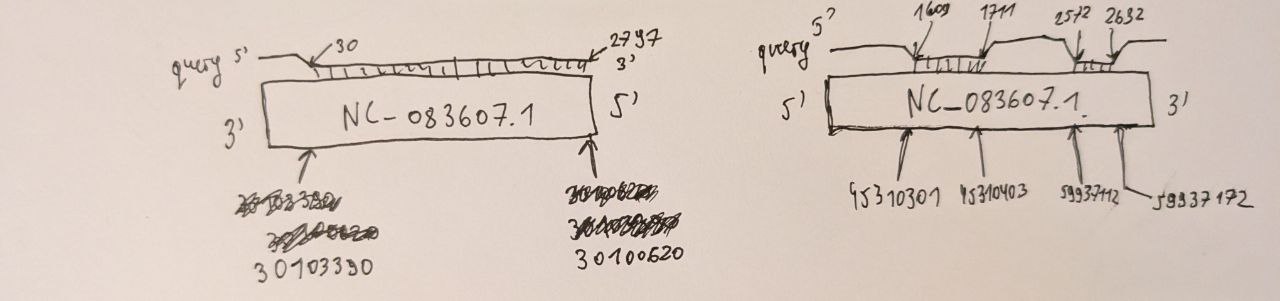
Рис. 7 Пример гомологов последовательности 23S рРНК E. coli, найденного на NC_083607.1 - хромосома 7 Cannabis sativa.
На координатах 30103390..30100620 на хромосоме нет ни одного гена, судя по таблице локальных особенностей. Однако её e-value 0. Взглянем на выравнивание:
>NC_083607.1 Cannabis sativa cultivar Pink pepper isolate KNU-18-1
chromosome 7, ASM2916894v1, whole genome shotgun sequence
Length=63199550
Score = 803 bits (890), Expect = 0.0
Identities = 1935/2861 (68%), Gaps = 183/2861 (6%)
Strand=Plus/Minus
Итак, можно сказать, что данным координатам соответствует гомолог, просто он не аннотирован
На координатах 45310301..45310403, судя по таблице локальных особенностей, также нет никаких генов, что странно, ведь e-value для находки, выровненной на данные координаты, очень низок(3.41e-13). Данные противоречивы, однако можно предположить, что находке соответствует гомолог, просто он не аннотирован в таблице.
На координатах 59937112..59937172 снова не нашлось никаких генов, а e-value по прежнему крайне низкий (2.15e-09). Снова можно предположить, что находке соответствует гомолог.
Итак, на 7 хромосоме каннабиса найдено 3 гомолога 23s рРНК E.coli
7. Сравнение геномов штаммов Halapricum desulfuricans
Выбранные штаммы: HSR12-2 и HSR-Bgl
Идентификаторы полностью собранных хромосом:
- NZ_CP064788.1 для HSR12-2
- NZ_CP064789.1 для HSR-Bgl
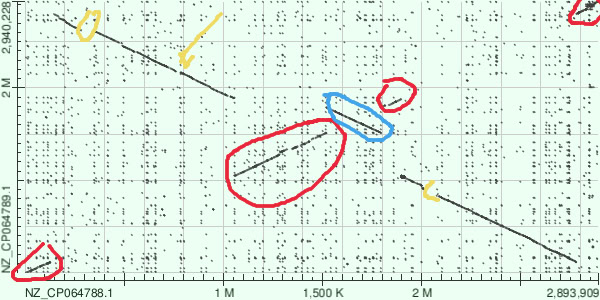
Рис. 8. Дотплот выравнивания мегабластом со стандартными параметрами. Жёлтым показаны делеции, красным – транслокации + инверсии, синим – транслокации.
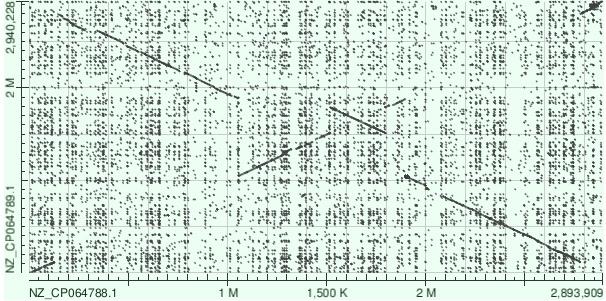
Рис. 9. Дотплот выравнивания blastn со стандартными параметрами.
Комментарий: Tblastx выдавал ошибку. Причина – имеет низкое значение длины слова по умолчанию + ищет по транслятам.







