С помощью меню Calculate -> Principal component analysis открылась 3D-схема
на которой сходство между последовательностями отображается расстоянием между ними.
Исходя из этой картинки, я выбрал как наименее похожие последовательности
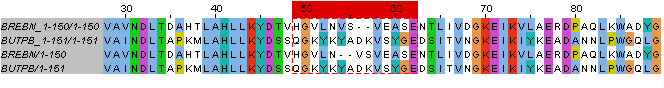
BREBN и BUTPB, fasta-последовательности которых можно увидеть в
файле. Исходное выравнивание можно увидеть в окне
"Base alignment".

| Выравнивание | Процент консервативных колонок | Процент колонок со сходными выравниваниями | Число гэпов |
| Полученное из множественного | 72 | 20 | 2 |
| Полученное программой needle | 73 | 21 | 2 |
| Полученное программой needle с изменением штрафа | 74 | 21 | 4 |
| Полученное программой water | 73 | 21 | 2 |
| Полученное программой water с изменением штрафа | 73 | 21 | 2 |
| Негомологичные последовательности, программа needle | 39 | 30 | 10 |
| Негомологичные последовательности, программа water | 26 | 15 | 6 |