Информация по домену
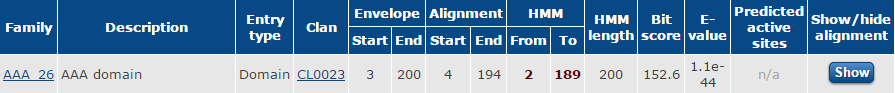
Для выполнения заданий я использовал белок YP_002729488 (Refseq ID)
бактерии Sulfurihydrogenibium azorense Az-Fu1.
В результате поиска Pfam нашёл в последовательности белка всего один
домен - AAA domain из семейства AAA_26.


Seed-выравнивание можно увидедь, перейдя по этой ссылке.
Консенсусную последовательность seed-выравнивания я получил с помощью
JalView, посмотреть её можно перейдя
сюда.
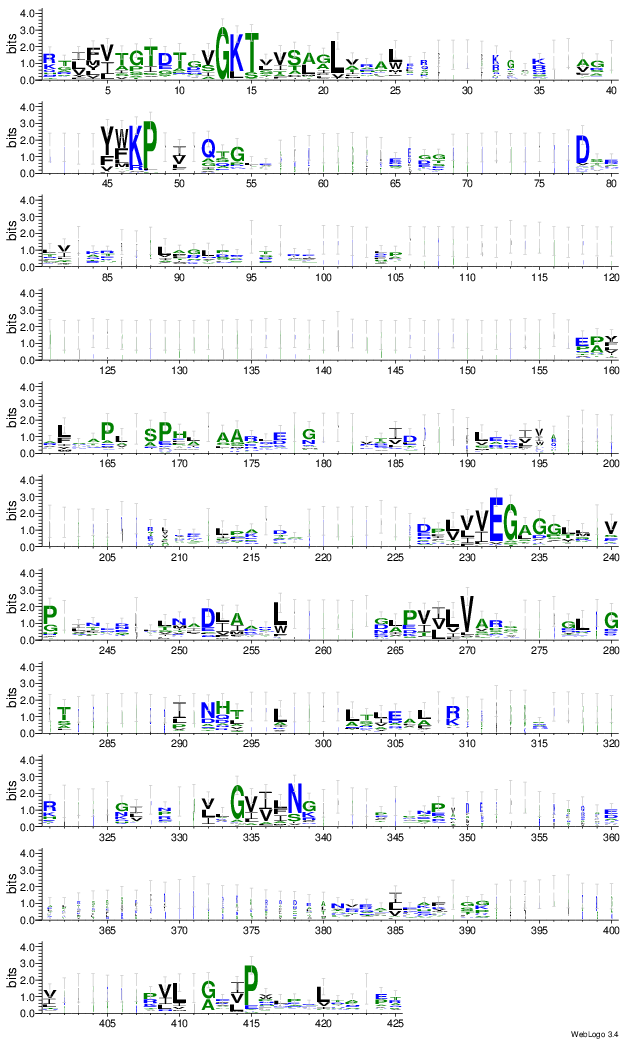
Logo можно было получить с помощью всё того же JalView, но теперь я
воспользовался сервисом
http://threeplusone.com/weblogo/.
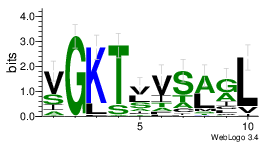
Открыв seed-выравнивание в JalView я выбрал блок выравнивания, с которым
буду работать в следующем задании.

Посмотреть последовательность выравнивания блока можно открыв
файл.
Посмотрев на блдок выравнивания и пользуясь сервисом
ScanProsite для проверки
написанных паттернов, я составил эти самые паттерны.
Слабый паттерн:
[VAIST]-G-[KL]-[TS]-[VFLSTI]-[VAILTF]-[STLAIC]-[ALMST]-[LIAGWSC]-[LVI].
Сильный паттерн:
[VIS]-G-[KL]-[TS]-[VLS]-[VAIL]-[STLAC]-[ALM]-[LIAGC]-[LV].
Количество находок по слабому паттерну в БД SwissProt - 1225
последовательностей. Не все последовательности из seed-выравнивания были
представлены в результатах поиска в ScanProsite, потому что некоторые
последовательности seed-выравнивания просто не рецензированы.
Количество находок по сильному паттерну в БД SwissProt - 100
последовательностей.
Довольно странно, что усиление такого короткого паттерна так сильно
повлияло на число находок в ProSite. Скорее всего, это связано с тем, что для
усиления паттерна я удалял из него все те аминокислоты, которые в колонке
выравнивания встречаются редко или не вписываются в общие свойства большинства
аминокислот в колонке (например, фенилаланин в шестой колонке).
JalView-проект с выравниваниями можно скачать по
ссылке.
Главная страница