Выравнивание геномов
Задание 1.
Для работы в данном практикуме я выбрал "свою" бактерию Sulfurihydrogenibium azorense и
родственную ей (из того же рода) Sulfurihydrogenibium subterraneum. Однако blastn отказался
строить карту локального сходства, поскольку геном второй бактерии секвенировани не полностью
(статус сборки - "Scaffold"). Тогда я взял другую бактерию из того же рода (и единственную, у кого, помимо
"моей" бактерии, полностью секвенирован геном) - Sulfurihydrogenibium sp. YO3AOP1. Полученную
карту локального сходства можно увидеть ниже.
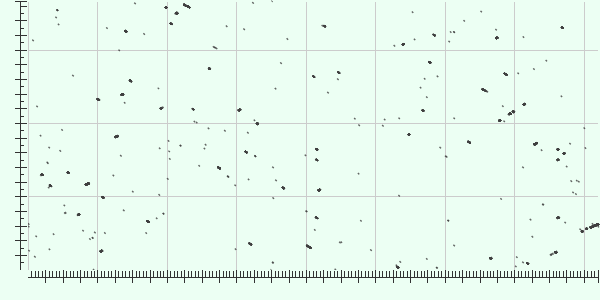
Удручающая картина... Поэтому я решил взять две другие бактерии, скажем, из рода Brucella -
Brucella sp. штамма F60 и Brucella abortus штамма BDW.

По оси абсцисс - поледовательность первой хромосомы Brucella abortus, по оси ординат - Brucella sp.
На этой карте локального сходства красными прямоугольниками я отметил инверсии, зелёными - вставки
или делеции.
Геном Sulfurihydrogenibium azorense, геном
Sulfurihydrogenibium subterraneum, геном
Sulfurihydrogenibium sp. YO3AOP1, геном
Brucella sp. штамма F60, геном
Brucella abortus штамма BDW.
Задание 2.
Для выполнения данного задания я взял три штамма бактерии Helicobacter pylori:
Helicobacter pylori strain 26695, Helicobacter pylori strain J99 и Helicobacter pylori
strain 51. Цель данного задания - сравнить геномы близкородственных бактерий (в нашем случае - штаммов
одного вида) и описать их сходства и различия с помощью построения нуклеотидного пангенома (NPG).
Вся работа производилась в директории npge.
NPG-explorer работает с файлами формата .tsv, в которых лежит информация, в какой базе данных и с
каким файлом будет работать эксплорер. Создав такой файл genomes.tsv,
я запустил команды npge Prepare и npge Examine - подготовил последовательности к работе.
Затем командой npge -g npge.conf получил параметры программы в файле
npge.conf, после чего изменил значение MIN_IDENTITY в нём на
рекомендованное в файле examine/identity_recommended.txt.
Потом я запустил команды npge MakePangenome и npge CheckPangenome, в результате чего
получил файл pangenome/pangenome.bs - файл с блоками.
Затем командой npge PostProcessing я получил множество разных файлов с информацией о
пангеноме в целом и конкретных блоках.
Информацию о количестве g-блоков - синтеничных участков, - я взял в файле
blocks.gbi. Всего g-блоков получилось 41. Информация
же о последовательности блоков в каждом геноме есть в файле
blocks.blocks.
Выравнивание блоков в трёх геномах в виде Excel-таблицы.
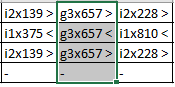
Совпадающих блоков во всех трёх геномах - 14.
Интересый случай: у штаммов 26695 и J99 блок g3x657 раправлен прямо, а у штамма 51 - обратно (инверсия). Такая
картина вообще характерна для данного штамма: у него многие блоки раправлены в обратную сторону.
Информация о количестве s-блоков - коровых (стабильных) блоках, - хранится в файле
pangenome.info. Количество s-блоков - 373, суммарная
длина - 1369661 нуклеотидов (73,42% генома), процент консервативных позиций - 0.920691 (92,0691%).
Информация о повторах (r-блоки - повторы по крайней мере в одном геноме) хранится в файле
pangenome/pangenome.bi (
таблица блоков, в том числе и r-блоков).
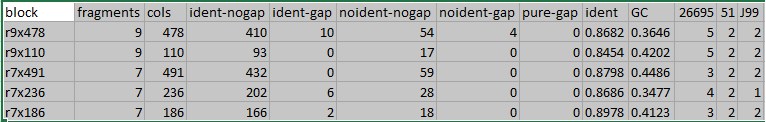
Фрагмент таблицы с r-блоками
Видно, что, скажем, r-блок r9x478 имеет 7 фрагментов и 441 позиция в выравнивании блока.
Информация об h-блоках - "полустадильных" блоках (по одному фрагменту лишь в части геномов), - можно
найти всё в том же файле pangenome/pangenome.bi.
Соответственно, если в каком-то геноме такого блока нет, а в других есть, то в этом геноме на этом месте -
длинная делеция.
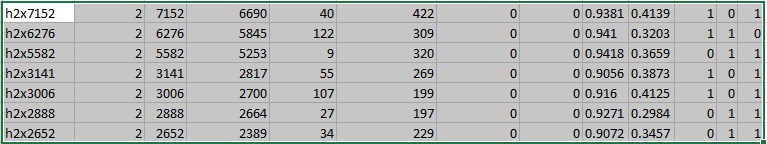
Фрагмент таблицы с h-блоками
u-блоки - уникальные последовательности из одного генома, у них нет гомологов среди всех геномов,
кроме самой себя.
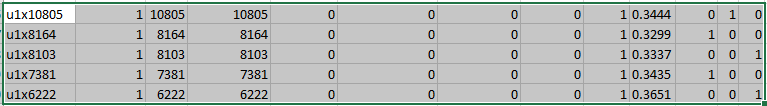
Фрагмент таблицы с u-блоками
Для аннотирования я выбрал самый крупный u-блок - u1x10805. В выдаче blastn видно, что последовательность
этого блока встречается преимущественно у Helicobacter pylori разных штаммов, лишь в паре случаев этот блок
встречается у другой бактерии этого же рода - Helicobacter cetorum. Исходя из этого, можно заключить,
что данная последовательность скорее всего не является результатом горизонтального переноса из другого вида.

Фрагмент выдачи blastn по последовательности u-блока u1x10805
Довольно интересный пример несовпадения аннотаций: гены в одном и том же фрагменте одного и того же
блока (s3x19027) у трёх разных штаммов Helicobacter pylori имеют разные названия: membrane fusion
protein (mtrC), uroporphyrinogen decarboxylase и putative wfflux transporter. Видно даже по названиям, что это
не разные наименования одного и того же гена, это именно названия разных генов, кодирующих совсем разные
белки. Точнее, у штаммов 26695 и J99 это может быть один и тот же белок, тогда как уропорфириногенкарбоксилаза
у штамма 51 сюда совсем не вписывается. Хотя имеются основания предполагать, что это ортологичные гены -
они начинаются в одном и том же месте и имеют одинаковую длину.



Ссылка на главную страницу
© Головачев Ярослав










