Гены прокариот
Задание 1.
Для работы в данном практикуме я выбрал последовательность генома вируса бешенства
Rabies virus. Геном данного организма полностью секвенирован, и его можно найти на
сайте NCBI, RefSeq AC генома
- NC_001542.
Аннотация генома вируса бешенства,
последовательность генома данного вируса и
его аннотированных белков в fasta-формате.
Аннотированных белков в геноме вируcа - 5 штук.
Таблица аннотированных генов в геноме вируса.

Таксономия вируса бешенства Rabies virus
Я зарегистировался на сайте RAST, после чего начал новый
проект, перейдя по ссылке Your Jobs > Upload new job, после чего следовал указаниям и запустил
задание по аннотированию генов вируса. Через некоторое время RAST выполнил работу, и мною был скачан
результат в виде таблицы. Также я скачал аннотацию генома с сервиса
RAST в формате gbk.
Обе таблицы с генами (с сервисов NCBI и RAST) я объединил в одну, после
чего сравнил результаты.
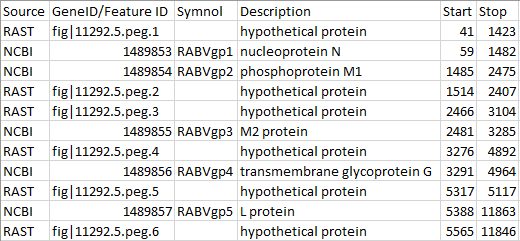
Таблица сравнения выдачи NCBI и RAST
Как видно из таблицы сравнения генов, одинаково аннотированных генов (в которых совпадают координаты
старт- и стоп-кодонов) нет ни одного. Все гены (и описанные RAST, и анноированные в NCBI) имеют пусть
схожие, но всё-таки различающиеся координаты. Также стоит отметить, что RAST аннотировал один ген, которого
нет в NCBI, в таблице он идёт под Feature ID: fig|11292.5.peg.5 . Этот же ген, что интересно, аннотирован
как ген обратной цепи, что несколько странно, поскольку генетическая информация вируса бешенства хранится
в одноцепочечной РНК.
Далее для первых трёх по счёту (в аннотации RAST) генов я проверил аннотацию с помощью blastp.
Ген 1
Для первого гена BLAST нашёл очень много соответствий, причём большую часть из них можно считать
очень хорошими (имели высокие значения идентичности, покрытия и низкие - E-value). Как видно ниже,
самая лучшая находка имеет процент покрытия 97% и идентичности - 100%. Значит, аннотированный
RAST белок - действительно нуклеопротеин N.
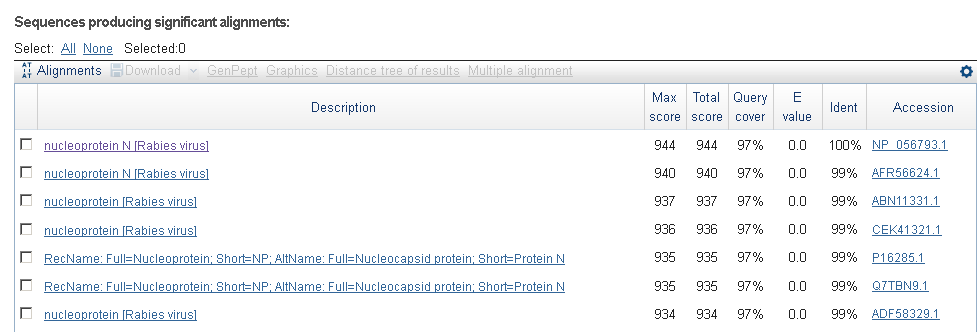
Находки blastp по первому аннотированному RAST белку
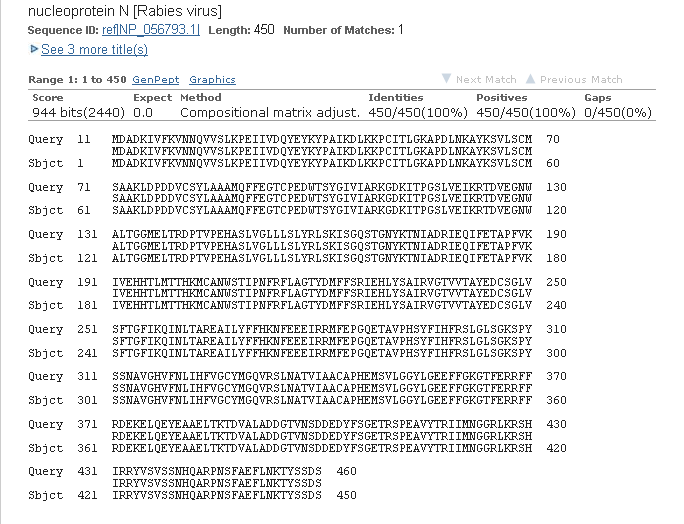
Выравнивание первого аннотированного RAST белка с лучшей находкой
Однако RAST аннотировал белок не совсем верно. Видно, что выравнивание начинается не с первой
аминокислоты, как белок из RefSeq, а только с одиннадцатой.
Последовательность же, взятая из GeneBank, полностью
совпадает с лучшей находкой BLAST

Выравнивание белка из GeneBank с лучшей находкой BLAST
Ген 2
Второй по счёту ген из аннотации RAST также при поиске в BLAST также получил множество схожих
с ним находок, причём лучшая из них имела стопроцентное покрытие и стопроцентную идентичность.
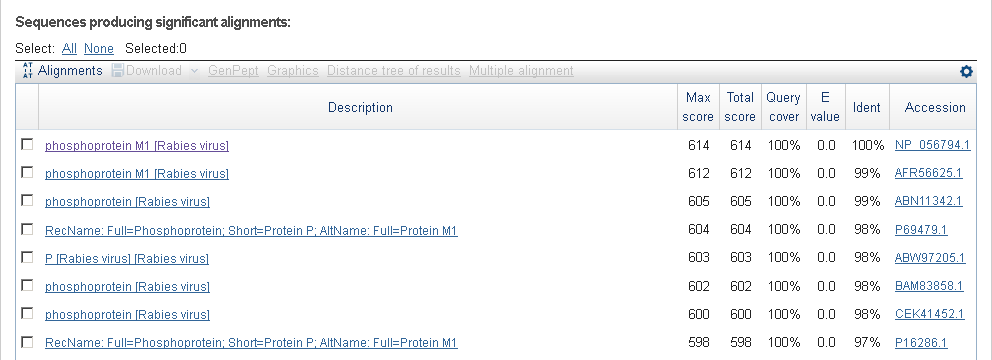
Находки blastp по второму аннотированному RAST белку
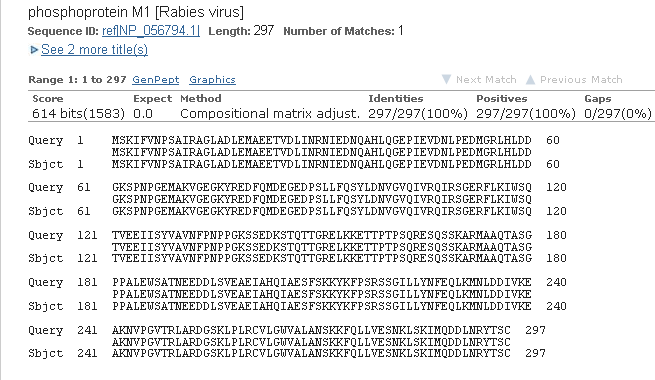
Выравнивание первого аннотированного RAST белка с лучшей находкой
Значит, это действительно фосфопротеин М1. Выравнивание же
последовательности этого же белка, взятой из GeneBank,
принесло точно такой же результат (абсолютная идентичность последовательности с лучшей находкой BLAST).
Ген 3
При проверке третьего по счёту гена, аннотированного RAST, BLAST выдал интересный результат.
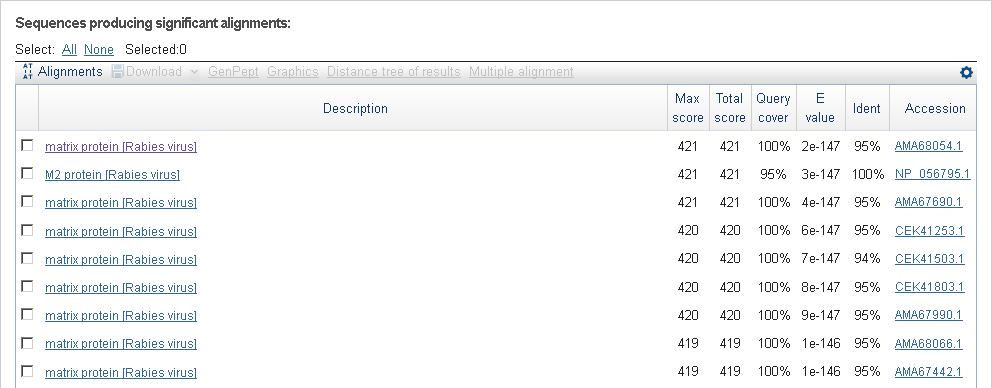
Находки blastp по третьему аннотированному RAST белку
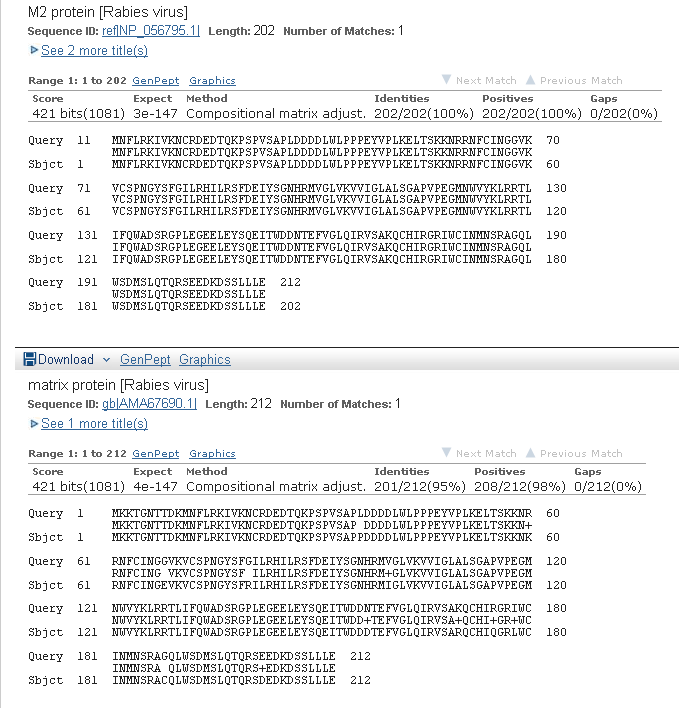
Выравнивание третьего аннотированного RAST белка с лучшими находками
Лучшую находку приходится выбирать из двух: либо та, у которой процент покрытия равен 95%, а
идентичность стопроцентная, либо та, у которой наоборот, идентичность равна 95%, а покрытие
стопроцентно. Однако не подлежит сомнению, что RAST аннотировал именно ген белка М2.
При поиске же последовательности этого белка из
GeneBank такой ситуации не возникло, и лучшая находка имела абсолютное сходство с данной
на вход алгоритму последовательностью.
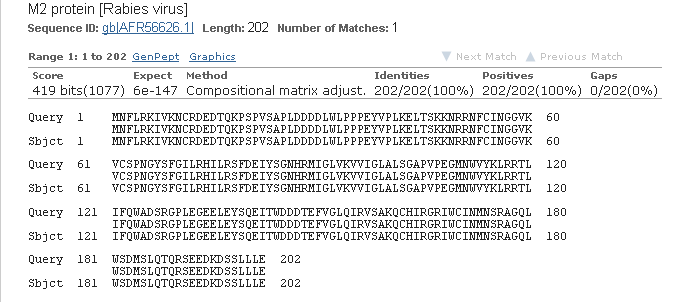
Выравнивание белка из GeneBank с лучшей находкой BLAST
Этот факт позволяет сделать вывод, что качество аннотации этого белка из GeneBank всё-таки выше, \
нежели из RAST, хотя в принадлежности последней именно белку М2 сомневаться не приходится.
Ссылка на главную страницу
© Головачев Ярослав
