Гены эукариот
Задание 1.
Для работы в данном задании я выбрал контиг unplaced-307.
Длина этого контига - 84991 нуклеотида. Информацию о длинах контигов получил с помощью команды
infoseq -only -snucleotide1 -name -length. Затем последовательность этого контига я дал
на вход алгоритму blastx (точнее, кусочек в первые 29880 нуклеотидов длиной, поскольку длина всего контига
для BLAST-а чересчур велика). В выдаче BLAST все организмы, которым принадлежат найденные последовательности
- грибы, так что, скорее всего, и организм Х5 - тоже гриб.
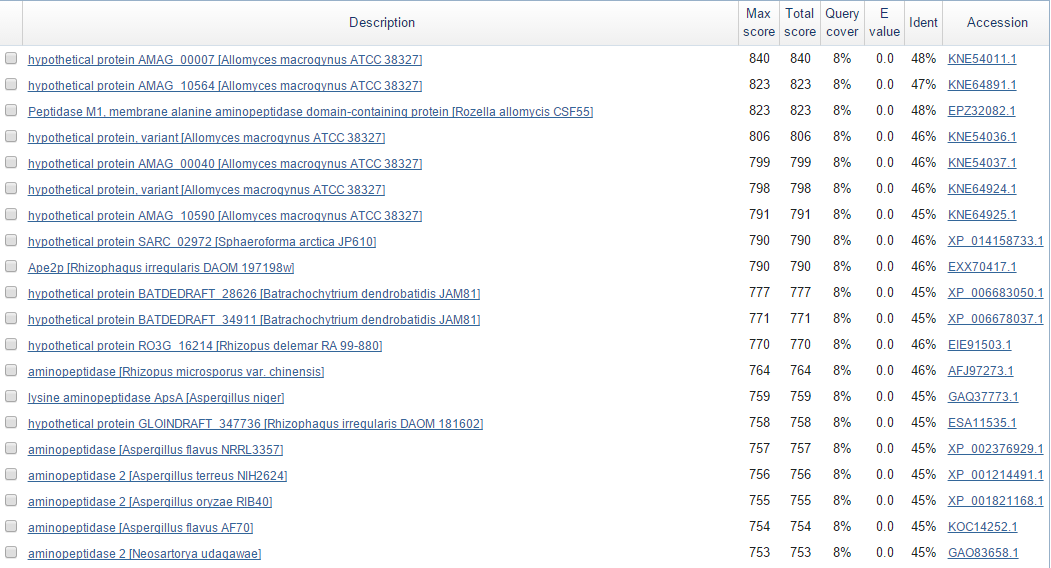
Результат выдачи алгоритма blastx
Затем я перешёл на сайт
AUGUSTUS, после чего выбрал в качестве
родственного организма Rhizopus oryzae (поскольку среди выдачи встречались результаты этого рода)
и запустил предсказание. Результат - архив
predictions.tar.gz.
Страница
с описанием статуса задания. В архиве содержатся файлы:
- augustus.gff - файл с предсказаниями генов, их последовательностями (в формате gff)
- augustus.aa - файл с предсказаниями генов в виде аминокислотных последовательностей белков
- augustus.cdsexons - файл с предсказаниями экзонов
- augustus.codingseq - файл с предсказаниями генов в виде CDS
- augustus.gbrowse - файл с информацией о структурных элементах пре-мРНК, соответствующих каждому конкретному предсказанному гену
- augustus.gtf - файл с предсказанием генов (в формате gtf)
Затем я проверил несколько предсказаний генов с помощью blastp, взяв в качестве query последовательности
белков из файла augustus.aa. Интрон-экзонную структуру же я взял
из файла augustus.gff.
Информация о выбранных генах помещена в данную таблицу.
Ген 1

Находки BLAST аминокислотной последовательности первого гена, предсказанного AUGUSTUS
Данный ген содержит 2 экзона. Конечно, встречались находки, имеющие почти такую же длину, но
принадлежали они насекомым.
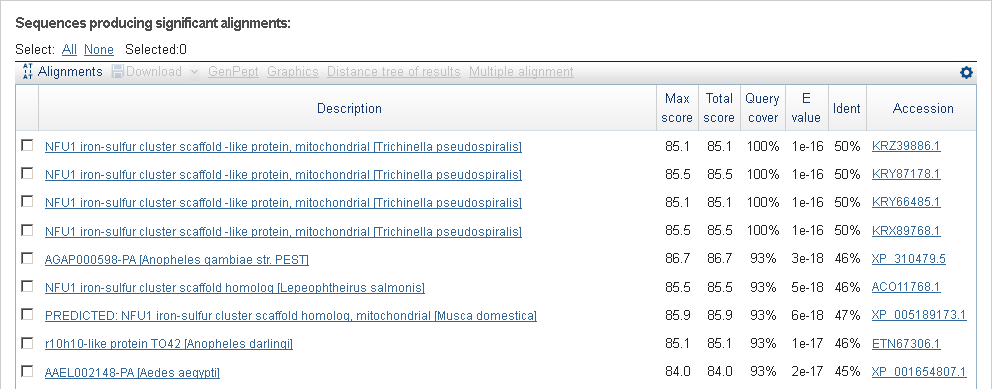
Находки BLAST аминокислотной последовательности первого гена, имеющие максимальное покрытие (находки отсортированы по Query cover)
Основная же часть находок blastp (поиск по БД nr) либо начинается позже, либо кончается раньше.
Затем я попробовал поискать по БД SwissProt.
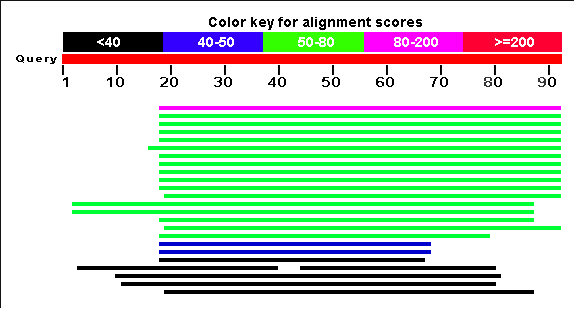
Находки BLAST по БД SwissProt
Исходя из полученных данных, можно заключить, что конец гена был найден AUGUSTUS верно. С чем
связано расхождение участков в начале последовательностей - не совсем ясно. Так, функции найденных
белков, к примеру, другие, что также может быть причиной расхождения.
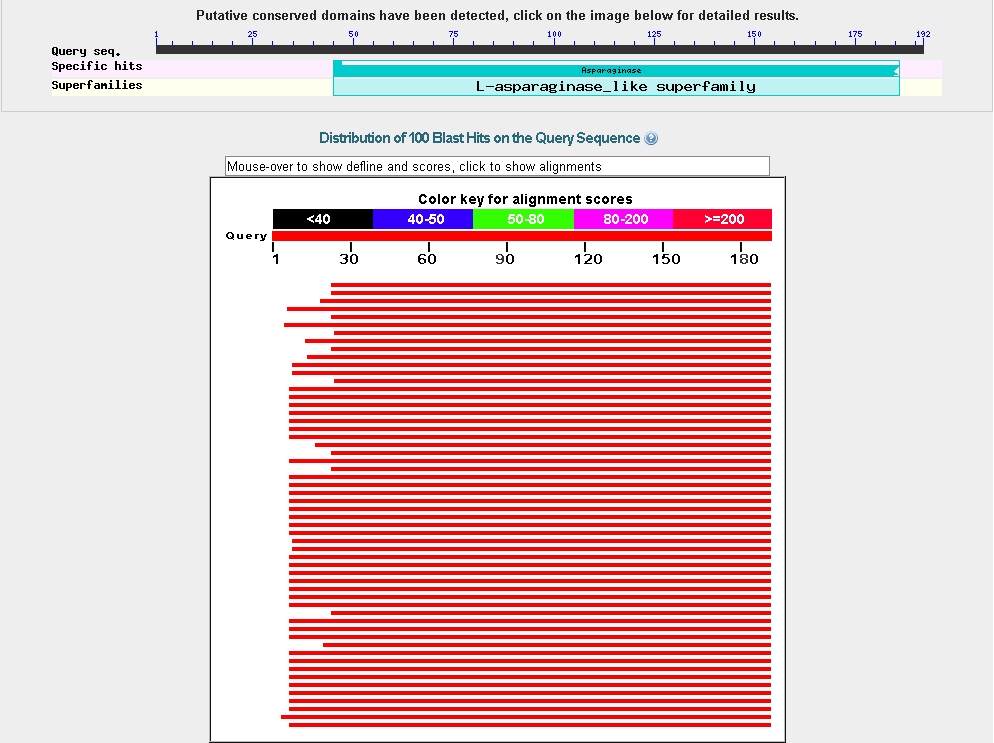
Судя по найденному при поиске белка консервативному домену, функция белка связана с железом
(транспорт железа).

Найденный BLAST консервативный домен
Ген 3
Находки BLAST аминокислотной последовательности третьего гена, предсказанного AUGUSTUS
Как видно, было найдено много хороших гомологов, причём начальные участки последовательностей
некоторых из них немного не совпадают с искомой последовательностью.
Находки BLAST, отсортированные по покрытию
Находки BLAST, отсортированные по покрытию (список)
Минимальный процент идентичности - 50%, максимальный - 65%. К тому же, выравнивания искомой
последовательности с лучшими находками довольно схожи. Всё это позволяет сделать вывод, что
интрон-экзонную структуру для данного предсказанного гена (скорее всего - некая лизофосфорилаза)
AUGUSTUS определил верно.
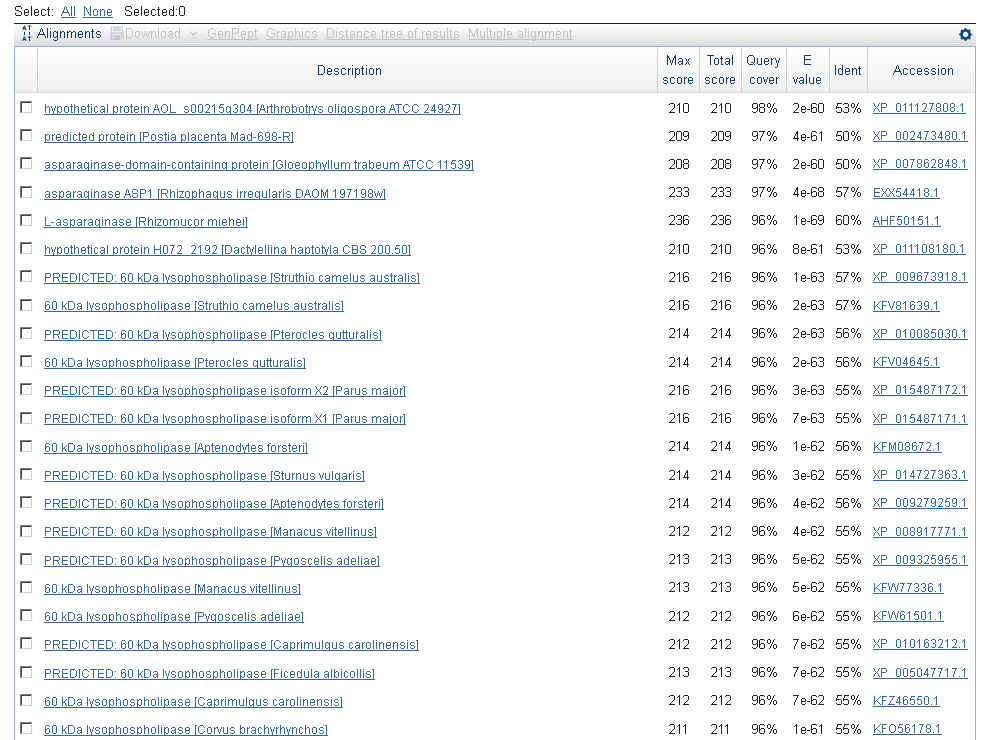
Ген 4
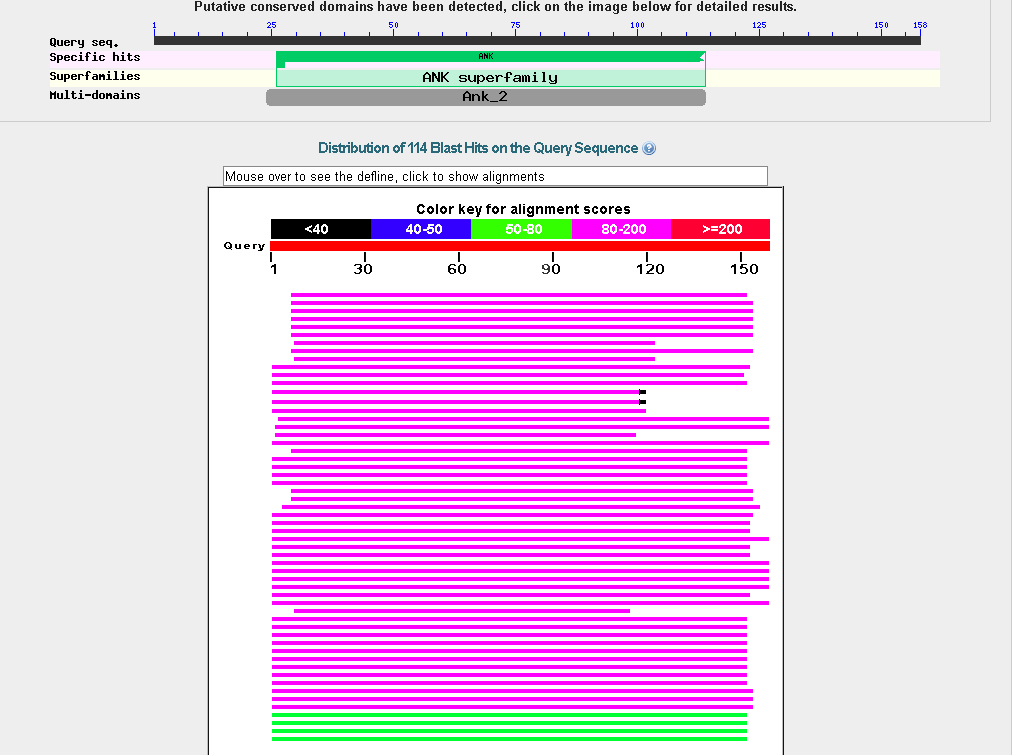
Находки BLAST аминокислотной последовательности четвёртого гена, предсказанного AUGUSTUS
Искомый ген - либо некая аспарагиназа, либо лизофосфорилаза (наиболее часто встречающиеся варианты
среди находок).
Как видно на скриншоте, некоторые находки имеют значительное отличие от искомой последовательности
в концевой части последовательностей. Хотя в лучших находках выравнивания сплошные, без
продолжительных гэпов.
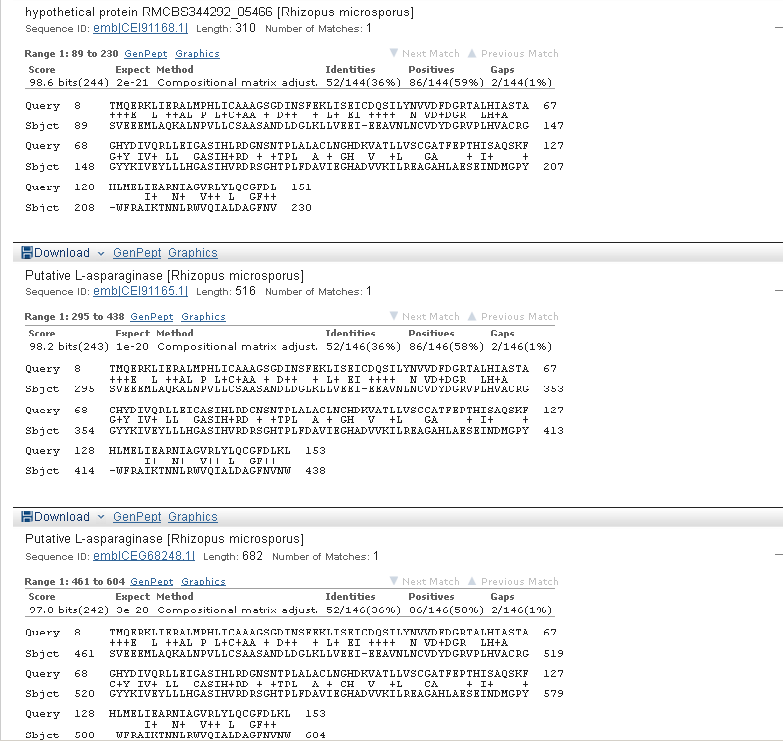
Выравнивания лучших находок с четвёртым геном
Последовательности же, отличающиеся по длине от искомой, часто принадлежат далёким организмам,
к примеру, гидрам.
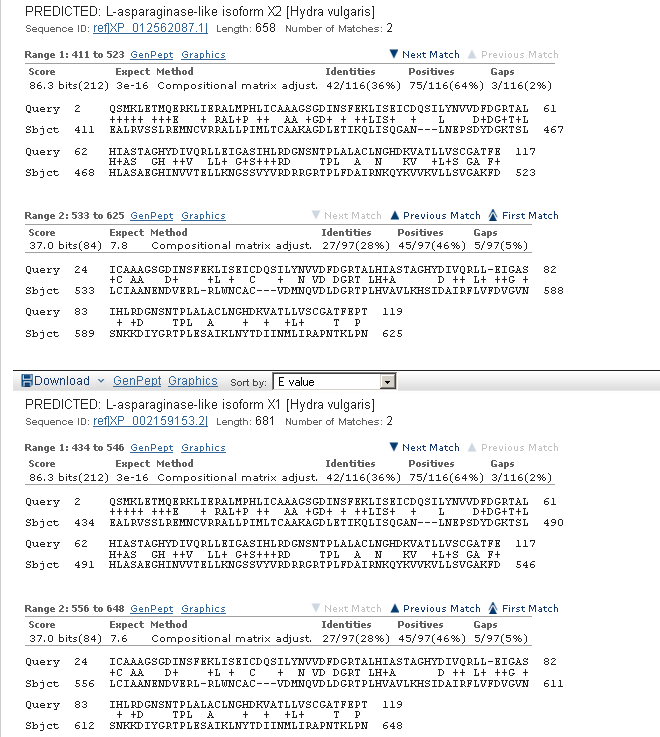
Выравнивания находок, принадлежащих гидрам с четвёртым геном
Эти выравнивания интересны ещё и тем, что в найденных гомологах части искомой последовательности
нашлись аж по два раза.
В целом, на основании имеющихся данных, можно сказать, что интрон-экзонная структура
определена верно.
Ген 9
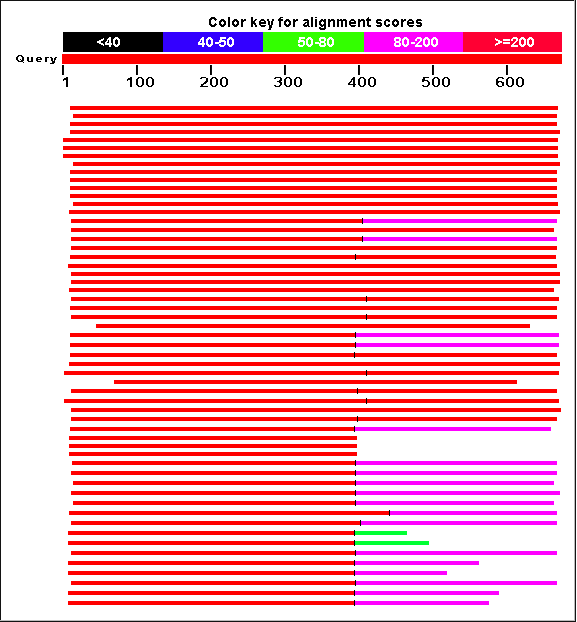
Находки BLAST аминокислотной последовательности девятого гена, предсказанного AUGUSTUS
Как видно, BLAsT нашёл много хороших гомологов, правда, максимальная идентичность находок
данной на вход последовательности - 50%.
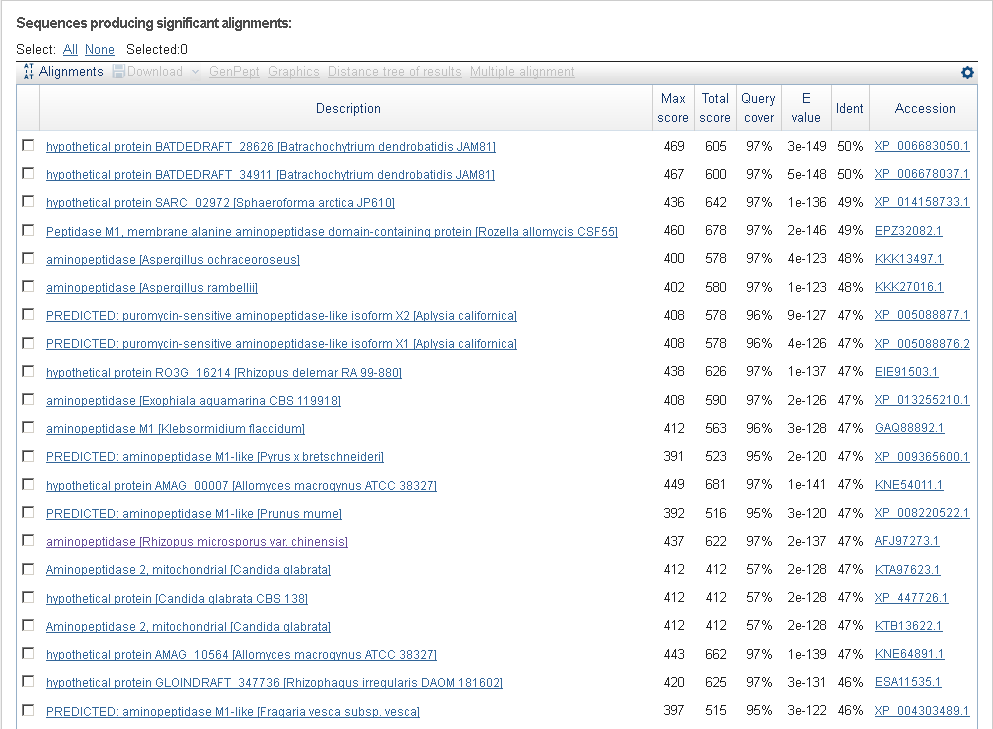
Находки BLAST аминокислотной последовательности девятого гена, имеющие максимальную идентичность
Большая часть находок - те или иные пептидазы, следовательно и ген, предсказанный AUGUSTUS,
вероятно, является пептидазой. Об этом свидетельствуют и найденные консервативные домены.
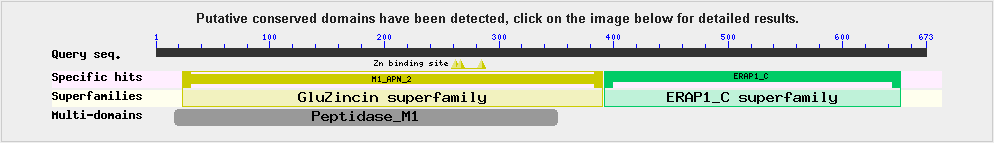
Консервативные домены предсказанного гена
Очень высокое покрытие, высокая идентичность и тот факт, что выравнивания лучших находок с
искомым белком похожи, говорят в пользу того, что AUGUSTUS предсказал интрон-экзонную структуру
верно.
Ген 23
Находок по данному гену было много, причём немаленькая часть из них (и из лучших) принадлежала
бактериям, поэтому я искал только среди грибов.
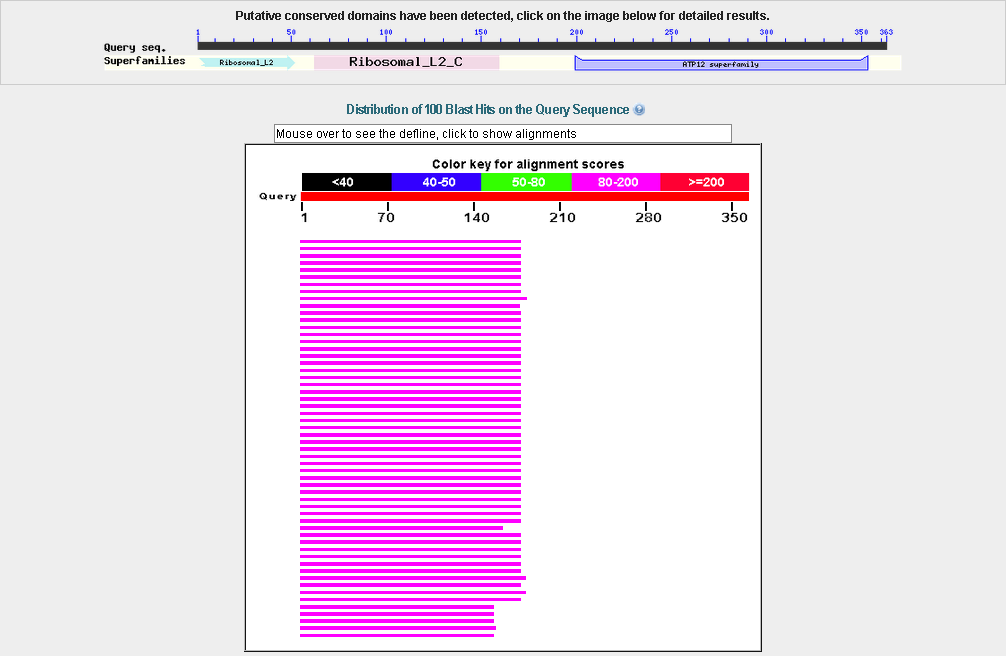
Находки BLAST аминокислотной последовательности (БД nr) двадцать третьего гена (среди грибов), предсказанного AUGUSTUS
Как видно, выравнивания всех находок сильно отличаются по величине от длины искомой
последовательности.
Причём при поиске по БД SwissProt (поиск шёл без ограничения по таксонам), ситуация похожая.
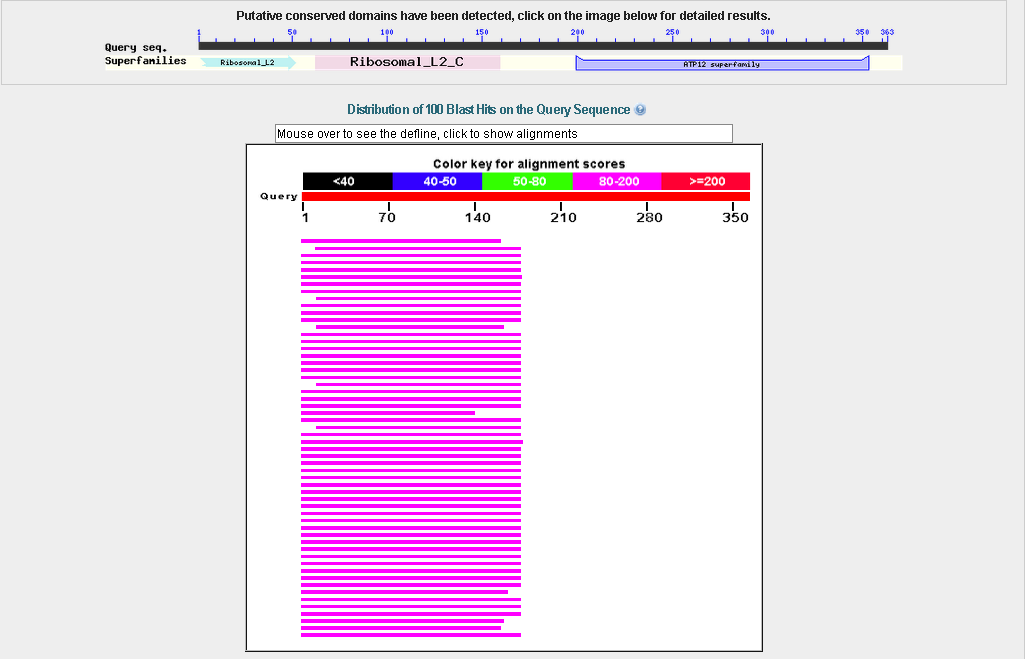
Находки BLAST аминокислотной последовательности по БД SwissProt
Много лучших находок - один и тот же белок - 50S ribosomal protein L2. Следовательно, и
предсказанный AUGUSTUS белок - с большой вероятностью он же.
Также можно заметить, что BLAST нашёл в последовательности искомого белка консервативный домен
ATP12, однако не было найдено ни одной находки, которая бы пересекалась в этом участке с искомой
последовательностью. Вывод: эта часть в ген не входит, и AUGUSTUS ошибся, включив её в состав гена.
Задание 2.
Для работы в данном задании я выбрал ген карнитинпальмитоилтрансферазы 2 (хромосома 1, RefSeq AC: NC_000001).
Ген расположен на прямой цепи, координаты гена: 53196859-53202736 (данные последней сборки hg38, взяты с сайта
UCSC Genome Browser). Далее было необходимо
сравнить аннотации RefSeq и AUGUSTUS, для чего я оставил в выдаче только три трека: base position, Refseq
и AUGUSTUS. Затем с помощью меню "Tools --> Table Browser" получидл интрон-экзонную структуру в текстовом
виде.

Изображение интрон-экзонной структуры гена карнитинпальмитоилтрансферазы 2 по данным RefSeq и AUGUSTUS.
Прямоугольниками обозначены экзоны, исчерченные отрезки между ними - интроны.
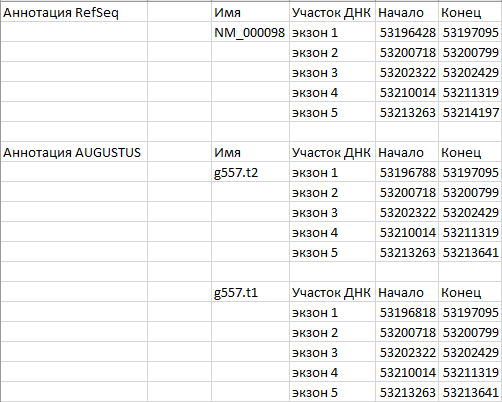
Координаты экзонов в различных аннотациях гена.
Как можно видеть, аннотация AUGUSTUS была проведена по двум транскриптам, данные которых немного
отличаются. В аннотации AUGUSTUS g557.t2 первый экзон начинается немного раньше, чем тот же экзон
в аннотации AUGUSTUS g557.t1 . Координата начала первого экзона также отличается и в аннотации RefSeq
(экзон начинается ещё раньше). Также можнро заметить, что координата конци последнего экзона отличается от
координат в аннотациях AUGUSTUS (экзон заканчивается позже).
Таблица экзон-интронной структуры, полученная с ресурса UCSC Genome Browser.
Таблица экзон-интронной структуры, отредактированная мной.
Ссылка на главную страницу
© Головачев Ярослав



