Сборка de novo.
Задание 1.
В этом задании было необходимо пакетом velvet cобрать из всех чтений своего набора (прошедших очистку)
контиги без использования референса. Пакет velvet стоит на kodomo и состоит из двух программ: velveth для
выделения k-меров из чтений ("hashing") и velvetg для сборки контигов на основе графа k-меров. Для
выполнения этого задания на сервере kodomo я использовал следующие команды:
velveth assembly 31 -fastq -short out.fastq
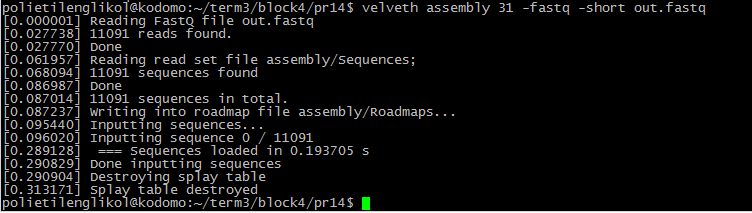
velvetg assembly
Как видно, была взята длина k-мера, равная 31. Получено 618 контигов, N50 = 247, максимальная длина
контига - 1392.

Файл с контигами, полученными программой velvet.
Задание 2.
В данном задании было необходимо с помощю программы blast (алгоритм megablast)
сравнить полученные программой velvet контиги с последовательностью хромосомы.
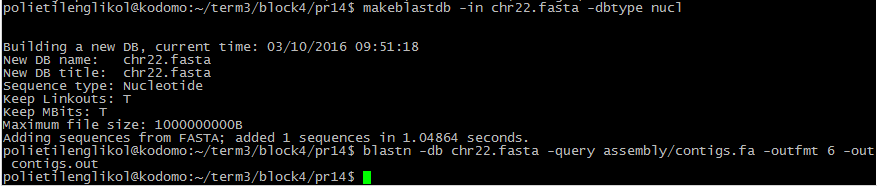
С помощью команды makeblastdb -in chr22.fasta -dbtype nucl файл с fasta-последовательностью
хромосомы 22 был задан как база данных, командой blastn -db chr22.fasta -query assembly/contigs.fa -outfmt 6 -out contigs.out
провёл поиск по заданной БД.
Результат работы blastn представлен в таблице Excel. В таблице
приведены все картировавшиеся контиги, на отдельных листах - контиги, встречавшиеся только один раз, либо
множество раз. Список контигов отсортирован в порядке возрастания меньшей координаты по хромосоме. Также
для контигов, встретившихся лишь один раз, посчитаны промежутки разрывов между ними, а также определено
наличие перекрывания.
Возможно, повторяющиеся контиги - это повторы, тогда количество таких повторов может достигать 4250.
Также встречаются контиги, которые очень сильно перекрываются, к примеру, представленные ниже на
скриншоте.

Однако контиги не объединяются, поскольку очень часты полиморфизмы, не позволяющие программе объединить
контиги.
Ссылка на главную страницу
© Головачев Ярослав