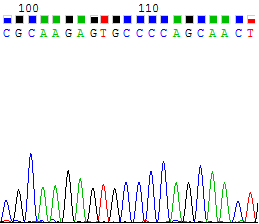
| Не читаемые 5'- и 3'- концы (координаты определены по прямой последовательности) | ||
| Прямая цепь | Обратная цепь | |
| 5'-нечитаемый конец | 1 - 20 | 1 - 25 |
| 3'-нечитаемый конец | ||

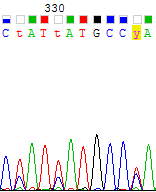
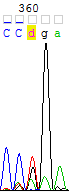
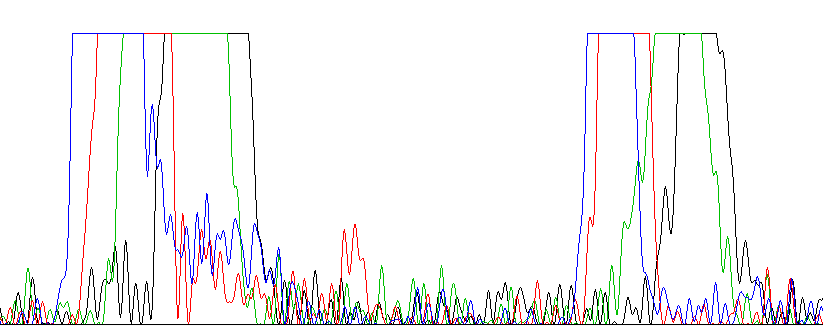
| Участок хроматограмм с чрезмерно высокими и широкими пиками. | |
| Прямая цепь | Комплементарная обратной цепь |
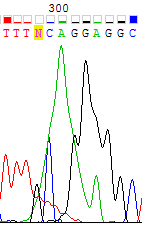 |
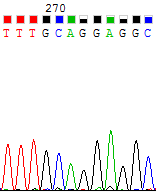 |
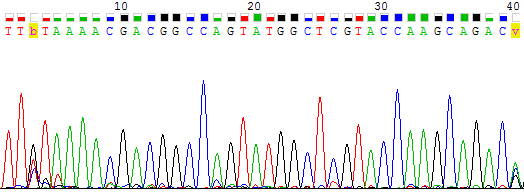
| Пики с высоким уровнем шума. | |
| Прямая цепь | Комплементарная обратной цепь |
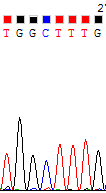 |
 |
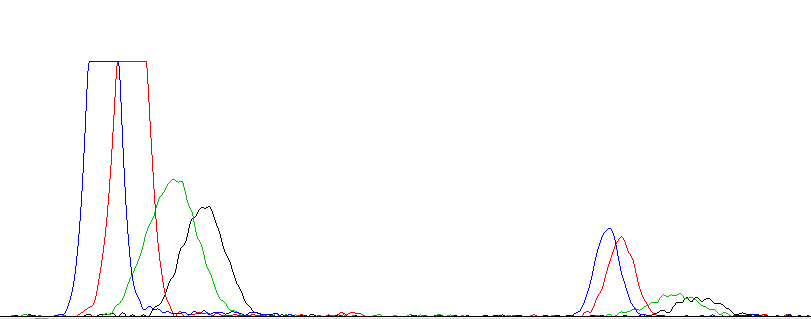
| Совмещённые пики. | |
| Прямая цепь | Комплементарная обратной цепь |
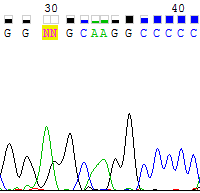 |
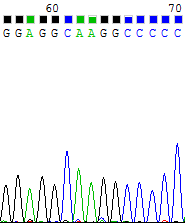 |