Укоренение и бутстрэп
Задание 1.
Выбранные для работы бактерии:
| Название |
Мнемоника |
| Bradyrhizobium diazoefficiens |
BRADU |
| Rhizobium etli |
RHIEC |
| Neisseria meningitidis |
NEIMA |
| Enterobacter sp. 638 |
ENT38 |
| Erwinia tasmaniensis |
ERWT9 |
| Escherichia coli |
ECOLI |
| Vibrio fischeri |
VIBFM |
| Proteus mirabilis |
PROMH |
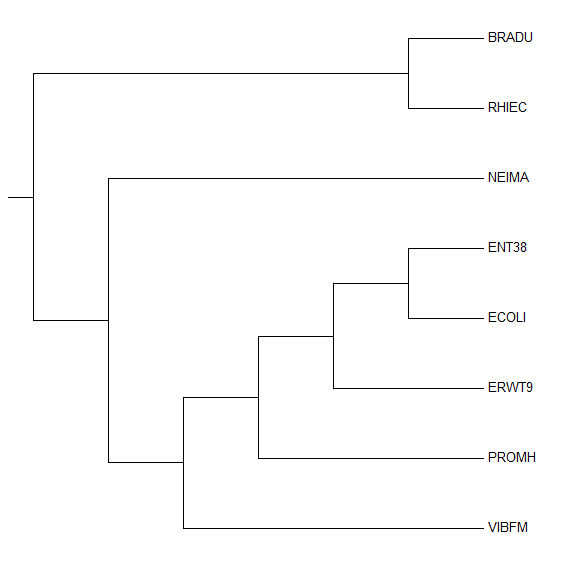
Филогенетическое дерево, построенное с помощью программы MEGA.
Филогенетическое дерево данных организмов было реконструировано с использованием метода Neighbor Joining Using % Identity
в программе JalView по выравниванию белковых последовательностей данных организмов.
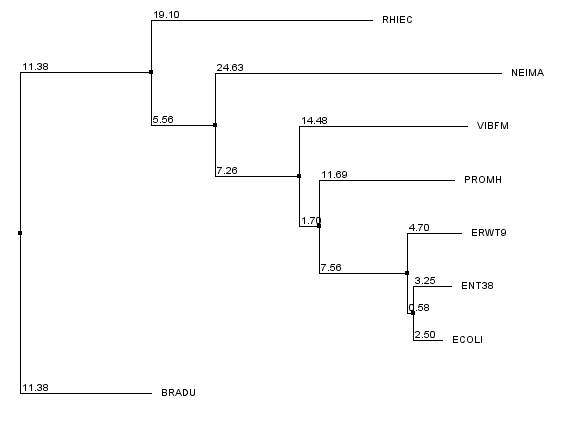
Филогенетическое дерево, построенное с помощью метода Neighbor Joining Using % Identity.
На рисунке видно, что дерево неукоренённое (метод Neighbor Joining строит именно такие). Поэтому, чтобы укоренить дерево
в среднюю точку, использовалась программа retree пакета PHYLIP.
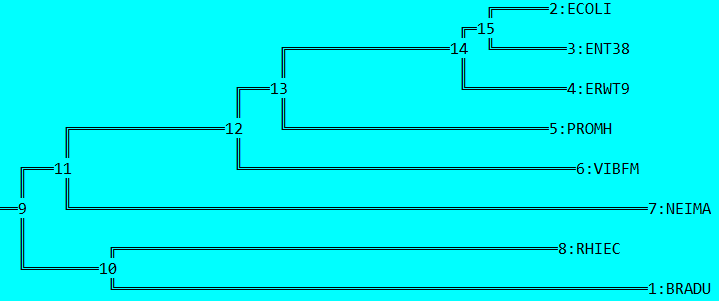
Филогенетическое дерево, укоренённое в среднюю точку программой retree пакета PHYLIP.
Филогенетическое дерево, укоренённое в среднюю точку программой retree пакета PHYLIP.
Видно, что дерево, укоренённое retree, не отличается от такового, построенного программой MEGA.
Задание 2.
Деревья, полученные методом максимальной экономии, невозможно укоренить в среднюю точку, поскольку этот метод предполагает
использование молекулярных часов. Однако можно воспользоваться методом укоренения с использованием внешней группы. В данном случае
роль внешней группы выполняла последовательность белка бактерии Bacillus subtilis. С использовании инструкции на
сайте было получено изображение укоренённого дерева.
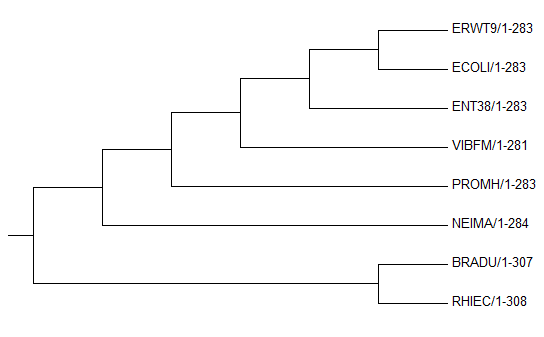
Филогенетическое дерево, укоренённое с использованием внешней группы.
Выравнивание с добавленной последовательностью белка бактерии Bacillus subtilis.
Задание 3.
Для проведения бутстрэп-анализа был использован метод MP (дерево реконструировалось по выравниванию из Задания 2, с добавленной
последовательностью белка бактерии Bacillus subtilis). Количество бутстрэп-реплик - 100. Программа MEGA выдала три дерева:
два "Original tree" и одно "Bootstrap consensus tree". Точно так же, как в Задании 2, в качестве корневой во всех деревьях была
указана ветвь, ведущая к BACSU, после чего были получены изображения деревьев без внешней группы (BACSU). Данные изображения можно
увидеть ниже.
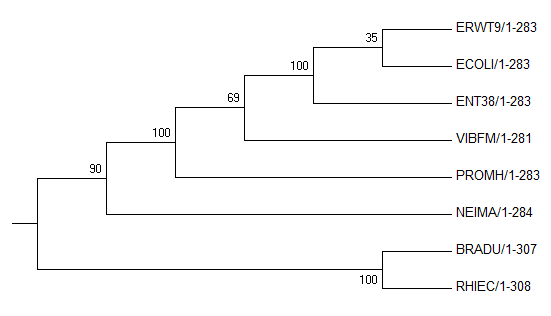
Original tree №1.
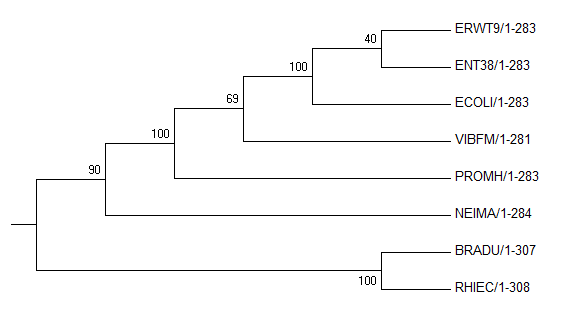
Original tree №2
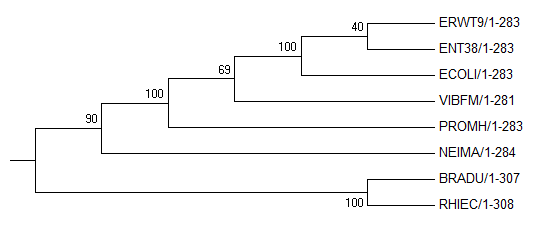
Bootstrap consensus tree.
Видно, что Original tree №2 и Bootstrap consensus tree совпадают. Также видно, что эти два дерева более вероятны, поскольку
ветвь, выделяющая кладу, содержащую ERWT9 и ENT38, имеет большую бутстрэп-поддержку, чем ветвь, выделяющую кладу, содержащую
ERWT9 и ECOLI.
Ссылка на главную страницу
© Головачев Ярослав
