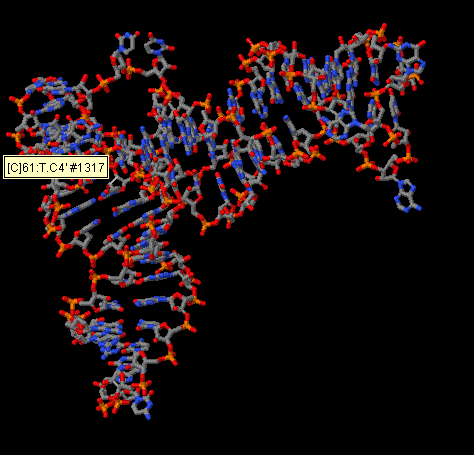
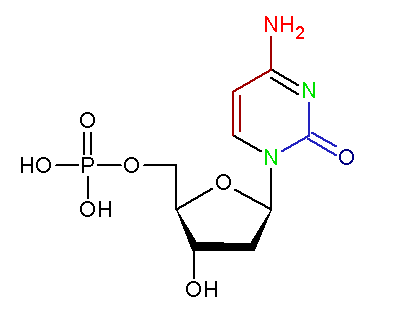
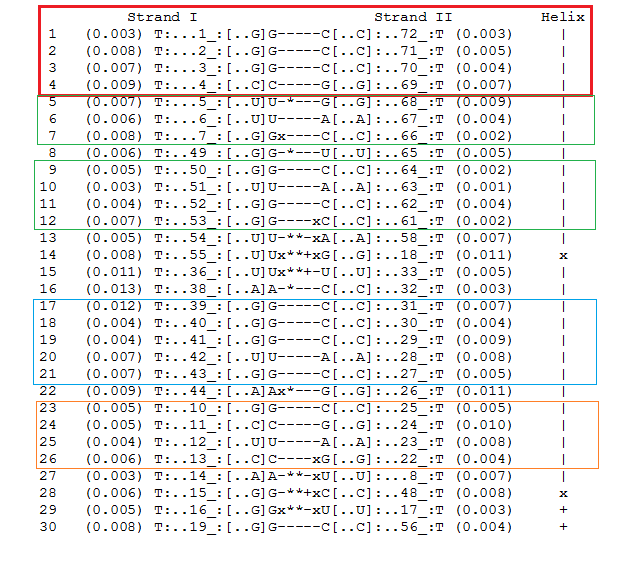
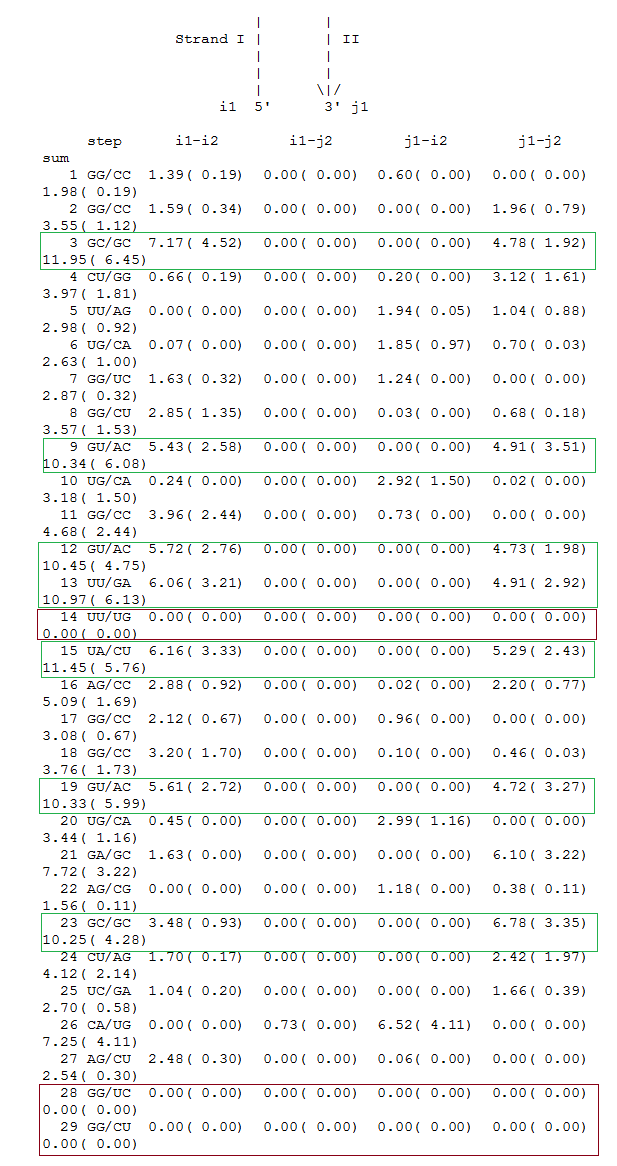
| base | alpha | beta | gamma | delta | epsilon | zeta | chi |
| 1 G | --- | --- | 65,3 | 85,4 | -147,1 | -75,8 | 178,8 |
| 2 G | -77,3 | -174,1 | 57,4 | 83,3 | -148,8 | -84,7 | -167,5 |
| 3 G | -66,7 | 175,0 | 50,9 | 81,3 | -138,0 | -81,6 | -160,8 |
| 4 C | -53,9 | 159,8 | 46,5 | 77,9 | -151,9 | -71,3 | -152,9 |
| 5 U | -50,3 | 170,2 | 53,9 | 81,9 | -157,4 | -80,6 | -163,5 |
| 6 U | -74,3 | -177,7 | 60,3 | 82,8 | -153,2 | -85,0 | -162,5 |
| 7 G | -56,7 | 161,7 | 69,8 | 113,6 | --- | --- | -154,5 |
| 8 G | --- | -172,5 | 44,5 | 81,6 | -149,9 | -66,4 | 179,0 |
| 9 G | -64,9 | -177,9 | 53,3 | 83,0 | -145,9 | -76,4 | -172,9 |
| 10 U | -59,7 | 173,5 | 49,0 | 80,9 | -153,1 | -72,7 | -162,6 |
| 11 G | -45,1 | 166,8 | 45,2 | 82,3 | -171,7 | -70,1 | -157,0 |
| 12 G | 152,4 | -163,7 | -179,4 | 85,6 | -145,1 | -65,6 | 179,6 |
| 13 U | -70,2 | -174,7 | 45,3 | 82,8 | -131,6 | -63,3 | -161,4 |
| 14 U | -69,2 | 170,7 | 52,6 | 81,7 | --- | --- | -152,0 |
| 15 U | --- | 126,2 | 56,5 | 114,1 | --- | --- | -150,8 |
| 16 A | --- | 109,6 | 144,5 | 82,7 | -132,9 | -60,5 | 176,4 |
| 17 G | -58,0 | 179,6 | 42,7 | 79,9 | -159,1 | -71,9 | -177,7 |
| 18 G | -66,7 | -175,0 | 55,5 | 82,5 | -149,0 | -72,3 | -174,4 |
| 19 G | -58,2 | 173,5 | 53,9 | 82,0 | -146,9 | -85,4 | -168,8 |
| 20 U | -59,6 | 159,2 | 62,3 | 83,9 | -154,7 | -61,9 | -166,5 |
| 21 G | -66,9 | 176,4 | 56,5 | 84,2 | -165,7 | -81,7 | -159,8 |
| 22 A | -47,4 | 171,9 | 46,3 | 83,9 | --- | --- | -145,1 |
| 23 G | --- | 159,2 | 49,8 | 87,2 | -137,0 | -77,1 | 170,7 |
| 24 C | -65,5 | -179,9 | 47,2 | 81,6 | -146,5 | -79,0 | -166,4 |
| 25 U | -57,4 | 172,5 | 47,9 | 84,0 | -158,9 | -69,2 | -153,7 |
| 26 C | 144,4 | -158,8 | -171,4 | 91,3 | -142,0 | -82,1 | -173,4 |
| 27 A | 32,7 | -161,7 | -66,5 | 92,6 | -162,9 | -59,0 | -160,6 |
| 28 G | 160,0 | -151,0 | 161,6 | 88,1 | -127,4 | 152,8 | -178,2 |
| 29 G | 167,3 | 118,5 | -176,0 | 82,9 | --- | --- | -38,9 |
| 30 G | --- | -135,6 | 56,9 | 94,1 | --- | --- | -89,9 |
|
| base | alpha | beta | gamma | delta | epsilon | zeta | chi |
| 1 C | 153,2 | -165,3 | 178,5 | 84,8 | --- | --- | -169,7 |
| 2 C | -64,0 | 169,6 | 56,9 | 81,1 | -161,8 | -76,4 | -160,5 |
| 3 C | -71,0 | -178,8 | 50,7 | 82,2 | -152,8 | -67,0 | -156,8 |
| 4 G | -49,2 | -178,2 | 29,6 | 82,7 | -167,2 | -72,9 | -156,6 |
| 5 G | 153,4 | -173,8 | -174,9 | 82,0 | -128,4 | -69,6 | 178,2 |
| 6 A | -66,4 | 175,7 | 47,5 | 81,1 | -171,3 | -65,3 | -155,2 |
| 7 C | -59,6 | 179,6 | 48,2 | 84,4 | -158,4 | -64,5 | -155,3 |
| 8 U | -57,9 | 172,9 | 51,8 | 81,3 | -156,6 | -79,4 | -161,7 |
| 9 C | -55,7 | 157,5 | 51,9 | 84,6 | -159,2 | -65,7 | -162,4 |
| 10 A | -66,1 | 177,4 | 50,3 | 83,6 | -150,3 | -82,0 | -159,3 |
| 11 C | -58,5 | 176,3 | 49,2 | 83,3 | -153,2 | -70,8 | -163,9 |
| 12 C | --- | 137,6 | 17,4 | 82,9 | -150,7 | -78,5 | -171,0 |
| 13 A | --- | -141,9 | 63,5 | 82,7 | --- | --- | -106,4 |
| 14 G | --- | -122,1 | 137,9 | 83,5 | --- | --- | -152,4 |
| 15 U | -68,2 | 163,1 | 67,1 | 114,9 | --- | --- | -145,9 |
| 16 C | -61,0 | 173,7 | 54,8 | 88,6 | -142,6 | -76,7 | -162,7 |
| 17 C | -61,4 | 176,1 | 56,3 | 83,9 | -153,4 | -67,5 | -161,0 |
| 18 C | -64,5 | 165,0 | 59,6 | 81,2 | -150,8 | -75,8 | -162,3 |
| 19 C | -70,9 | 164,6 | 55,1 | 80,7 | -152,1 | -75,5 | -155,6 |
| 20 A | -72,9 | -175,9 | 53,6 | 86,3 | -143,7 | -73,2 | -155,3 |
| 21 C | -65,5 | 177,9 | 48,4 | 84,4 | -153,9 | -70,9 | -159,2 |
| 22 G | -62,6 | 173,2 | 52,4 | 85,0 | -150,6 | -61,1 | -163,5 |
| 23 C | -49,5 | 158,0 | 56,6 | 80,8 | -142,3 | -64,0 | -165,7 |
| 24 G | -60,4 | 161,0 | 47,4 | 82,2 | -148,0 | -78,0 | -160,9 |
| 25 A | -43,6 | 170,9 | 39,0 | 83,4 | -141,9 | -88,6 | -161,9 |
| 26 G | --- | -138,2 | -175,2 | 84,0 | -148,6 | -84,8 | -174,3 |
| 27 U | --- | -159,6 | 57,5 | 84,6 | --- | --- | -159,8 |
| 28 C | --- | 98,9 | 49,2 | 98,9 | --- | --- | -157,5 |
| 29 U | --- | -158,0 | 173,2 | 83,5 | --- | --- | -166,7 |
| 30 C | --- | -178,9 | 53,7 | 85,9 | --- | --- | -169,9 |
|